How to design estimators tting with accuracy measures Theories and applications in bioinformatics
|
|
|
- Χλόη Λιακόπουλος
- 8 χρόνια πριν
- Προβολές:
Transcript
1 How to design estimators tting with accuracy measures Theories and applications in bioinformatics Michiaki HAMADA Toshiyuki SATO (): 1, Miyazawa 22) HMM 4) RNA 2 Miyazawa ( ) 22) 2 ( ) 1 1 D, Y () Y p(y D) y Y 1 (decoding) p(y D) ( ) 1( ) θ Y y Y G : Y Y R +, G(θ, y) (gain function). 2(MEG) 1 (MEG ) ŷ (MEG) =argmax G(θ, y)p(θ D)dθ y Y Θ
2 MEG (Maximum Expected Accuracy Estimator; MEA ) (loss function) MEA ( ) 3 γ 1 Y Y {0, 1} n MEG γ RNA 2 Y {0, 1} n x x y {0, 1} x x : x i x k y ik =1 y ik =0 y Y {0, 1} n y 1 0 ( ) ( ) y Y θ Y,, TP(θ, y), TN(θ, y), FP(θ, y), FN(θ, y) (TP TN) (FP FN) G(θ, y) =α 1 TP(θ, y)+α 2 TN(θ, y) α 3 FP(θ, y) α 4 FN(θ, y). (1) α k (k =1, 2, 3, 4). Seisitivity (SEN), Positive Predictive Value (PPV), Matthews correlation coef cient (MCC) F-score ( TP, TN, FP, FN ; 1) ) MEG 3(γ-centroid estimator) γ 0 γ G(θ, y) =γtp(θ, y)+tp(θ, y) (2) MEG γ =1 γ 2) γ (1) MEG 1 (1) MEG γ = α1+α4 α 2+α 3 γ γ SEN PPV γ 2 Y y = {y i } Y y = {y i } Y i y i {y i, 0}. γ 1/(γ +1) p i = θ Y I(θ i =1)p(θ D) p i () RNA 2 (2 ) i {p i } i 21) γ p i 1/(γ +1) Y Y 0 γ 1 γ 1/(γ +1) 1 0 ( Y ) 9) 1( γ ) γ 1/(γ +1) ( 2) γ [0, 1] γ 1/(γ +1) 16 2
3 γ >1 γ Needleman-Wunsch 24) M i 1,k 1 +(γ +1)p ik 1 M i,k =max M i 1,k. M i,k 1 M i,k x 1 x i x 1 x k 2(2 γ ) γ 2 1/(γ +1) ( 2) γ [0, 1] γ 1/(γ +1) γ >1 γ Nussinov 25) M i,j =max M i+1,j M i,j 1 M i+1,j 1 +(γ +1)p ij 1 max k [M i,k + M k+1,j ] M i,j x i x i+1 x j 2 γ ( ) γ S ( ) S 2 n 1 n 1 (n S ) 2 Hamming 26) 1-centroid γ>1 γ 4 MEG (1) SEN, PPV, F-score, MCC. MEG RNA 2 MCC/F-score 12) Hamada (pseudo-expected accuracy) 12) TP, TN, FP, FN ( MCC F-score; 1) ) Acc = f(tp, TN, FP, FN) y Âcc 0 (y) =f( TP, TN, FP, FN). X X(=TP,FP,TN,FN) {p i } ( RNA 2 ) 2 MCC F-score MCC F-score 12) γ γ ) ( MCC F-score ) SEN PPV 5 γ x, x x x z x, x x, x, z γ 13, 14) () 4, 28) (Probabilistic consistency transformation; PCT) 9). 17 3
4 6.3 γ (CentroidAlign) 13) RNA 2 γ (CentroidHomfod) 14) Kato RNA-RNA γ 17) (RactIP) RNA- RNA 2 RNA γ 2 NP γ ( 2) γ 1 Do 7) 6.1 RNA 2 Kall HMM 2 RNA 2 RNA 5). MEG 16) γ SEN, PPV 7 10). γ ( 1) γ 6.2 γ Schwartz AMA (Alignment Metric Accuracy) AMA 29) AMA γ 6) SEN PPV RNA 2 AMA SEN, PPV γ γ ( SPS ) SEN, PPV, MCC, F-score ( 1) 5, 18, 20, 30) 8, 31) γ 6, 32). NEDO 18 4
5 1 Holmes & Durbin 15) -centroid a SPS b Miyazawa 22) 1-centroid Schwartz et al. 29) AMA c AMA Do et al. 4) ProbCons -centroid ( ) d SPS Roshan et al. 27) ProbAlign -centroid ( ) SPS Sahraeian et al. 28) PicXAA -centroid ( ) SPS Frith et al. 6) LAST γ-centroid SEN, PPV Hamada et al. 10) CentroidFold RNA2 γ-centroid SEN, PPV Hamada et al. 12) CentroidFold RNA2 MCC/F-score e MCC, F-score Do et al. 5) CONTRAfold RNA2 f Lu et al. 20) MaxExpect RNA2 Ding et al. 3) Sfold RNA2 1-centroid g Hamada et al. 14) CentroidHomfold RNA2 h γ-centroid ( ) SEN, PPV Hamada et al. 11) CentroidAlifold RNA 2 γ-centroid SEN, PPV Seemann et al. 30) PETfold RNA 2 Knudsen & Hein 19) Pfold RNA 2 Kiryu et al. 18) McCaskill-MEA RNA 2 Hamada et al. 13) CentroidAlign RNA γ-centroid ( ) SEN, PPV Tabei et al. 32) SCARNA-LM RNA γ-centroid SEN, PPV Kato et al. 17) RactIP RNA-RNA γ-centroid SEN, PPV Kall et al. 16) i Do et al. 7) CONTRAST Michal et al. 23) HIV a γ γ ((2) ); b Sum-of-pairs score; c Alignment metric accuracy; d γ (5 ); e γ γ 2 ; f RNA ( ) SEN PPV ; g 2 ; h 2 ; i RNA CBRC 1) P. Baldi, S. Brunak, Y. Chauvin, C. A. Andersen, and H. Nielsen. Assessing the accuracy of prediction algorithms for classi cation: an overview. Bioinformatics, 16: , May ) L. Carvalho and C. Lawrence. Centroid estimation in discrete high-dimensional spaces with applications in biology. Proc. Natl. Acad. Sci. U.S.A., 105: , ) Y. Ding, C. Chan, and C. Lawrence. RNA secondary structure prediction by centroids in a Boltzmann weighted ensemble. RNA, 11: , Aug ) C. Do, M. Mahabhashyam, M. Brudno, and S. Batzoglou. ProbCons: Probabilistic consistency-based multiple sequence alignment. Genome Res., 15: , Feb ) C. Do, D. Woods, and S. Batzoglou. CONTRAfold: RNA secondary structure prediction without physicsbased models. Bioinformatics, 22:e90 98, Jul ) M. C. Frith, M. Hamada, and P. Horton. Parameters for accurate genome alignment. BMC Bioinformatics, 11:80, Feb ) S. Gross, C. Do, M. Sirota, and S. Batzoglou. CON- TRAST: a discriminative, phylogeny-free approach to multiple informant de novo gene prediction. Genome Biol., 8:R269, ) S. S. Gross, O. Russakovsky, C. B. Do, and S. Batzoglou. Training conditional random elds for maximum labelwise accuracy. In B. Schölkopf, J. Platt, and T. Hoffman, editors, Advances in Neural Infor- 19 5
6 mation Processing Systems 19, pages MIT Press, Cambridge, MA, ) M. Hamada, H. Kiryu, W. Iwasaki, and K. Asai. Generalized Centroid Estimators in Bioinformatics. PLoS ONE 6(2): e16450, ) M. Hamada, H. Kiryu, K. Sato, T. Mituyama, and K. Asai. Prediction of RNA secondary structure using generalized centroid estimators. Bioinformatics, 25: , ) M. Hamada, K. Sato, and K. Asai. Improving the accuracy of predicting secondary structure for aligned RNA sequences. Nucleic Acids Res., 2010 (in press). 12) M. Hamada, K. Sato, and K. Asai. Prediction of RNA secondary structure by maximizing pseudo-expected accuracy. BMC Bioinformatics, 11:586, ) M. Hamada, K. Sato, H. Kiryu, T. Mituyama, and K. Asai. CentroidAlign: fast and accurate aligner for structured RNAs by maximizing expected sum-ofpairs score. Bioinformatics, 25: , ) M. Hamada, K. Sato, H. Kiryu, T. Mituyama, and K. Asai. Predictions of RNA secondary structure by combining homologous sequence information. Bioinformatics, 25:i , ) I. Holmes and R. Durbin. Dynamic programming alignment accuracy. J. Comput. Biol., 5: , ) L. Kall, A. Krogh, and E. L. Sonnhammer. An HMM posterior decoder for sequence feature prediction that includes homology information. Bioinformatics, 21 Suppl 1:i , ) Y. Kato, K. Sato, M. Hamada, Y. Watanabe, K. Asai, and T. Akutsu. RactIP: fast accurate prediction of RNA-RNA interaction using integer programming. Bioinformatics, 2010 (in press). 18) H. Kiryu, T. Kin, and K. Asai. Robust prediction of consensus secondary structures using averaged base pairing probability matrices. Bioinformatics, 23: , ) B. Knudsen and J. Hein. Pfold: RNA secondary structure prediction using stochastic context-free grammars. Nucleic Acids Res, 31(13): , Jul ) Z. J. Lu, J. W. Gloor, and D. H. Mathews. Improved RNA secondary structure prediction by maximizing expected pair accuracy. RNA, 15: , Oct ) J. S. McCaskill. The equilibrium partition function and base pair binding probabilities for RNA secondary structure. Biopolymers, 29(6-7): , May ) S. Miyazawa. A reliable sequence alignment method based on probabilities of residue correspondences. Protein Eng., 8: , Oct ) M. Nánási, T. Vinar, and B. Brejová. The Highest Expected Reward Decoding for HMMs with Application to Recombination Detection. CoRR, abs/ , ) S. Needleman and C. Wunsch. A general method applicable to the search for similarities in the amino acid sequence of two proteins. J. Mol. Biol., 48: , Mar ) R. Nussinov, G. Pieczenk, J. Griggs, and D. Kleitman. Algorithms for loop matchings. SIAM Journal of Applied Mathematics, 35:68 82, ) D. F. Robinson and L. R. Foulds. Comparison of phylogenetic trees. Mathematical Biosciences, 53(1-2): , February ) U. Roshan and D. Livesay. Probalign: multiple sequence alignment using partition function posterior probabilities. Bioinformatics, 22: , Nov ) S. M. Sahraeian and B. J. Yoon. PicXAA: greedy probabilistic construction of maximum expected accuracy alignment of multiple sequences. Nucleic Acids Res., 38: , Aug ) A. S. Schwartz, E. W. Myers, and L. Pachter. Alignment metric accuracy, ) S. Seemann, J. Gorodkin, and R. Backofen. Unifying evolutionary and thermodynamic information for RNA folding of multiple alignments. Nucleic Acids Res., 36: , ) J. Suzuki, E. McDermott, and H. Isozaki. Training conditional random elds with multivariate evaluation measures. In Proceedings of the 21st International Conference on Computational Linguistics and 44th Annual Meeting of the Association for Computational Linguistics, pages , Sydney, Australia, July Association for Computational Linguistics. 32) Y. Tabei and K. Asai. A local multiple alignment method for detection of non-coding RNA sequences. Bioinformatics, 25: , Jun
A sequence alignment algorithm using the transition quantity
 1 1 1 MTRAP A sequence alignment algorithm using the transition quantity Toshihide Hara, 1 Keiko Sato 1 and Masanori Ohya 1 We have been developed a sequence alignment algorithm using the transition quantity.
1 1 1 MTRAP A sequence alignment algorithm using the transition quantity Toshihide Hara, 1 Keiko Sato 1 and Masanori Ohya 1 We have been developed a sequence alignment algorithm using the transition quantity.
BMI/CS 776 Lecture #14: Multiple Alignment - MUSCLE. Colin Dewey
 BMI/CS 776 Lecture #14: Multiple Alignment - MUSCLE Colin Dewey 2007.03.08 1 Importance of protein multiple alignment Phylogenetic tree estimation Prediction of protein secondary structure Critical residue
BMI/CS 776 Lecture #14: Multiple Alignment - MUSCLE Colin Dewey 2007.03.08 1 Importance of protein multiple alignment Phylogenetic tree estimation Prediction of protein secondary structure Critical residue
th International Conference on Machine Learning and Applications. E d. h. U h h b w k. b b f d h b f. h w k by v y
 212 11th International Conference on Machine Learning and Applications C b G E P fi d P P I f Id fy F M d D d W, M O h, E Z,T L C f C S, U v y f M, C G b, FL 33146, USA E : d.w 1@. d, h @.. d D f C S d
212 11th International Conference on Machine Learning and Applications C b G E P fi d P P I f Id fy F M d D d W, M O h, E Z,T L C f C S, U v y f M, C G b, FL 33146, USA E : d.w 1@. d, h @.. d D f C S d
1530 ( ) 2014,54(12),, E (, 1, X ) [4],,, α, T α, β,, T β, c, P(T β 1 T α,α, β,c) 1 1,,X X F, X E F X E X F X F E X E 1 [1-2] , 2 : X X 1 X 2 ;
![1530 ( ) 2014,54(12),, E (, 1, X ) [4],,, α, T α, β,, T β, c, P(T β 1 T α,α, β,c) 1 1,,X X F, X E F X E X F X F E X E 1 [1-2] , 2 : X X 1 X 2 ; 1530 ( ) 2014,54(12),, E (, 1, X ) [4],,, α, T α, β,, T β, c, P(T β 1 T α,α, β,c) 1 1,,X X F, X E F X E X F X F E X E 1 [1-2] , 2 : X X 1 X 2 ;](/thumbs/91/107463875.jpg) ISSN1000-0054 CN11-2223/N ( ) 2014 54 12 JTsinghuaUniv(Sci& Technol), 2014,Vol.54, No.12 4/20 1529-1533,, (,, (), 100084) [1-2] :,,,,,,,, :, 0.3~ [3] 0.8BLEU,, : ; ; [4], ; :TP391.2 :A, :1000-0054(2014)12-1529-05,
ISSN1000-0054 CN11-2223/N ( ) 2014 54 12 JTsinghuaUniv(Sci& Technol), 2014,Vol.54, No.12 4/20 1529-1533,, (,, (), 100084) [1-2] :,,,,,,,, :, 0.3~ [3] 0.8BLEU,, : ; ; [4], ; :TP391.2 :A, :1000-0054(2014)12-1529-05,
Μηχανική μάθηση (Machine Learning)
 Μηχανική μάθηση (Machine Learning) Μηχανική Μάθηση The field of study that gives computers the ability to learn without being explicitly programmed Arthur Samuel (1959) "A computer program is said to learn
Μηχανική μάθηση (Machine Learning) Μηχανική Μάθηση The field of study that gives computers the ability to learn without being explicitly programmed Arthur Samuel (1959) "A computer program is said to learn
: Monte Carlo EM 313, Louis (1982) EM, EM Newton-Raphson, /. EM, 2 Monte Carlo EM Newton-Raphson, Monte Carlo EM, Monte Carlo EM, /. 3, Monte Carlo EM
 2008 6 Chinese Journal of Applied Probability and Statistics Vol.24 No.3 Jun. 2008 Monte Carlo EM 1,2 ( 1,, 200241; 2,, 310018) EM, E,,. Monte Carlo EM, EM E Monte Carlo,. EM, Monte Carlo EM,,,,. Newton-Raphson.
2008 6 Chinese Journal of Applied Probability and Statistics Vol.24 No.3 Jun. 2008 Monte Carlo EM 1,2 ( 1,, 200241; 2,, 310018) EM, E,,. Monte Carlo EM, EM E Monte Carlo,. EM, Monte Carlo EM,,,,. Newton-Raphson.
Research on vehicle routing problem with stochastic demand and PSO2DP algorithm with Inver2over operator
 2008 10 10 :100026788 (2008) 1020076206 (, 400074) :, Inver2over,,, : ; ; ; Inver2over ; : F54015 : A Research on vehicle routing problem with stochastic demand and PSO2DP algorithm with Inver2over operator
2008 10 10 :100026788 (2008) 1020076206 (, 400074) :, Inver2over,,, : ; ; ; Inver2over ; : F54015 : A Research on vehicle routing problem with stochastic demand and PSO2DP algorithm with Inver2over operator
Stabilization of stock price prediction by cross entropy optimization
 ,,,,,,,, Stabilization of stock prediction by cross entropy optimization Kazuki Miura, Hideitsu Hino and Noboru Murata Prediction of series data is a long standing important problem Especially, prediction
,,,,,,,, Stabilization of stock prediction by cross entropy optimization Kazuki Miura, Hideitsu Hino and Noboru Murata Prediction of series data is a long standing important problem Especially, prediction
ΒΙΟΠΛΗΡΟΦΟΡΙΚΗ ΙΙ. Δυναμικός Προγραμματισμός. Παντελής Μπάγκος
 ΒΙΟΠΛΗΡΟΦΟΡΙΚΗ ΙΙ Δυναμικός Προγραμματισμός Παντελής Μπάγκος Δυναμικός Προγραμματισμός Στοίχιση (τοπική-ολική) RNA secondary structure prediction Διαμεμβρανικά τμήματα Hidden Markov Models Άλλες εφαρμογές
ΒΙΟΠΛΗΡΟΦΟΡΙΚΗ ΙΙ Δυναμικός Προγραμματισμός Παντελής Μπάγκος Δυναμικός Προγραμματισμός Στοίχιση (τοπική-ολική) RNA secondary structure prediction Διαμεμβρανικά τμήματα Hidden Markov Models Άλλες εφαρμογές
Buried Markov Model Pairwise
 Buried Markov Model 1 2 2 HMM Buried Markov Model J. Bilmes Buried Markov Model Pairwise 0.6 0.6 1.3 Structuring Model for Speech Recognition using Buried Markov Model Takayuki Yamamoto, 1 Tetsuya Takiguchi
Buried Markov Model 1 2 2 HMM Buried Markov Model J. Bilmes Buried Markov Model Pairwise 0.6 0.6 1.3 Structuring Model for Speech Recognition using Buried Markov Model Takayuki Yamamoto, 1 Tetsuya Takiguchi
Medium Data on Big Data
 IT 17081 Examensarbete 15 hp November 2017 Medium Data on Big Data Predicting Disk Failures in CERNs NetApp-based Data Storage System Albin Stjerna Institutionen för informationsteknologi Department of
IT 17081 Examensarbete 15 hp November 2017 Medium Data on Big Data Predicting Disk Failures in CERNs NetApp-based Data Storage System Albin Stjerna Institutionen för informationsteknologi Department of
 DEIM Forum 2018 F3-5 657 8501 1-1 657 8501 1-1 E-mail: yuta@cs25.scitec.kobe-u.ac.jp, eguchi@port.kobe-u.ac.jp, ( ) ( )..,,,.,.,.,,..,.,,, 2..., 1.,., (Autoencoder: AE) [1] (Generative Stochastic Networks:
DEIM Forum 2018 F3-5 657 8501 1-1 657 8501 1-1 E-mail: yuta@cs25.scitec.kobe-u.ac.jp, eguchi@port.kobe-u.ac.jp, ( ) ( )..,,,.,.,.,,..,.,,, 2..., 1.,., (Autoencoder: AE) [1] (Generative Stochastic Networks:
Vol. 31,No JOURNAL OF CHINA UNIVERSITY OF SCIENCE AND TECHNOLOGY Feb
 Ξ 31 Vol 31,No 1 2 0 0 1 2 JOURNAL OF CHINA UNIVERSITY OF SCIENCE AND TECHNOLOGY Feb 2 0 0 1 :025322778 (2001) 0120016205 (, 230026) : Q ( m 1, m 2,, m n ) k = m 1 + m 2 + + m n - n : Q ( m 1, m 2,, m
Ξ 31 Vol 31,No 1 2 0 0 1 2 JOURNAL OF CHINA UNIVERSITY OF SCIENCE AND TECHNOLOGY Feb 2 0 0 1 :025322778 (2001) 0120016205 (, 230026) : Q ( m 1, m 2,, m n ) k = m 1 + m 2 + + m n - n : Q ( m 1, m 2,, m
{takasu, Conditional Random Field
 DEIM Forum 2016 C8-6 CRF 700 8530 3 1 1 700 8530 3 1 1 101 8430 2-1-2 E-mail: pobp52cw@s.okayama-u.ac.jp, ohta@de.cs.okayama-u.ac.jp, {takasu, adachi}@nii.ac.jp Conditional Random Field 1. Conditional
DEIM Forum 2016 C8-6 CRF 700 8530 3 1 1 700 8530 3 1 1 101 8430 2-1-2 E-mail: pobp52cw@s.okayama-u.ac.jp, ohta@de.cs.okayama-u.ac.jp, {takasu, adachi}@nii.ac.jp Conditional Random Field 1. Conditional
Οι απόψεις και τα συμπεράσματα που περιέχονται σε αυτό το έγγραφο, εκφράζουν τον συγγραφέα και δεν πρέπει να ερμηνευτεί ότι αντιπροσωπεύουν τις
 Οι απόψεις και τα συμπεράσματα που περιέχονται σε αυτό το έγγραφο, εκφράζουν τον συγγραφέα και δεν πρέπει να ερμηνευτεί ότι αντιπροσωπεύουν τις επίσημες θέσεις των εξεταστών. i ΠΡΟΛΟΓΟΣ ΕΥΧΑΡΙΣΤΙΕΣ Η παρούσα
Οι απόψεις και τα συμπεράσματα που περιέχονται σε αυτό το έγγραφο, εκφράζουν τον συγγραφέα και δεν πρέπει να ερμηνευτεί ότι αντιπροσωπεύουν τις επίσημες θέσεις των εξεταστών. i ΠΡΟΛΟΓΟΣ ΕΥΧΑΡΙΣΤΙΕΣ Η παρούσα
Anomaly Detection with Neighborhood Preservation Principle
 27 27 Workshop on Information-Based Induction Sciences (IBIS27) Tokyo, Japan, November 5-7, 27. Anomaly Detection with Neighborhood Preservation Principle Tsuyoshi Idé Abstract: We consider a task of anomaly
27 27 Workshop on Information-Based Induction Sciences (IBIS27) Tokyo, Japan, November 5-7, 27. Anomaly Detection with Neighborhood Preservation Principle Tsuyoshi Idé Abstract: We consider a task of anomaly
ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΥΠΡΟΥ ΤΜΗΜΑ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΥΠΡΟΥ ΤΜΗΜΑ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ (ΒΙΟ 650) Ειδικά Θέματα Βιοπληροφορικής Διδάσκων: Βασίλειος Ι. Προμπονάς, Ph.D. Λέκτορας Βιοπληροφορικής ΓΕΝΙΚΕΣ ΠΛΗΡΟΦΟΡΙΕΣ Διαλέξεις Δευτέρα και Πέμπτη
ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΥΠΡΟΥ ΤΜΗΜΑ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ (ΒΙΟ 650) Ειδικά Θέματα Βιοπληροφορικής Διδάσκων: Βασίλειος Ι. Προμπονάς, Ph.D. Λέκτορας Βιοπληροφορικής ΓΕΝΙΚΕΣ ΠΛΗΡΟΦΟΡΙΕΣ Διαλέξεις Δευτέρα και Πέμπτη
Optimization, PSO) DE [1, 2, 3, 4] PSO [5, 6, 7, 8, 9, 10, 11] (P)
![Optimization, PSO) DE [1, 2, 3, 4] PSO [5, 6, 7, 8, 9, 10, 11] (P) Optimization, PSO) DE [1, 2, 3, 4] PSO [5, 6, 7, 8, 9, 10, 11] (P)](/thumbs/85/92153760.jpg) ( ) 1 ( ) : : (Differential Evolution, DE) (Particle Swarm Optimization, PSO) DE [1, 2, 3, 4] PSO [5, 6, 7, 8, 9, 10, 11] 2 2.1 (P) (P ) minimize f(x) subject to g j (x) 0, j = 1,..., q h j (x) = 0, j
( ) 1 ( ) : : (Differential Evolution, DE) (Particle Swarm Optimization, PSO) DE [1, 2, 3, 4] PSO [5, 6, 7, 8, 9, 10, 11] 2 2.1 (P) (P ) minimize f(x) subject to g j (x) 0, j = 1,..., q h j (x) = 0, j
3: A convolution-pooling layer in PS-CNN 1: Partially Shared Deep Neural Network 2.2 Partially Shared Convolutional Neural Network 2: A hidden layer o
 Sound Source Identification based on Deep Learning with Partially-Shared Architecture 1 2 1 1,3 Takayuki MORITO 1, Osamu SUGIYAMA 2, Ryosuke KOJIMA 1, Kazuhiro NAKADAI 1,3 1 2 ( ) 3 Tokyo Institute of
Sound Source Identification based on Deep Learning with Partially-Shared Architecture 1 2 1 1,3 Takayuki MORITO 1, Osamu SUGIYAMA 2, Ryosuke KOJIMA 1, Kazuhiro NAKADAI 1,3 1 2 ( ) 3 Tokyo Institute of
Βιοπληροφορική. Ενότητα 21: Υπολογιστικός Προσδιορισμός Δομής (3/3), 1 ΔΩ. Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου
 Βιοπληροφορική Ενότητα 21: Υπολογιστικός Προσδιορισμός Δομής (3/3), 1 ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι επισκόπηση των μεθόδων αναγνώρισης διπλώματος και απ αρχής πρόγνωσης
Βιοπληροφορική Ενότητα 21: Υπολογιστικός Προσδιορισμός Δομής (3/3), 1 ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι επισκόπηση των μεθόδων αναγνώρισης διπλώματος και απ αρχής πρόγνωσης
A Systematic Review of Procalcitonin for Early Detection of Septicemia of Newborn
 56 [ ] 167-3619(009)03-056-05 Journal of Tropical Medicine Vol.9 No.3 Mar.009 * ( 510515) (PCT) CNKI VIP PCT QUADAS Metadisc1.4 DOR (SROC) SROC (AUC) Review Manager5 Meta 8 ; 7 8 (χ =3.8P=0.858I =0.0%)
56 [ ] 167-3619(009)03-056-05 Journal of Tropical Medicine Vol.9 No.3 Mar.009 * ( 510515) (PCT) CNKI VIP PCT QUADAS Metadisc1.4 DOR (SROC) SROC (AUC) Review Manager5 Meta 8 ; 7 8 (χ =3.8P=0.858I =0.0%)
Molecular evolutionary dynamics of respiratory syncytial virus group A in
 Molecular evolutionary dynamics of respiratory syncytial virus group A in recurrent epidemics in coastal Kenya James R. Otieno 1#, Charles N. Agoti 1, 2, Caroline W. Gitahi 1, Ann Bett 1, Mwanajuma Ngama
Molecular evolutionary dynamics of respiratory syncytial virus group A in recurrent epidemics in coastal Kenya James R. Otieno 1#, Charles N. Agoti 1, 2, Caroline W. Gitahi 1, Ann Bett 1, Mwanajuma Ngama
Quick algorithm f or computing core attribute
 24 5 Vol. 24 No. 5 Cont rol an d Decision 2009 5 May 2009 : 100120920 (2009) 0520738205 1a, 2, 1b (1. a., b., 239012 ; 2., 230039) :,,.,.,. : ; ; ; : TP181 : A Quick algorithm f or computing core attribute
24 5 Vol. 24 No. 5 Cont rol an d Decision 2009 5 May 2009 : 100120920 (2009) 0520738205 1a, 2, 1b (1. a., b., 239012 ; 2., 230039) :,,.,.,. : ; ; ; : TP181 : A Quick algorithm f or computing core attribute
Web-based supplementary materials for Bayesian Quantile Regression for Ordinal Longitudinal Data
 Web-based supplementary materials for Bayesian Quantile Regression for Ordinal Longitudinal Data Rahim Alhamzawi, Haithem Taha Mohammad Ali Department of Statistics, College of Administration and Economics,
Web-based supplementary materials for Bayesian Quantile Regression for Ordinal Longitudinal Data Rahim Alhamzawi, Haithem Taha Mohammad Ali Department of Statistics, College of Administration and Economics,
EM Baum-Welch. Step by Step the Baum-Welch Algorithm and its Application 2. HMM Baum-Welch. Baum-Welch. Baum-Welch Baum-Welch.
 Baum-Welch Step by Step the Baum-Welch Algorithm and its Application Jin ichi MURAKAMI EM EM EM Baum-Welch Baum-Welch Baum-Welch Baum-Welch, EM 1. EM 2. HMM EM (Expectationmaximization algorithm) 1 3.
Baum-Welch Step by Step the Baum-Welch Algorithm and its Application Jin ichi MURAKAMI EM EM EM Baum-Welch Baum-Welch Baum-Welch Baum-Welch, EM 1. EM 2. HMM EM (Expectationmaximization algorithm) 1 3.
2016 IEEE/ACM International Conference on Mobile Software Engineering and Systems
 2016 IEEE/ACM International Conference on Mobile Software Engineering and Systems Multiple User Interfaces MobileSoft'16, Multi-User Experience (MUX) S1: Insourcing S2: Outsourcing S3: Responsive design
2016 IEEE/ACM International Conference on Mobile Software Engineering and Systems Multiple User Interfaces MobileSoft'16, Multi-User Experience (MUX) S1: Insourcing S2: Outsourcing S3: Responsive design
Estimation, Evaluation and Guarantee of the Reverberant Speech Recognition Performance based on Room Acoustic Parameters
 Vol.21-SLP-83 No.9 21/1/29 1 Estimation, Evaluation and Guarantee of the Reverberant Speech Recognition Performance based on Room Acoustic Parameters Takanobu Nishiura 1 We study on estimation, evaluation
Vol.21-SLP-83 No.9 21/1/29 1 Estimation, Evaluation and Guarantee of the Reverberant Speech Recognition Performance based on Room Acoustic Parameters Takanobu Nishiura 1 We study on estimation, evaluation
Present and Future Prospects of Protein Structure Prediction
 1 Present and Future Prospects of Protein Structure Prediction Sung-Joon Park George Chikenji Takatsugu Hirokawa Kentaro Tomii Shoji Takada Department of Chemistry, Faculty of Science, Kobe University
1 Present and Future Prospects of Protein Structure Prediction Sung-Joon Park George Chikenji Takatsugu Hirokawa Kentaro Tomii Shoji Takada Department of Chemistry, Faculty of Science, Kobe University
Βιοπληροφορική. Ενότητα 14: Μοντέλα Πολλαπλής Στοίχισης (2/2), 1.5ΔΩ. Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου
 Βιοπληροφορική Ενότητα 14: Μοντέλα Πολλαπλής Στοίχισης (2/2), 1.5ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι παρουσίαση των μοντέλων πολλαπλής στοίχισης. κατανόηση των εφαρμογών
Βιοπληροφορική Ενότητα 14: Μοντέλα Πολλαπλής Στοίχισης (2/2), 1.5ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι παρουσίαση των μοντέλων πολλαπλής στοίχισης. κατανόηση των εφαρμογών
 DEIM Forum 2 D3-6 819 39 744 66 8 E-mail: kawamoto@inf.kyushu-u.ac.jp, tawara@db.soc.i.kyoto-u.ac.jp, {asano,yoshikawa}@i.kyoto-u.ac.jp 1.,, Amazon.com The Internet Movie Database (IMDb) 1 Social spammers
DEIM Forum 2 D3-6 819 39 744 66 8 E-mail: kawamoto@inf.kyushu-u.ac.jp, tawara@db.soc.i.kyoto-u.ac.jp, {asano,yoshikawa}@i.kyoto-u.ac.jp 1.,, Amazon.com The Internet Movie Database (IMDb) 1 Social spammers
ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΥΠΡΟΥ ΤΜΗΜΑ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΥΠΡΟΥ ΤΜΗΜΑ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ (ΒΙΟ 650) Ειδικά Θέματα Βιοπληροφορικής Διδάσκων: Βασίλειος Ι. Προμπονάς, Ph.D. Επίκουρος Καθηγητής Βιοπληροφορικής ΓΕΝΙΚΕΣ ΠΛΗΡΟΦΟΡΙΕΣ Διαλέξεις Δευτέρα
ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΥΠΡΟΥ ΤΜΗΜΑ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ (ΒΙΟ 650) Ειδικά Θέματα Βιοπληροφορικής Διδάσκων: Βασίλειος Ι. Προμπονάς, Ph.D. Επίκουρος Καθηγητής Βιοπληροφορικής ΓΕΝΙΚΕΣ ΠΛΗΡΟΦΟΡΙΕΣ Διαλέξεις Δευτέρα
ΚΩΝΣΤΑΝΤΙΝΟΣ Σ. ΠΟΛΙΤΗΣ Διπλ. Φυσικός Πανεπιστημίου Πατρών Υποψήφιος Διδάκτωρ Ε.Μ.Π. ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ
 ΚΩΝΣΤΑΝΤΙΝΟΣ Σ. ΠΟΛΙΤΗΣ Διπλ. Φυσικός Πανεπιστημίου Πατρών Υποψήφιος Διδάκτωρ Ε.Μ.Π. ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ 1. ΒΙΟΓΡΑΦΙΚΑ ΣΤΟΙΧΕΙΑ 1.1 ΠΡΟΣΩΠΙΚΑ ΣΤΟΙΧΕΙΑ Επώνυμο ΠΟΛΙΤΗΣ Όνομα Όνομα πατρός Διεύθυνση Ηλ. διεύθυνση
ΚΩΝΣΤΑΝΤΙΝΟΣ Σ. ΠΟΛΙΤΗΣ Διπλ. Φυσικός Πανεπιστημίου Πατρών Υποψήφιος Διδάκτωρ Ε.Μ.Π. ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ 1. ΒΙΟΓΡΑΦΙΚΑ ΣΤΟΙΧΕΙΑ 1.1 ΠΡΟΣΩΠΙΚΑ ΣΤΟΙΧΕΙΑ Επώνυμο ΠΟΛΙΤΗΣ Όνομα Όνομα πατρός Διεύθυνση Ηλ. διεύθυνση
Βιογραφικό Σημείωμα. Διεύθυνση επικοινωνίας: Τμήμα Μαθηματικών, Πανεπιστήμιο Πατρών
 Βιογραφικό Σημείωμα Προσωπικά στοιχεία Όνομα: Σταύρος Επώνυμο: Κουρούκλης Έτος γέννησης: 1952 Τόπος γέννησης: Ληξούρι Κεφαλλονιάς Στρατιωτική θητεία: Φεβρουάριος 2002 Οκτώβριος 2003 Οικογενειακή κατάσταση:
Βιογραφικό Σημείωμα Προσωπικά στοιχεία Όνομα: Σταύρος Επώνυμο: Κουρούκλης Έτος γέννησης: 1952 Τόπος γέννησης: Ληξούρι Κεφαλλονιάς Στρατιωτική θητεία: Φεβρουάριος 2002 Οκτώβριος 2003 Οικογενειακή κατάσταση:
Statistics 104: Quantitative Methods for Economics Formula and Theorem Review
 Harvard College Statistics 104: Quantitative Methods for Economics Formula and Theorem Review Tommy MacWilliam, 13 tmacwilliam@college.harvard.edu March 10, 2011 Contents 1 Introduction to Data 5 1.1 Sample
Harvard College Statistics 104: Quantitative Methods for Economics Formula and Theorem Review Tommy MacWilliam, 13 tmacwilliam@college.harvard.edu March 10, 2011 Contents 1 Introduction to Data 5 1.1 Sample
MIDI [8] MIDI. [9] Hsu [1], [2] [10] Salamon [11] [5] Song [6] Sony, Minato, Tokyo , Japan a) b)
![MIDI [8] MIDI. [9] Hsu [1], [2] [10] Salamon [11] [5] Song [6] Sony, Minato, Tokyo , Japan a) b) MIDI [8] MIDI. [9] Hsu [1], [2] [10] Salamon [11] [5] Song [6] Sony, Minato, Tokyo , Japan a) b)](/thumbs/93/113459486.jpg) 1,a) 1,b) 1,c) 1. MIDI [1], [2] U/D/S 3 [3], [4] 1 [5] Song [6] 1 Sony, Minato, Tokyo 108 0075, Japan a) Emiru.Tsunoo@jp.sony.com b) AkiraB.Inoue@jp.sony.com c) Masayuki.Nishiguchi@jp.sony.com MIDI [7]
1,a) 1,b) 1,c) 1. MIDI [1], [2] U/D/S 3 [3], [4] 1 [5] Song [6] 1 Sony, Minato, Tokyo 108 0075, Japan a) Emiru.Tsunoo@jp.sony.com b) AkiraB.Inoue@jp.sony.com c) Masayuki.Nishiguchi@jp.sony.com MIDI [7]
Chalkou I. C. [PROJECT] Ανάθεση εργασιών.
![Chalkou I. C. [PROJECT] Ανάθεση εργασιών. Chalkou I. C. [PROJECT] Ανάθεση εργασιών.](/thumbs/29/13219375.jpg) Πληροφορική της Υγείας 2014 Chalkou I. C. [PROJECT] Ανάθεση εργασιών. Περιεχόμενα 1. Ομάδα ΣΤ... 3 1.1 ΜΑΡΚΟΠΟΥΛΟΥ- ΣΠΥΡΟΠΟΥΛΟΥ -ΚΩΝΣΤΑΝΤΟΠΟΥΛΟΥ... 3 1.2 ΜΑΡΚΟΣ- ΚΟΥΤΣΟΠΟΥΛΟΣ ΑΥΓΕΡΗ - ΜΠΟΥΖΑΛΑ... 3 1.3
Πληροφορική της Υγείας 2014 Chalkou I. C. [PROJECT] Ανάθεση εργασιών. Περιεχόμενα 1. Ομάδα ΣΤ... 3 1.1 ΜΑΡΚΟΠΟΥΛΟΥ- ΣΠΥΡΟΠΟΥΛΟΥ -ΚΩΝΣΤΑΝΤΟΠΟΥΛΟΥ... 3 1.2 ΜΑΡΚΟΣ- ΚΟΥΤΣΟΠΟΥΛΟΣ ΑΥΓΕΡΗ - ΜΠΟΥΖΑΛΑ... 3 1.3
Chapter 1 Introduction to Observational Studies Part 2 Cross-Sectional Selection Bias Adjustment
 Contents Preface ix Part 1 Introduction Chapter 1 Introduction to Observational Studies... 3 1.1 Observational vs. Experimental Studies... 3 1.2 Issues in Observational Studies... 5 1.3 Study Design...
Contents Preface ix Part 1 Introduction Chapter 1 Introduction to Observational Studies... 3 1.1 Observational vs. Experimental Studies... 3 1.2 Issues in Observational Studies... 5 1.3 Study Design...
Κατηγοριοποίηση. 3 ο Φροντιστήριο. Ε Ξ Ό Ρ Υ Ξ Η Δ Ε Δ Ο Μ Έ Ν Ω Ν Κ Α Ι Α Λ Γ Ό Ρ Ι Θ Μ Ο Ι Μ Ά Θ Η Σ Η ς. Σκούρα Αγγελική
 Κατηγοριοποίηση Ε Ξ Ό Ρ Υ Ξ Η Δ Ε Δ Ο Μ Έ Ν Ω Ν Κ Α Ι Α Λ Γ Ό Ρ Ι Θ Μ Ο Ι Μ Ά Θ Η Σ Η ς 3 ο Φροντιστήριο Σκούρα Αγγελική skoura@ceid.upatras.gr Κατηγοριοποίηση (Classification) Σκοπός: Learn a method for
Κατηγοριοποίηση Ε Ξ Ό Ρ Υ Ξ Η Δ Ε Δ Ο Μ Έ Ν Ω Ν Κ Α Ι Α Λ Γ Ό Ρ Ι Θ Μ Ο Ι Μ Ά Θ Η Σ Η ς 3 ο Φροντιστήριο Σκούρα Αγγελική skoura@ceid.upatras.gr Κατηγοριοποίηση (Classification) Σκοπός: Learn a method for
ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ ΠΡΟΣΩΠΙΚΑ ΣΤΟΙΧΕΙΑ ΣΠΟΥΔΕΣ
 ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ ΠΡΟΣΩΠΙΚΑ ΣΤΟΙΧΕΙΑ ΕΠΩΝΥΜΟ: ΡΟΜΠΟΛΗΣ ΟΝΟΜΑ: ΛΕΩΝΙΔΑΣ ΟΝΟΜΑ ΠΑΤΡΟΣ: ΣΑΒΒΑΣ ΧΡΟΝΟΛΟΓΙΑ ΓΕΝΝΗΣΗΣ: 16/1/1977 ΤΟΠΟΣ ΓΕΝΝΗΣΗΣ: ΑΘΗΝΑ ΔΙΕΥΘΥΝΣΗ ΚΑΤΟΙΚΙΑΣ: ΟΙΚΟΝΟΜΟΥ 29, 16122, ΑΘΗΝΑ ΔΙΕΥΘΥΝΣΗ
ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ ΠΡΟΣΩΠΙΚΑ ΣΤΟΙΧΕΙΑ ΕΠΩΝΥΜΟ: ΡΟΜΠΟΛΗΣ ΟΝΟΜΑ: ΛΕΩΝΙΔΑΣ ΟΝΟΜΑ ΠΑΤΡΟΣ: ΣΑΒΒΑΣ ΧΡΟΝΟΛΟΓΙΑ ΓΕΝΝΗΣΗΣ: 16/1/1977 ΤΟΠΟΣ ΓΕΝΝΗΣΗΣ: ΑΘΗΝΑ ΔΙΕΥΘΥΝΣΗ ΚΑΤΟΙΚΙΑΣ: ΟΙΚΟΝΟΜΟΥ 29, 16122, ΑΘΗΝΑ ΔΙΕΥΘΥΝΣΗ
[15], [16], [17] [6] [2] [5] Jiang [6] 2.1 [6], [10] Score(x, y) y ( 1) ( 1 ) b e ( 1 ) b e. O(n 2 ) 2.3. 2.2 Jiang [6] (word lattice reranking)
![[15], [16], [17] [6] [2] [5] Jiang [6] 2.1 [6], [10] Score(x, y) y ( 1) ( 1 ) b e ( 1 ) b e. O(n 2 ) 2.3. 2.2 Jiang [6] (word lattice reranking) [15], [16], [17] [6] [2] [5] Jiang [6] 2.1 [6], [10] Score(x, y) y ( 1) ( 1 ) b e ( 1 ) b e. O(n 2 ) 2.3. 2.2 Jiang [6] (word lattice reranking)](/thumbs/39/20145561.jpg) 1,a) 1 2 10 1. [6] [1], [6], [8], [10], [11] 2 n n+1 C 2 O(n 2 ) 1 153-8505 4-6-1 a) kaji@tkl.iis.u-tokyo.ac.jp [10] [19], [23] [6] [6] (3 ) 10 (1) (2) 3 c 2012 Information Processing Society of Japan
1,a) 1 2 10 1. [6] [1], [6], [8], [10], [11] 2 n n+1 C 2 O(n 2 ) 1 153-8505 4-6-1 a) kaji@tkl.iis.u-tokyo.ac.jp [10] [19], [23] [6] [6] (3 ) 10 (1) (2) 3 c 2012 Information Processing Society of Japan
Δομές Δεδομένων. Δημήτρης Μιχαήλ. Συμβολοσειρές. Τμήμα Πληροφορικής και Τηλεματικής Χαροκόπειο Πανεπιστήμιο
 Δομές Δεδομένων Συμβολοσειρές Δημήτρης Μιχαήλ Τμήμα Πληροφορικής και Τηλεματικής Χαροκόπειο Πανεπιστήμιο Συμβολοσειρές Συμβολοσειρές και προβλήματα που αφορούν συμβολοσειρές εμφανίζονται τόσο συχνά που
Δομές Δεδομένων Συμβολοσειρές Δημήτρης Μιχαήλ Τμήμα Πληροφορικής και Τηλεματικής Χαροκόπειο Πανεπιστήμιο Συμβολοσειρές Συμβολοσειρές και προβλήματα που αφορούν συμβολοσειρές εμφανίζονται τόσο συχνά που
ΔΙΠΛΩΜΑΤΙΚΕΣ ΕΡΓΑΣΙΕΣ
 ΔΙΠΛΩΜΑΤΙΚΕΣ ΕΡΓΑΣΙΕΣ ΤΜ. ΜΗΧΑΝΙΚΩΝ ΠΛΗΡΟΦΟΡΙΚΗΣ & ΤΗΛΕΠΙΚΟΙΝΩΝΙΩΝ 2018-2019 Επιβλέπουσα: Μπίμπη Ματίνα Ανάλυση της πλατφόρμας ανοιχτού κώδικα Home Assistant Το Home Assistant είναι μία πλατφόρμα ανοιχτού
ΔΙΠΛΩΜΑΤΙΚΕΣ ΕΡΓΑΣΙΕΣ ΤΜ. ΜΗΧΑΝΙΚΩΝ ΠΛΗΡΟΦΟΡΙΚΗΣ & ΤΗΛΕΠΙΚΟΙΝΩΝΙΩΝ 2018-2019 Επιβλέπουσα: Μπίμπη Ματίνα Ανάλυση της πλατφόρμας ανοιχτού κώδικα Home Assistant Το Home Assistant είναι μία πλατφόρμα ανοιχτού
ΣΥΝΔΥΑΣΤΙΚΗ ΒΕΛΤΙΣΤΟΠΟΙΗΣΗ
 Ενότητα Τμήμα Εφαρμοσμένης Πληροφορικής Άδειες Χρήσης Το παρόν εκπαιδευτικό υλικό υπόκειται σε άδειες χρήσης Creative Commons. Για εκπαιδευτικό υλικό, όπως εικόνες, που υπόκειται σε άλλου τύπου άδειας
Ενότητα Τμήμα Εφαρμοσμένης Πληροφορικής Άδειες Χρήσης Το παρόν εκπαιδευτικό υλικό υπόκειται σε άδειες χρήσης Creative Commons. Για εκπαιδευτικό υλικό, όπως εικόνες, που υπόκειται σε άλλου τύπου άδειας
ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ. Παναγιώτης Μερκούρης ΕΚΠΑΙΔΕΥΣΗ
 ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ Παναγιώτης Μερκούρης ΕΚΠΑΙΔΕΥΣΗ Πρώτο Πτυχίο: Μαθηματικά, 1979 Εθνικό Καποδιστριακό Πανεπιστήμιο Αθηνών, Τμήμα Μαθηματικών MSc: Στατιστική, 1983 McGill University, Department of Mathematics
ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ Παναγιώτης Μερκούρης ΕΚΠΑΙΔΕΥΣΗ Πρώτο Πτυχίο: Μαθηματικά, 1979 Εθνικό Καποδιστριακό Πανεπιστήμιο Αθηνών, Τμήμα Μαθηματικών MSc: Στατιστική, 1983 McGill University, Department of Mathematics
ER-Tree (Extended R*-Tree)
 1-9825/22/13(4)768-6 22 Journal of Software Vol13, No4 1, 1, 2, 1 1, 1 (, 2327) 2 (, 3127) E-mail xhzhou@ustceducn,,,,,,, 1, TP311 A,,,, Elias s Rivest,Cleary Arya Mount [1] O(2 d ) Arya Mount [1] Friedman,Bentley
1-9825/22/13(4)768-6 22 Journal of Software Vol13, No4 1, 1, 2, 1 1, 1 (, 2327) 2 (, 3127) E-mail xhzhou@ustceducn,,,,,,, 1, TP311 A,,,, Elias s Rivest,Cleary Arya Mount [1] O(2 d ) Arya Mount [1] Friedman,Bentley
ΓΙΑΝΝΟΥΛΑ Σ. ΦΛΩΡΟΥ Ι ΑΚΤΟΡΑΣ ΤΟΥ ΤΜΗΜΑΤΟΣ ΕΦΑΡΜΟΣΜΕΝΗΣ ΠΛΗΡΟΦΟΡΙΚΗΣ ΤΟΥ ΠΑΝΕΠΙΣΤΗΜΙΟΥ ΜΑΚΕ ΟΝΙΑΣ ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ
 ΓΙΑΝΝΟΥΛΑ Σ. ΦΛΩΡΟΥ Ι ΑΚΤΟΡΑΣ ΤΟΥ ΤΜΗΜΑΤΟΣ ΕΦΑΡΜΟΣΜΕΝΗΣ ΠΛΗΡΟΦΟΡΙΚΗΣ ΤΟΥ ΠΑΝΕΠΙΣΤΗΜΙΟΥ ΜΑΚΕ ΟΝΙΑΣ ΑΝΑΠΛΗΡΩΤΡΙΑ ΚΑΘΗΓΗΤΡΙΑ ΤΟΥ ΤΜΗΜΑΤΟΣ ΛΟΓΙΣΤΙΚΗΣ ΤΟΥ ΤΕΙ ΚΑΒΑΛΑΣ ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ ΙΑΝΟΥΑΡΙΟΣ 2008 ΒΙΟΓΡΑΦΙΚΟ
ΓΙΑΝΝΟΥΛΑ Σ. ΦΛΩΡΟΥ Ι ΑΚΤΟΡΑΣ ΤΟΥ ΤΜΗΜΑΤΟΣ ΕΦΑΡΜΟΣΜΕΝΗΣ ΠΛΗΡΟΦΟΡΙΚΗΣ ΤΟΥ ΠΑΝΕΠΙΣΤΗΜΙΟΥ ΜΑΚΕ ΟΝΙΑΣ ΑΝΑΠΛΗΡΩΤΡΙΑ ΚΑΘΗΓΗΤΡΙΑ ΤΟΥ ΤΜΗΜΑΤΟΣ ΛΟΓΙΣΤΙΚΗΣ ΤΟΥ ΤΕΙ ΚΑΒΑΛΑΣ ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ ΙΑΝΟΥΑΡΙΟΣ 2008 ΒΙΟΓΡΑΦΙΚΟ
ΜΕΘΟΔΟΙ ΥΠΟΛΟΓΙΣΜΟΥ ΤΗΣ ΖΕΝΙΘΕΙΑΣ ΤΡΟΠΟΣΦΑΙΡΙΚΗΣ ΥΣΤΕΡΗΣΗΣ ΣΕ ΜΟΝΙΜΟΥΣ ΣΤΑΘΜΟΥΣ GNSS
 ΤΟΜΕΑΣ ΓΕΩΔΑΙΣΙΑΣ ΚΑΙ ΤΟΠΟΓΡΑΦΙΑΣ ΤΕΙ ΣΕΡΡΩΝ, ΣΧΟΛΗ ΤΕΧΝΟΛΟΓΙΚΩΝ ΕΦΑΡΜΟΓΩΝ Τμήμα Γεωπληροφορικής & Τοπογραφίας ΜΕΘΟΔΟΙ ΥΠΟΛΟΓΙΣΜΟΥ ΤΗΣ ΖΕΝΙΘΕΙΑΣ ΤΡΟΠΟΣΦΑΙΡΙΚΗΣ ΥΣΤΕΡΗΣΗΣ ΣΕ ΜΟΝΙΜΟΥΣ ΣΤΑΘΜΟΥΣ GNSS ΣΥΜΕΩΝ
ΤΟΜΕΑΣ ΓΕΩΔΑΙΣΙΑΣ ΚΑΙ ΤΟΠΟΓΡΑΦΙΑΣ ΤΕΙ ΣΕΡΡΩΝ, ΣΧΟΛΗ ΤΕΧΝΟΛΟΓΙΚΩΝ ΕΦΑΡΜΟΓΩΝ Τμήμα Γεωπληροφορικής & Τοπογραφίας ΜΕΘΟΔΟΙ ΥΠΟΛΟΓΙΣΜΟΥ ΤΗΣ ΖΕΝΙΘΕΙΑΣ ΤΡΟΠΟΣΦΑΙΡΙΚΗΣ ΥΣΤΕΡΗΣΗΣ ΣΕ ΜΟΝΙΜΟΥΣ ΣΤΑΘΜΟΥΣ GNSS ΣΥΜΕΩΝ
CorV CVAC. CorV TU317. 1
 30 8 JOURNAL OF VIBRATION AND SHOCK Vol. 30 No. 8 2011 1 2 1 2 2 1. 100044 2. 361005 TU317. 1 A Structural damage detection method based on correlation function analysis of vibration measurement data LEI
30 8 JOURNAL OF VIBRATION AND SHOCK Vol. 30 No. 8 2011 1 2 1 2 2 1. 100044 2. 361005 TU317. 1 A Structural damage detection method based on correlation function analysis of vibration measurement data LEI
Area Location and Recognition of Video Text Based on Depth Learning Method
 21 6 2016 12 Vol 21 No 6 JOURNAL OF HARBIN UNIVERSITY OF SCIENCE AND TECHNOLOGY Dec 2016 1 1 1 2 1 150080 2 130300 Gabor RBM OCR DOI 10 15938 /j jhust 2016 06 012 TP391 43 A 1007-2683 2016 06-0061- 06
21 6 2016 12 Vol 21 No 6 JOURNAL OF HARBIN UNIVERSITY OF SCIENCE AND TECHNOLOGY Dec 2016 1 1 1 2 1 150080 2 130300 Gabor RBM OCR DOI 10 15938 /j jhust 2016 06 012 TP391 43 A 1007-2683 2016 06-0061- 06
ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ ΛΕΩΝΙΔΑΣ Α. ΣΠΥΡΟΥ. 2004 2009 Διδακτορικό σε Υπολογιστική Εμβιομηχανική, Τμήμα Μηχανολόγων Μηχανικών, Πανεπιστήμιο Θεσσαλίας.
 ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ ΛΕΩΝΙΔΑΣ Α. ΣΠΥΡΟΥ ΔΙΕΥΘΥΝΣΗ Ινστιτούτο Έρευνας και Τεχνολογίας Θεσσαλίας (ΙΕΤΕΘ) Εθνικό Κέντρο Έρευνας και Τεχνολογικής Ανάπτυξης (ΕΚΕΤΑ) Δημητριάδος 95 και Παύλου Μελά 38333 Βόλος
ΒΙΟΓΡΑΦΙΚΟ ΣΗΜΕΙΩΜΑ ΛΕΩΝΙΔΑΣ Α. ΣΠΥΡΟΥ ΔΙΕΥΘΥΝΣΗ Ινστιτούτο Έρευνας και Τεχνολογίας Θεσσαλίας (ΙΕΤΕΘ) Εθνικό Κέντρο Έρευνας και Τεχνολογικής Ανάπτυξης (ΕΚΕΤΑ) Δημητριάδος 95 και Παύλου Μελά 38333 Βόλος
No. 7 Modular Machine Tool & Automatic Manufacturing Technique. Jul TH166 TG659 A
 7 2016 7 No. 7 Modular Machine Tool & Automatic Manufacturing Technique Jul. 2016 1001-2265 2016 07-0122 - 05 DOI 10. 13462 /j. cnki. mmtamt. 2016. 07. 035 * 100124 TH166 TG659 A Precision Modeling and
7 2016 7 No. 7 Modular Machine Tool & Automatic Manufacturing Technique Jul. 2016 1001-2265 2016 07-0122 - 05 DOI 10. 13462 /j. cnki. mmtamt. 2016. 07. 035 * 100124 TH166 TG659 A Precision Modeling and
ΕΓΓΡΑΦΟ ΕΡΓΑΣΙΑΣ ΤΩΝ ΥΠΗΡΕΣΙΩΝ ΤΗΣ ΕΠΙΤΡΟΠΗΣ. Εκθεση χώρας - Κύπρος 2015. {COM(2015) 85 final}
 ΕΥΡΩΠΑΪΚΗ ΕΠΙΤΡΟΠΗ Βρυξέλλες, 26.2.2015 SWD(2015) 32 final ΕΓΓΡΑΦΟ ΕΡΓΑΣΙΑΣ ΤΩΝ ΥΠΗΡΕΣΙΩΝ ΤΗΣ ΕΠΙΤΡΟΠΗΣ Εκθεση χώρας - Κύπρος 2015 {COM(2015) 85 final} Το παρόν έγγραφο δεν συνιστά επίσημη θέση της Ευρωπαϊκής
ΕΥΡΩΠΑΪΚΗ ΕΠΙΤΡΟΠΗ Βρυξέλλες, 26.2.2015 SWD(2015) 32 final ΕΓΓΡΑΦΟ ΕΡΓΑΣΙΑΣ ΤΩΝ ΥΠΗΡΕΣΙΩΝ ΤΗΣ ΕΠΙΤΡΟΠΗΣ Εκθεση χώρας - Κύπρος 2015 {COM(2015) 85 final} Το παρόν έγγραφο δεν συνιστά επίσημη θέση της Ευρωπαϊκής
Newman Modularity Newman [4], [5] Newman Q Q Q greedy algorithm[6] Newman Newman Q 1 Tabu Search[7] Newman Newman Newman Q Newman 1 2 Newman 3
![Newman Modularity Newman [4], [5] Newman Q Q Q greedy algorithm[6] Newman Newman Q 1 Tabu Search[7] Newman Newman Newman Q Newman 1 2 Newman 3 Newman Modularity Newman [4], [5] Newman Q Q Q greedy algorithm[6] Newman Newman Q 1 Tabu Search[7] Newman Newman Newman Q Newman 1 2 Newman 3](/thumbs/50/25933291.jpg) DEWS2007 D3-6 y yy y y y y yy / DC 7313194 341 E-mail: yfktamura,mori,kuroki,kitakamig@its.hiroshima-cu.ac.jp, yymakoto@db.its.hiroshima-cu.ac.jp Newman Newman Newman Newman Newman A Clustering Algorithm
DEWS2007 D3-6 y yy y y y y yy / DC 7313194 341 E-mail: yfktamura,mori,kuroki,kitakamig@its.hiroshima-cu.ac.jp, yymakoto@db.its.hiroshima-cu.ac.jp Newman Newman Newman Newman Newman A Clustering Algorithm
Gemini, FastMap, Applications. Εαρινό Εξάμηνο Τμήμα Μηχανικών Η/Υ και Πληροϕορικής Πολυτεχνική Σχολή, Πανεπιστήμιο Πατρών
 Gemini,, Applications Τμήμα Μηχανικών Η/Υ και Πληροϕορικής Πολυτεχνική Σχολή, Πανεπιστήμιο Πατρών Εαρινό Εξάμηνο 2011-2012 Table of contents 1 Table of contents 1 2 Table of contents 1 2 3 Table of contents
Gemini,, Applications Τμήμα Μηχανικών Η/Υ και Πληροϕορικής Πολυτεχνική Σχολή, Πανεπιστήμιο Πατρών Εαρινό Εξάμηνο 2011-2012 Table of contents 1 Table of contents 1 2 Table of contents 1 2 3 Table of contents
Bayesian Discriminant Feature Selection
 1,a) 2 1... DNA. Lasso. Bayesian Discriminant Feature Selection Tanaka Yusuke 1,a) Ueda Naonori 2 Tanaka Toshiyuki 1 Abstract: Focusing on categorical data, we propose a Bayesian feature selection method
1,a) 2 1... DNA. Lasso. Bayesian Discriminant Feature Selection Tanaka Yusuke 1,a) Ueda Naonori 2 Tanaka Toshiyuki 1 Abstract: Focusing on categorical data, we propose a Bayesian feature selection method
Supplementary Materials for Evolutionary Multiobjective Optimization Based Multimodal Optimization: Fitness Landscape Approximation and Peak Detection
 IEEE TRANSACTIONS ON EVOLUTIONARY COMPUTATION, VOL. XX, NO. X, XXXX XXXX Supplementary Materials for Evolutionary Multiobjective Optimization Based Multimodal Optimization: Fitness Landscape Approximation
IEEE TRANSACTIONS ON EVOLUTIONARY COMPUTATION, VOL. XX, NO. X, XXXX XXXX Supplementary Materials for Evolutionary Multiobjective Optimization Based Multimodal Optimization: Fitness Landscape Approximation
 DEIM Forum 2014 A8-1, 606 8501 E-mail: {tsukuda,ohshima,kato,tanaka}@dl.kuis.kyoto-u.ac.jp 1 2,, 1. Google 1 Yahoo 2 Bing 3 Web Web BM25 [1] HITS [2] PageRank [3] Web 1 [4] 1http://www.google.com 2http://www.yahoo.com
DEIM Forum 2014 A8-1, 606 8501 E-mail: {tsukuda,ohshima,kato,tanaka}@dl.kuis.kyoto-u.ac.jp 1 2,, 1. Google 1 Yahoo 2 Bing 3 Web Web BM25 [1] HITS [2] PageRank [3] Web 1 [4] 1http://www.google.com 2http://www.yahoo.com
FORMULAS FOR STATISTICS 1
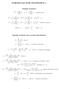 FORMULAS FOR STATISTICS 1 X = 1 n Sample statistics X i or x = 1 n x i (sample mean) S 2 = 1 n 1 s 2 = 1 n 1 (X i X) 2 = 1 n 1 (x i x) 2 = 1 n 1 Xi 2 n n 1 X 2 x 2 i n n 1 x 2 or (sample variance) E(X)
FORMULAS FOR STATISTICS 1 X = 1 n Sample statistics X i or x = 1 n x i (sample mean) S 2 = 1 n 1 s 2 = 1 n 1 (X i X) 2 = 1 n 1 (x i x) 2 = 1 n 1 Xi 2 n n 1 X 2 x 2 i n n 1 x 2 or (sample variance) E(X)
Ανάλυση σχημάτων βασισμένη σε μεθόδους αναζήτησης ομοιότητας υποακολουθιών (C589)
 Ανάλυση σχημάτων βασισμένη σε μεθόδους αναζήτησης ομοιότητας υποακολουθιών (C589) Μεγαλοοικονόμου Βασίλειος Τμήμα Μηχ. Η/ΥκαιΠληροφορικής Επιστημονικός Υπεύθυνος Στόχος Προτεινόμενου Έργου Ανάπτυξη μεθόδων
Ανάλυση σχημάτων βασισμένη σε μεθόδους αναζήτησης ομοιότητας υποακολουθιών (C589) Μεγαλοοικονόμου Βασίλειος Τμήμα Μηχ. Η/ΥκαιΠληροφορικής Επιστημονικός Υπεύθυνος Στόχος Προτεινόμενου Έργου Ανάπτυξη μεθόδων
Αποθήκες εδοµένων και Εξόρυξη Γνώσης (Data Warehousing & Data Mining)
 Πανεπιστήµιο Πειραιώς Τµήµα Πληροφορικής Αποθήκες εδοµένων και Εξόρυξη Γνώσης (Data Warehousing & Data Mining) Εξόρυξη Χρονικής Γνώσης (temporal data mining) Γιάννης Θεοδωρίδης, Νίκος Πελέκης Οµάδα ιαχείρισης
Πανεπιστήµιο Πειραιώς Τµήµα Πληροφορικής Αποθήκες εδοµένων και Εξόρυξη Γνώσης (Data Warehousing & Data Mining) Εξόρυξη Χρονικής Γνώσης (temporal data mining) Γιάννης Θεοδωρίδης, Νίκος Πελέκης Οµάδα ιαχείρισης
Βιοπληροφορική. Μαργαρίτα Θεοδωροπούλου. Πανεπιστήμιο Θεσσαλίας, Λαμία 2016
 Βιοπληροφορική Μαργαρίτα Θεοδωροπούλου Πανεπιστήμιο Θεσσαλίας, Λαμία 2016 Βιοπληροφορική Εισαγωγή στη Μοριακή Βιολογία, Γενωμική και Βιοπληροφορική. Βάσεις Βιολογικών Δεδομένων. Ακολουθίες Πρωτεϊνών και
Βιοπληροφορική Μαργαρίτα Θεοδωροπούλου Πανεπιστήμιο Θεσσαλίας, Λαμία 2016 Βιοπληροφορική Εισαγωγή στη Μοριακή Βιολογία, Γενωμική και Βιοπληροφορική. Βάσεις Βιολογικών Δεδομένων. Ακολουθίες Πρωτεϊνών και
Parameter Estimation of Stochastic Grammars with Probabilistic Logic Programs
 人工知能学会研究会資料 SIG-FPAI-B502-20 Parameter Estimation of Stochastic Grammars with Probabilistic Logic Programs 1 1 1 Satoru Yamaguchi 1 Ryo Yoshinaka 1 Akihiro Yamamoto 1 1 1 Graduate School of Informatics,
人工知能学会研究会資料 SIG-FPAI-B502-20 Parameter Estimation of Stochastic Grammars with Probabilistic Logic Programs 1 1 1 Satoru Yamaguchi 1 Ryo Yoshinaka 1 Akihiro Yamamoto 1 1 1 Graduate School of Informatics,
ΠΡΟΤΕΙΝΟΜΕΝΑ ΘΕΜΑΤΑ ΔΙΠΛΩΜΑΤΙΚΩΝ ΕΡΓΑΣΙΩΝ ΓΙΑ ΤΟ ΕΑΡΙΝΟ ΕΞΑΜΗΝΟ Εισηγητής: Νίκος Πλόσκας Επίκουρος Καθηγητής ΤΜΠΤ
 ΠΡΟΤΕΙΝΟΜΕΝΑ ΘΕΜΑΤΑ ΔΙΠΛΩΜΑΤΙΚΩΝ ΕΡΓΑΣΙΩΝ ΓΙΑ ΤΟ ΕΑΡΙΝΟ ΕΞΑΜΗΝΟ 2018-2019 Εισηγητής: Νίκος Πλόσκας (nploskas@uowm.gr), Επίκουρος Καθηγητής ΤΜΠΤ 1. Τεχνικές μηχανικής μάθησης για τον εντοπισμό προβλημάτων
ΠΡΟΤΕΙΝΟΜΕΝΑ ΘΕΜΑΤΑ ΔΙΠΛΩΜΑΤΙΚΩΝ ΕΡΓΑΣΙΩΝ ΓΙΑ ΤΟ ΕΑΡΙΝΟ ΕΞΑΜΗΝΟ 2018-2019 Εισηγητής: Νίκος Πλόσκας (nploskas@uowm.gr), Επίκουρος Καθηγητής ΤΜΠΤ 1. Τεχνικές μηχανικής μάθησης για τον εντοπισμό προβλημάτων
Analysis of Protein Structure in Silico
 49 20 : 49 53 2006 Analysis of Protein Structure in Silico Michihiro OGAWA* *Department of Microbiology, Kyoto Pharmaceutical University N 20 N 20 N 3 http://sosui.proteome.bio.tuat.ac.jp/ sosuiframe0.html
49 20 : 49 53 2006 Analysis of Protein Structure in Silico Michihiro OGAWA* *Department of Microbiology, Kyoto Pharmaceutical University N 20 N 20 N 3 http://sosui.proteome.bio.tuat.ac.jp/ sosuiframe0.html
Περιεχόµενα. 1. Γενικό πλαίσιο. 2. Η ΚΑΠ σήµερα. 3. Γιατί χρειαζόµαστε τη µεταρρύθµιση; 4. Νέοι στόχοι, µελλοντικά εργαλεία και πολιτικές επιλογές
 Ανακοίνωση για το µέλλον της ΚAΠ «Η ΚΑΠπροςτο2020: αντιµετωπίζοντας τις προκλήσεις στον τοµέα των τροφίµων, στους φυσικούς πόρους και στις περιφέρειες» Γ Γεωργίας και Αγροτικής Ανάπτυξης Ευρωπαϊκή Επιτροπή
Ανακοίνωση για το µέλλον της ΚAΠ «Η ΚΑΠπροςτο2020: αντιµετωπίζοντας τις προκλήσεις στον τοµέα των τροφίµων, στους φυσικούς πόρους και στις περιφέρειες» Γ Γεωργίας και Αγροτικής Ανάπτυξης Ευρωπαϊκή Επιτροπή
Bayesian modeling of inseparable space-time variation in disease risk
 Bayesian modeling of inseparable space-time variation in disease risk Leonhard Knorr-Held Laina Mercer Department of Statistics UW May, 013 Motivation Ohio Lung Cancer Example Lung Cancer Mortality Rates
Bayesian modeling of inseparable space-time variation in disease risk Leonhard Knorr-Held Laina Mercer Department of Statistics UW May, 013 Motivation Ohio Lung Cancer Example Lung Cancer Mortality Rates
CSJ. Speaker clustering based on non-negative matrix factorization using i-vector-based speaker similarity
 i-vector 1 1 1 1 i-vector CSJ i-vector Speaker clustering based on non-negative matrix factorization using i-vector-based speaker similarity Fukuchi Yusuke 1 Tawara Naohiro 1 Ogawa Tetsuji 1 Kobayashi
i-vector 1 1 1 1 i-vector CSJ i-vector Speaker clustering based on non-negative matrix factorization using i-vector-based speaker similarity Fukuchi Yusuke 1 Tawara Naohiro 1 Ogawa Tetsuji 1 Kobayashi
Δημήτριος Θ. Τόμτσης, Ph.D. Αναλυτικό Βιογραφικό Σημείωμα
 Δημήτριος Θ. Τόμτσης, Ph.D. Αναλυτικό Βιογραφικό Σημείωμα Τίτλοι σπουδών Πτυχίο: Μηχανικός Πληροφοριακών Συστημάτων, Πανεπιστήμιο Coventry, U.K,, 1991. Μεταπτυχιακό: Μηχανικός Τηλεπικοινωνιακών Συστημάτων
Δημήτριος Θ. Τόμτσης, Ph.D. Αναλυτικό Βιογραφικό Σημείωμα Τίτλοι σπουδών Πτυχίο: Μηχανικός Πληροφοριακών Συστημάτων, Πανεπιστήμιο Coventry, U.K,, 1991. Μεταπτυχιακό: Μηχανικός Τηλεπικοινωνιακών Συστημάτων
Bayesian statistics. DS GA 1002 Probability and Statistics for Data Science.
 Bayesian statistics DS GA 1002 Probability and Statistics for Data Science http://www.cims.nyu.edu/~cfgranda/pages/dsga1002_fall17 Carlos Fernandez-Granda Frequentist vs Bayesian statistics In frequentist
Bayesian statistics DS GA 1002 Probability and Statistics for Data Science http://www.cims.nyu.edu/~cfgranda/pages/dsga1002_fall17 Carlos Fernandez-Granda Frequentist vs Bayesian statistics In frequentist
A Method for Creating Shortcut Links by Considering Popularity of Contents in Structured P2P Networks
 P2P 1,a) 1 1 1 P2P P2P P2P P2P A Method for Creating Shortcut Links by Considering Popularity of Contents in Structured P2P Networks NARISHIGE Yuki 1,a) ABE Kota 1 ISHIBASHI Hayato 1 MATSUURA Toshio 1
P2P 1,a) 1 1 1 P2P P2P P2P P2P A Method for Creating Shortcut Links by Considering Popularity of Contents in Structured P2P Networks NARISHIGE Yuki 1,a) ABE Kota 1 ISHIBASHI Hayato 1 MATSUURA Toshio 1
High order interpolation function for surface contact problem
 3 016 5 Journal of East China Normal University Natural Science No 3 May 016 : 1000-564101603-0009-1 1 1 1 00444; E- 00030 : Lagrange Lobatto Matlab : ; Lagrange; : O41 : A DOI: 103969/jissn1000-56410160300
3 016 5 Journal of East China Normal University Natural Science No 3 May 016 : 1000-564101603-0009-1 1 1 1 00444; E- 00030 : Lagrange Lobatto Matlab : ; Lagrange; : O41 : A DOI: 103969/jissn1000-56410160300
substructure similarity search using features in graph databases
 substructure similarity search using features in graph databases Aleksandros Gkogkas Distributed Management of Data Laboratory intro Θα ενασχοληθούμε με το πρόβλημα των ερωτήσεων σε βάσεις γραφημάτων.
substructure similarity search using features in graph databases Aleksandros Gkogkas Distributed Management of Data Laboratory intro Θα ενασχοληθούμε με το πρόβλημα των ερωτήσεων σε βάσεις γραφημάτων.
Research on model of early2warning of enterprise crisis based on entropy
 24 1 Vol. 24 No. 1 ont rol an d Decision 2009 1 Jan. 2009 : 100120920 (2009) 0120113205 1, 1, 2 (1., 100083 ; 2., 100846) :. ;,,. 2.,,. : ; ; ; : F270. 5 : A Research on model of early2warning of enterprise
24 1 Vol. 24 No. 1 ont rol an d Decision 2009 1 Jan. 2009 : 100120920 (2009) 0120113205 1, 1, 2 (1., 100083 ; 2., 100846) :. ;,,. 2.,,. : ; ; ; : F270. 5 : A Research on model of early2warning of enterprise
794 Appendix A:Tables
 Appendix A Tables A Table Contents Page A.1 Random numbers 794 A.2 Orthogonal polynomial trend contrast coefficients 800 A.3 Standard normal distribution 801 A.4 Student s t-distribution 802 A.5 Chi-squared
Appendix A Tables A Table Contents Page A.1 Random numbers 794 A.2 Orthogonal polynomial trend contrast coefficients 800 A.3 Standard normal distribution 801 A.4 Student s t-distribution 802 A.5 Chi-squared
90 [, ] p Panel nested error structure) : Lagrange-multiple LM) Honda [3] LM ; King Wu, Baltagi, Chang Li [4] Moulton Randolph ANOVA) F p Panel,, p Z
![90 [, ] p Panel nested error structure) : Lagrange-multiple LM) Honda [3] LM ; King Wu, Baltagi, Chang Li [4] Moulton Randolph ANOVA) F p Panel,, p Z 90 [, ] p Panel nested error structure) : Lagrange-multiple LM) Honda [3] LM ; King Wu, Baltagi, Chang Li [4] Moulton Randolph ANOVA) F p Panel,, p Z](/thumbs/91/107678282.jpg) 00 Chinese Journal of Applied Probability and Statistics Vol6 No Feb 00 Panel, 3,, 0034;,, 38000) 3,, 000) p Panel,, p Panel : Panel,, p,, : O,,, nuisance parameter), Tsui Weerahandi [] Weerahandi [] p
00 Chinese Journal of Applied Probability and Statistics Vol6 No Feb 00 Panel, 3,, 0034;,, 38000) 3,, 000) p Panel,, p Panel : Panel,, p,, : O,,, nuisance parameter), Tsui Weerahandi [] Weerahandi [] p
Στοίχιση Ακολουθιών. Μέθοδοι σύγκρισης ακολουθιών. Είδος στοίχισης. match. gap. mismatch
 Οµολογία ΣΤΟΙΧΙΣΗ ΑΚΟΛΟΥΘΙΩΝ ΑΝΑ ΖΕΥΓΗ Σελίδα 1 Σελίδα 2 Οµολογία Οµολογία Οµολογία κοινή εξελικτική καταγωγή Ορθόλογα γονίδια ειδογένεση συνήθως, ίδια βιολογική λειτουργία Παράλογα γονίδια γονιδιακός
Οµολογία ΣΤΟΙΧΙΣΗ ΑΚΟΛΟΥΘΙΩΝ ΑΝΑ ΖΕΥΓΗ Σελίδα 1 Σελίδα 2 Οµολογία Οµολογία Οµολογία κοινή εξελικτική καταγωγή Ορθόλογα γονίδια ειδογένεση συνήθως, ίδια βιολογική λειτουργία Παράλογα γονίδια γονιδιακός
Βιοπληροφορική. Εισαγωγή. Αλέξανδρος Τζάλλας Σχολή Τεχνολογικών Εφαρμογών Τμήμα Μηχανικών Πληροφορικής ΤΕ.
 Βιοπληροφορική Αλέξανδρος Τζάλλας e-mail: tzallas@teiep.gr ΤΕΙ Ηπείρου Σχολή Τεχνολογικών Εφαρμογών Τμήμα Μηχανικών Πληροφορικής ΤΕ Περιεχόμενα Διάλεξης Βιολογικό υπόβαθρο Το κεντρικό αξίωμα Σύνοψη της
Βιοπληροφορική Αλέξανδρος Τζάλλας e-mail: tzallas@teiep.gr ΤΕΙ Ηπείρου Σχολή Τεχνολογικών Εφαρμογών Τμήμα Μηχανικών Πληροφορικής ΤΕ Περιεχόμενα Διάλεξης Βιολογικό υπόβαθρο Το κεντρικό αξίωμα Σύνοψη της
Βιοπληροφορική. Ενότητα 7: Στοίχιση ακολουθιών ανά ζεύγη Τεχνικές Στοίχισης Ακολουθιών, (1/2) 1ΔΩ. Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ.
 Βιοπληροφορική Ενότητα 7: Στοίχιση ακολουθιών ανά ζεύγη Τεχνικές Στοίχισης Ακολουθιών, (1/2) 1ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι Παρουσίαση της μεθόδου κατασκευής και
Βιοπληροφορική Ενότητα 7: Στοίχιση ακολουθιών ανά ζεύγη Τεχνικές Στοίχισης Ακολουθιών, (1/2) 1ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι Παρουσίαση της μεθόδου κατασκευής και
Βιοπληροφορική. Ενότητα 13: Μοντέλα Πολλαπλής Στοίχισης (1/2), 1.5ΔΩ. Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου
 Βιοπληροφορική Ενότητα 13: Μοντέλα Πολλαπλής Στοίχισης (1/2), 1.5ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι παρουσίαση των μοντέλων πολλαπλής στοίχισης. κατανόηση των εφαρμογών
Βιοπληροφορική Ενότητα 13: Μοντέλα Πολλαπλής Στοίχισης (1/2), 1.5ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι παρουσίαση των μοντέλων πολλαπλής στοίχισης. κατανόηση των εφαρμογών
1 n-gram n-gram n-gram [11], [15] n-best [16] n-gram. n-gram. 1,a) Graham Neubig 1,b) Sakriani Sakti 1,c) 1,d) 1,e)
![1 n-gram n-gram n-gram [11], [15] n-best [16] n-gram. n-gram. 1,a) Graham Neubig 1,b) Sakriani Sakti 1,c) 1,d) 1,e) 1 n-gram n-gram n-gram [11], [15] n-best [16] n-gram. n-gram. 1,a) Graham Neubig 1,b) Sakriani Sakti 1,c) 1,d) 1,e)](/thumbs/39/20145714.jpg) 1,a) Graham Neubig 1,b) Sakriani Sakti 1,c) 1,d) 1,e) 1. [11], [15] 1 Nara Institute of Science and Technology a) akabe.koichi.zx8@is.naist.jp b) neubig@is.naist.jp c) ssakti@is.naist.jp d) tomoki@is.naist.jp
1,a) Graham Neubig 1,b) Sakriani Sakti 1,c) 1,d) 1,e) 1. [11], [15] 1 Nara Institute of Science and Technology a) akabe.koichi.zx8@is.naist.jp b) neubig@is.naist.jp c) ssakti@is.naist.jp d) tomoki@is.naist.jp
[2] T.S.G. Peiris and R.O. Thattil, An Alternative Model to Estimate Solar Radiation
![[2] T.S.G. Peiris and R.O. Thattil, An Alternative Model to Estimate Solar Radiation [2] T.S.G. Peiris and R.O. Thattil, An Alternative Model to Estimate Solar Radiation](/thumbs/74/71129035.jpg) References [1] B.V.R. Punyawardena and Don Kulasiri, Stochastic Simulation of Solar Radiation from Sunshine Duration in Srilanka [2] T.S.G. Peiris and R.O. Thattil, An Alternative Model to Estimate Solar
References [1] B.V.R. Punyawardena and Don Kulasiri, Stochastic Simulation of Solar Radiation from Sunshine Duration in Srilanka [2] T.S.G. Peiris and R.O. Thattil, An Alternative Model to Estimate Solar
HOSVD. Higher Order Data Classification Method with Autocorrelation Matrix Correcting on HOSVD. Junichi MORIGAKI and Kaoru KATAYAMA
 DEIM Forum 2010 D1-4 HOSVD 191-0065 6-6 E-mail: j.morigaki@gmail.com, katayama@tmu.ac.jp Lathauwer (HOSVD) (Tensor) HOSVD Savas HOSVD Sun HOSVD,, Higher Order Data Classification Method with Autocorrelation
DEIM Forum 2010 D1-4 HOSVD 191-0065 6-6 E-mail: j.morigaki@gmail.com, katayama@tmu.ac.jp Lathauwer (HOSVD) (Tensor) HOSVD Savas HOSVD Sun HOSVD,, Higher Order Data Classification Method with Autocorrelation
1 (forward modeling) 2 (data-driven modeling) e- Quest EnergyPlus DeST 1.1. {X t } ARMA. S.Sp. Pappas [4]
![1 (forward modeling) 2 (data-driven modeling) e- Quest EnergyPlus DeST 1.1. {X t } ARMA. S.Sp. Pappas [4] 1 (forward modeling) 2 (data-driven modeling) e- Quest EnergyPlus DeST 1.1. {X t } ARMA. S.Sp. Pappas [4]](/thumbs/91/106768733.jpg) 212 2 ( 4 252 ) No.2 in 212 (Total No.252 Vol.4) doi 1.3969/j.issn.1673-7237.212.2.16 STANDARD & TESTING 1 2 2 (1. 2184 2. 2184) CensusX12 ARMA ARMA TU111.19 A 1673-7237(212)2-55-5 Time Series Analysis
212 2 ( 4 252 ) No.2 in 212 (Total No.252 Vol.4) doi 1.3969/j.issn.1673-7237.212.2.16 STANDARD & TESTING 1 2 2 (1. 2184 2. 2184) CensusX12 ARMA ARMA TU111.19 A 1673-7237(212)2-55-5 Time Series Analysis
Simplex Crossover for Real-coded Genetic Algolithms
 Technical Papers GA Simplex Crossover for Real-coded Genetic Algolithms 47 Takahide Higuchi Shigeyoshi Tsutsui Masayuki Yamamura Interdisciplinary Graduate school of Science and Engineering, Tokyo Institute
Technical Papers GA Simplex Crossover for Real-coded Genetic Algolithms 47 Takahide Higuchi Shigeyoshi Tsutsui Masayuki Yamamura Interdisciplinary Graduate school of Science and Engineering, Tokyo Institute
Coupling of a Jet-Slot Oscillator With the Flow-Supply Duct: Flow-Acoustic Interaction Modeling
 1th AIAA/CEAS Aeroacoustics Conference, May 006 interactions Coupling of a Jet-Slot Oscillator With the Flow-Supply Duct: Interaction M. Glesser 1, A. Billon 1, V. Valeau, and A. Sakout 1 mglesser@univ-lr.fr
1th AIAA/CEAS Aeroacoustics Conference, May 006 interactions Coupling of a Jet-Slot Oscillator With the Flow-Supply Duct: Interaction M. Glesser 1, A. Billon 1, V. Valeau, and A. Sakout 1 mglesser@univ-lr.fr
Tutorial on Multinomial Logistic Regression
 Tutorial on Multinomial Logistic Regression Javier R Movellan June 19, 2013 1 1 General Model The inputs are n-dimensional vectors the outputs are c-dimensional vectors The training sample consist of m
Tutorial on Multinomial Logistic Regression Javier R Movellan June 19, 2013 1 1 General Model The inputs are n-dimensional vectors the outputs are c-dimensional vectors The training sample consist of m
Nov Journal of Zhengzhou University Engineering Science Vol. 36 No FCM. A doi /j. issn
 2015 11 Nov 2015 36 6 Journal of Zhengzhou University Engineering Science Vol 36 No 6 1671-6833 2015 06-0056 - 05 C 1 1 2 2 1 450001 2 461000 C FCM FCM MIA MDC MDC MIA I FCM c FCM m FCM C TP18 A doi 10
2015 11 Nov 2015 36 6 Journal of Zhengzhou University Engineering Science Vol 36 No 6 1671-6833 2015 06-0056 - 05 C 1 1 2 2 1 450001 2 461000 C FCM FCM MIA MDC MDC MIA I FCM c FCM m FCM C TP18 A doi 10
Βιοπληροφορική. Ενότητα 14: Μοντέλα Πολλαπλής Στοίχισης (2/2), 1.5ΔΩ. Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου
 Βιοπληροφορική Ενότητα 14: Μοντέλα Πολλαπλής Στοίχισης (2/2), 1.5ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι παρουσίαση των μοντέλων πολλαπλής στοίχισης. κατανόηση των εφαρμογών
Βιοπληροφορική Ενότητα 14: Μοντέλα Πολλαπλής Στοίχισης (2/2), 1.5ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι παρουσίαση των μοντέλων πολλαπλής στοίχισης. κατανόηση των εφαρμογών
Βιοπληροφορική. Ενότητα 7: Στοίχιση ακολουθιών ανά ζεύγη Τεχνικές Στοίχισης Ακολουθιών,(2/2) 2 ΔΩ. Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ.
 Βιοπληροφορική Ενότητα 7: Στοίχιση ακολουθιών ανά ζεύγη Τεχνικές Στοίχισης Ακολουθιών,(2/2) 2 ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι Παρουσίαση της μεθόδου κατασκευής και
Βιοπληροφορική Ενότητα 7: Στοίχιση ακολουθιών ανά ζεύγη Τεχνικές Στοίχισης Ακολουθιών,(2/2) 2 ΔΩ Τμήμα: Βιοτεχνολογίας Όνομα καθηγητή: Τ. Θηραίου Μαθησιακοί Στόχοι Παρουσίαση της μεθόδου κατασκευής και
Matrices and vectors. Matrix and vector. a 11 a 12 a 1n a 21 a 22 a 2n A = b 1 b 2. b m. R m n, b = = ( a ij. a m1 a m2 a mn. def
 Matrices and vectors Matrix and vector a 11 a 12 a 1n a 21 a 22 a 2n A = a m1 a m2 a mn def = ( a ij ) R m n, b = b 1 b 2 b m Rm Matrix and vectors in linear equations: example E 1 : x 1 + x 2 + 3x 4 =
Matrices and vectors Matrix and vector a 11 a 12 a 1n a 21 a 22 a 2n A = a m1 a m2 a mn def = ( a ij ) R m n, b = b 1 b 2 b m Rm Matrix and vectors in linear equations: example E 1 : x 1 + x 2 + 3x 4 =
Biostatistics for Health Sciences Review Sheet
 Biostatistics for Health Sciences Review Sheet http://mathvault.ca June 1, 2017 Contents 1 Descriptive Statistics 2 1.1 Variables.............................................. 2 1.1.1 Qualitative........................................
Biostatistics for Health Sciences Review Sheet http://mathvault.ca June 1, 2017 Contents 1 Descriptive Statistics 2 1.1 Variables.............................................. 2 1.1.1 Qualitative........................................
(1) (2) MFA/SFA: material flow analysis/ substance flow analysis MFA/SFA
 (1) (2) MFA/SFA: material flow analysis/ substance flow analysis MFA/SFA 18 1) I. Daigo, Y. Matsuno, Y. Adachi: Substance Flow Analysis of Chromium and Nickel in the Material Flow of Stainless Steel in
(1) (2) MFA/SFA: material flow analysis/ substance flow analysis MFA/SFA 18 1) I. Daigo, Y. Matsuno, Y. Adachi: Substance Flow Analysis of Chromium and Nickel in the Material Flow of Stainless Steel in
Applying Markov Decision Processes to Role-playing Game
 1,a) 1 1 1 1 2011 8 25, 2012 3 2 MDPRPG RPG MDP RPG MDP RPG MDP RPG MDP RPG Applying Markov Decision Processes to Role-playing Game Yasunari Maeda 1,a) Fumitaro Goto 1 Hiroshi Masui 1 Fumito Masui 1 Masakiyo
1,a) 1 1 1 1 2011 8 25, 2012 3 2 MDPRPG RPG MDP RPG MDP RPG MDP RPG MDP RPG Applying Markov Decision Processes to Role-playing Game Yasunari Maeda 1,a) Fumitaro Goto 1 Hiroshi Masui 1 Fumito Masui 1 Masakiyo
Toward a SPARQL Query Execution Mechanism using Dynamic Mapping Adaptation -A Preliminary Report- Takuya Adachi 1 Naoki Fukuta 2.
 SIG-SWO-041-05 SPAIDA: SPARQL Toward a SPARQL Query Execution Mechanism using Dynamic Mapping Adaptation -A Preliminary Report- 1 2 Takuya Adachi 1 Naoki Fukuta 2 1 1 Faculty of Informatics, Shizuoka University
SIG-SWO-041-05 SPAIDA: SPARQL Toward a SPARQL Query Execution Mechanism using Dynamic Mapping Adaptation -A Preliminary Report- 1 2 Takuya Adachi 1 Naoki Fukuta 2 1 1 Faculty of Informatics, Shizuoka University
 DEIM Forum 2012 D2-1 606 8501 150 0002 2-15-1 28F E-mail: {tsukuda,ohshima,tanaka}@dl.kuis.kyoto-u.ac.jp, {miyamamoto,hiwasaki}@d-itlab.co.jp 1 Wikipedia Wikipedia HITS 1. Web Web Web 1 3 Wikipedia 2 Web
DEIM Forum 2012 D2-1 606 8501 150 0002 2-15-1 28F E-mail: {tsukuda,ohshima,tanaka}@dl.kuis.kyoto-u.ac.jp, {miyamamoto,hiwasaki}@d-itlab.co.jp 1 Wikipedia Wikipedia HITS 1. Web Web Web 1 3 Wikipedia 2 Web
Math 6 SL Probability Distributions Practice Test Mark Scheme
 Math 6 SL Probability Distributions Practice Test Mark Scheme. (a) Note: Award A for vertical line to right of mean, A for shading to right of their vertical line. AA N (b) evidence of recognizing symmetry
Math 6 SL Probability Distributions Practice Test Mark Scheme. (a) Note: Award A for vertical line to right of mean, A for shading to right of their vertical line. AA N (b) evidence of recognizing symmetry
A method for identifying TSS from CAGE data using a Genomic Signal Processing approach
 NATIONAL AND KAPODISTRIAN UNIVERSITY OF ATHENS SCHOOL OF SCIENCE DEPARTMENT OF INFORMATICS & TELECOMMUNICATIONS POSTGRADUATE PROGRAM "INFORMATION TECHNOLOGIES IN MEDICINE AND BIOLOGY" MASTER THESIS A method
NATIONAL AND KAPODISTRIAN UNIVERSITY OF ATHENS SCHOOL OF SCIENCE DEPARTMENT OF INFORMATICS & TELECOMMUNICATIONS POSTGRADUATE PROGRAM "INFORMATION TECHNOLOGIES IN MEDICINE AND BIOLOGY" MASTER THESIS A method
Chinese Journal of Applied Probability and Statistics Vol.28 No.3 Jun (,, ) 应用概率统计 版权所用,,, EM,,. :,,, P-. : O (count data)
 2012 6 Chinese Journal of Applied Probability and Statistics Vol.28 No.3 Jun. 2012 (,, 675000),,, EM,,. :,,, P-. : O212.7. 1. (count data), Poisson Poisson,, (zero-inflation).,.,, ;,,.,, Fahrmeir Echavarrri
2012 6 Chinese Journal of Applied Probability and Statistics Vol.28 No.3 Jun. 2012 (,, 675000),,, EM,,. :,,, P-. : O212.7. 1. (count data), Poisson Poisson,, (zero-inflation).,.,, ;,,.,, Fahrmeir Echavarrri
Εφαρμοσμένη Βιοτεχνολογία Σημειώσεις. Νίκος Τσουκιάς Σχολή Χημικών Μηχανικών ΕΜΠ
 Εφαρμοσμένη Βιοτεχνολογία Σημειώσεις Νίκος Τσουκιάς Σχολή Χημικών Μηχανικών ΕΜΠ Γενωμική και Συστημική Βιολογία [Ch 8.5] Genomics is a discipline in genetics that applies recombinant DNA, DNA sequencing
Εφαρμοσμένη Βιοτεχνολογία Σημειώσεις Νίκος Τσουκιάς Σχολή Χημικών Μηχανικών ΕΜΠ Γενωμική και Συστημική Βιολογία [Ch 8.5] Genomics is a discipline in genetics that applies recombinant DNA, DNA sequencing
: Active Learning 2017/11/12
 : Active Learning 2017/11/12 Contents 0.1 Introduction............................................ 2 0.2..................................... 2 0.2.1 Membership Query Synthesis..............................
: Active Learning 2017/11/12 Contents 0.1 Introduction............................................ 2 0.2..................................... 2 0.2.1 Membership Query Synthesis..............................
