Figure S1. Localization of GiOR-1 with truncated N-terminal domain of 29 amino acid residues (ΔL-GiOR-1-HA).
|
|
|
- Γερασιμος Βαρουξής
- 7 χρόνια πριν
- Προβολές:
Transcript
1 Figure S1. Localization of GiOR-1 with truncated N-terminal domain of 29 amino acid residues (ΔL-GiOR-1-HA). A L-GiOR-1-HA Tom40 DAPI merge DIC B LYS CYT ORG ORG Trypsin L-GiOR-1-HA
2 Figure S2A. Phylogeny of hydrogenases, Hyd-CPR fusion proteins and Nar1 Mastigamoeba balamuthi H Blastocystis hominis H 100/1 Trichomonas vaginalis F Trichomonas vaginalis F 68 /1 Carpediemonas membranifera F Chilomastix cuspidata F Dysnectes brevis F Trimastix PCT F Hydrogenases and 75 Trichomonas vaginalis H Hyd-CPR fusion proteins /1 100 /1 Trichomonas vaginalis H Trichomonas vaginalis H HYD FMN FAD 66 /1 Chlamydomonas reinhardtii H Naegleria gruberi H HYD 88/ 73/ Entamoeba histolytica H Giardia intestinalis H Spironucleus salmonicida H Spironucleus salmonicida H Encephalitozoon cuniculi 100/1 Chlamydomonas reinhardtii Arabidopsis thaliana 50/ 0.94 Mastigamoeba balamuthi 62 /0.58 Dictyostelium discoideum Tetrahymena thermophila Homo sapiens Saccharomyces cerevisiae Monosiga brevicollis Naegleria gruberi Cryptosporidium parvum Nar1 Blastocystis hominis Entamoeba histolytica 58/ 0.64 Carpediemonas membranifera Chilomastix cuspidata Dysnectes brevis 99/1 Giardia intestinalis 92/1 87/1 Spironucleus vortens Trepomonas Sp Spironucleus salmonicida 98/1 Bodo saltans 97/1 Leishmania major Trypanosoma brucei Trichomonas vaginalis 0.6 HYD
3 Figure S2B. Comparison of HC1 domains among hydrogenases (Hyd), hydrogenases that are fused with CPR (Hyd-CPR), and Nar1 components of CIA pathway. HC1 Naegleria gruberi Trichomonas vaginalis Giardia intestinalis Spironucleus salmonicida Blastocystis hominis Entamoeba histolytica Trimastix marina PCT Trichomonas vaginalis Trichomonas vaginalis Carpediemonas membranifera Chilomastix cuspidata Naegleria gruberi Bodo saltans Trichomonas vaginalis Carpediemonas membranifera Chilomastix cuspidata Dysnectes brevis Giardia intestinalis Spironucleus salmonicida Trepomonas PC1 Blastocystis hominis Entamoeba histolytica Saccharomyces cerevisiae MYTSCCPGWVNLVESDFPE - I I PNVSSCKSPQQMLGS- VI RN MFTSCCPAWVNYVEKSDPS -LI PHLSSCRSPMSMLSS- VI KN VFTSCCPAWVNLCEMKYPE - LLPQVSTAKSCVAMVATL- VKR VFTSCCPAWVNLCERKYPQ- LLGRLSTAKSPHGSI CT- LI KR MFTSCCPGWVNMAEKCYPE- LI PNLSSCRSPHMMLGSC- VKT AI TSCCPGWLEYLEKKYPD- LI PRVSTARSPI ACLAGC- I KN MFTSCCPGWI NLLEQKYPE - FI PNVSSCKSPQQMLGA- VI KT - FTSWCPAWVNFVEKLHPE -LIPNL STVKSPHI L LSV- I I RN MI TSS CPSWVNFI EKI FPD- LTENL STTMSPHI I TGS- LI KN MFTSCCPAWI NNI ETRFPD- YLDNLSTARSPMMMMGS- VVKT MFTSCCPGWI NFVEQEAPD- FI PNLSSCKSPMGMQSAL- LRT VFASACPGWI CYAEKTQPE - I I PSI STVKSPQQI MGT- FVKK MI VSS CPGWVCYCEKQYPE- LLPHLSRVMSPQGI AGS- LLKR - MI SRCAGSVVYYERKTS- - YADHLAQI KPYPQLYAMYEKKI LVVSS CPGFVCVCEKQNPE - VLPKMSRVGSPMAYTAA LFVSACPGWVCLAQKRYPD- TVARLARAQSSMTVFGTALLAR RI VTVCPAVRLYI GKRGPS -LIPHLDNTPSPTEI AGHGL AI I THCPAVRLFI TKRNRE- LI SYI VSTASPMELFGASYCGI --I THCPAVRLYI LKKKPE -LYKYVNTVGSMLEFQ LI VSHCPAAKLFLNKKDS S -LSKFFSKSASPLEI FFS FI TSNCPGWVCYAEKKTHA- LVPFLSTTKSAQQI MGS- LLRK - LTST CPGWVCYSEKMQGKWMFEYMSKVASSMTI AGMI - MKK LLSAVCPGFLI YTEKTKPQ- LVPMLLNVKSPQQI TGS- LI RA Hyd Hyd-CPR Nar1
4 Figure S3. Distribution of Tah18, Dre2, RNR1 and RNR2 proteins in Metamonada and various anaerobes. Tah18 Dre2 RNR1 RNR2 N. gruberi T. brucei B. saltans T.marina PCT T. vaginalis C. membranifera E. cyprinoides CLO NY171 C. cuspidata Excavata Anaerobes G. intestinalis S. salmonicida S. vortens Trepomonas PC1 B. hominis E. histolytica M. balamuthi D. dictyostelium D. brevis Amoebas
5 Figure Legends Figure S1. Localization of GiOR-1 with truncated N-terminal domain of 29 amino acid residues (ΔL-GiOR-1-HA). (A) Immunofluorescence microscopy revealed that ΔL-GiOR-1- HA localizes to mitosomes. ΔL-GiOR-1-HA was detected using anti hemagglutinin (HA) antibody (green). Tom40 was detected as mitosomal marker protein using polyclonal rabbit anti-tom40 antibody (red). Nuclei were visualized by DAPI (4,6-Diamidine-2 -phenylindole dihydrochloride). DIC, differential interference contrast. (B) Alternatively, the cellular localization of ΔL-GiOR-1-HA was investigated using western blot analysis of cellular fractions (LYS, cell lysate; CYT, the cytosol; ORG, the organellar fraction) and protein protection assay (treatment of the organellar fraction with 200 μg/ml of trypsin for 10 min at 37 C). Figure S2. Phylogeny of hydrogenase like domain containing proteins A. Maximum likelihood-based phylogeny of Nar1 and hydrogenase domain-containing proteins in eukaryotes. Hydrogenases cluster together with the CPR-hydrogenase fusion from metamonads. Numbers indicate statistical support in the form of bootstrap values or PP values. The scale bar represents the estimated number of amino acid substitutions per site. Only bootstrap support values greater than 50 and PP values greater than 0.5 are shown. Green indicates Viridiplantae, red Opisthokonta, yellow the SAR supergroup, brown Amoebozoa, and blue Excavata. H, only hydrogenase domain-containing protein; F, CPRhydrogenase fusion protein. B. Alignment of HC1 site of CPR-hydrogenase fusion proteins in metamonads with representative protein sequences for hydrogenase and nar1 protein. The HC1 motif is highlighted.
6 Figure S3. Distribution of Tah18, Dre2, RNR1 and RNR2 proteins in Metamonada and various anaerobes. A function in the synthesis of diferric-tyrosyl radical (Fe(III)2-Y ) cofactor of ribonucleotide reductase (RNR) was recently suggested for Tah18 and Dre2 (17). Hence, we searched to determine whether genes for RNR proteins are missing in organisms lacking Tah18-Dre2 proteins. Nevertheless, we failed to find any such correlation to support this hypothesis. RNR1, Ribonucleotide reductase 1; RNR2, Ribonucleotide reductase 2. Black shading, an ortholog is present; White, an ortholog is absent from the genomic database; Grey, an ortholog is absent from a given EST database.
7 Table S1. Analysis of immunoprecipitated proteins by mass spectrometry Identified proteins Acc. Number Molecular weight Protein identification probability Total unique spectra count GiOR1 Cia2 CBirA CBirA CBirA CBirA Wt Wt GiOR1 Cia2 CBirA CBirA CBirA CBirA Wt Wt Nitric oxide synthase, inducible location=glchr04: ( ) length=604 GL50803_ kda 100% 7 Chaperone protein DnaK HSP70 location=glchr03: ( ) length=640 GL50803_ kda 100% 9 Chaperonin 60 location=glchr02: ( ) length=547 GL50803_ kda 100% 8 Cysteine desulfurase location=glchr03: (+) length=433 GL50803_ kda 100% 4 Alpha mitochondrial protein peptidase like protein location=glchr01: ( GL50803_ kda 100% 3 Glutaredoxin related protein location=glchr01: (+) length=202 GL50803_ kda 100% 3 Hypothetical protein location=glchr05: ( ) length=286 GL50803_ kda 100% 2 Hypothetical protein location=glchr01: (+) length=278 GL50803_ kda 100% 2 Hypothetical protein location=glchr04: ( ) length=197 GL50803_ kda 100% 2 [2Fe 2S] ferredoxin location=glchr02: ( ) length=133 GL50803_ kda 100% 2 Hypothetical protein location=glchr03: ( ) length=125 GL50803_ kda 100% 2 Hypothetical protein location=glchr01: (+) length=361 GL50803_ kda 100% 96% 2 1 HesB domain containing protein location=glchr01: ( ) length=131 GL50803_ kda 100% 1 Beta adaptin location=glchr05: ( ) length=1132 GL50803_ kda 100% 1 Hypothetical protein location=ch991767: (+) length=1095 GL50803_ kda 100% 1 Gamma giardin location=glchr03: ( ) length=311 GL50803_ kda 100% 1 Axonemal p66.0 location=glchr03: (+) length=552 GL50803_ kda 100% 1 Hypothetical protein location=glchr01: (+) length=745 GL50803_ kda 100% 1 Hypothetical protein location=glchr01: (+) length=745 GL50803_ kda 100% 1 Hypothetical protein location=glchr02: (+) length=464 GL50803_ kda 100% 1 Kinase, NEK location=glchr04: ( ) length=898 GL50803_ kda 100% 1 Hypothetical protein location=glchr05: ( ) length=264 GL50803_ kda 99% 1 Hypothetical protein location=glchr03: ( ) length=669 GL50803_ kda 99% 1 Hypothetical protein location=glchr02: (+) length=228 GL50803_ kda 99% 1 Hypothetical protein location=glchr04: (+) length=472 GL50803_ kda 98% 1 Hypothetical protein location=glchr05: (+) length=805 GL50803_ kda 95% 1 Protein required for cell viability location=glchr05: (+) length=145 GL50803_ kda 100% 7 Coiled coil protein location=glchr04: ( ) length=1675 GL50803_ kda 100% 7 ELKS location=glchr01: (+) length=924 GL50803_ kda 100% 100% 1 4 Nuclear ATP/GTP binding protein location=glchr02: ( ) length=1422 GL50803_ kda 100% 3 MDR type permease location=glchr05: ( ) length=666 GL50803_ kda 100% 3 Hypothetical protein location=glchr05: (+) length=1471 GL50803_ kda 100% 100% 1 2 Hypothetical protein location=glchr03: (+) length=702 GL50803_ kda 100% 2 Hypothetical protein location=glchr02: ( ) length=252 GL50803_ kda 100% 2 Hypothetical protein location=glchr05: ( ) length=772 GL50803_ kda 100% 2 Hypothetical protein location=glchr02: (+) length=2292 GL50803_ kda 100% 1 hypothetical protein location=glchr05: ( ) length=255 GL50803_ kda 96% 1 Kinase, NEK location=glchr04: ( ) length=823 GL50803_ kda 100% 1 Potassium transporting ATPase alpha chain 1 location=glchr02: (+) GL50803_ kda 100% 1 Rac/Rho like protein location=glchr03: (+) length=218 GL50803_ kda 96% 1 Hypothetical protein location=glchr01: (+) length=199 GL50803_ kda 96% 1 Hypothetical protein location=glchr04: ( ) length=1261 GL50803_ kda 96% 1 Protein 21.1 location=glchr05: ( ) length=330 GL50803_ kda 100% 95% 2 1 Coiled coil protein location=glchr02: (+) length=2275 GL50803_ kda 100% 99% 1 1 Kinase, NEK location=glchr03: ( ) length=287 GL50803_ kda 96% 1 DNA directed RNA polymerase RPB3 location=glchr05: (+) GL50803_ kda 96% 1 Manganese dependent inorganic pyrophosphatase, putative GL50803_ kda 98% 1 Vacuolar ATP synthase subunit H location=glchr01: (+) length=530 GL50803_ kda 100% 1 Centromere/microtubule binding protein CBF5 location=glchr05: (+) GL50803_ kda 99% 1 H SHIPPO 1 location=glchr05: ( ) length=615 GL50803_ kda 99% 1 Alpha 11 giardin location=glchr05: ( ) length=307 GL50803_ kda 96% 1
8 Sec23 location=glchr02: (+) length=861 GL50803_ kda 96% 1 Kinase, CMGC CDK location=glchr02: (+) length=340 GL50803_ kda 96% 1 Hypothetical protein location=glchr05: ( ) length=974 GL50803_ kda 95% 1 Nucleolar GTP binding protein 1, putative location=glchr04: ( ) GL50803_ kda 99% 1 Phosphatase location=glchr01: ( ) length=310 GL50803_ kda 100% 1 Kinase, CAMK CAMKL location=glchr03: ( ) length=432 GL50803_ kda 95% 96% 1 1 UTP glucose 1 phosphate uridylyltransferase location=glchr04: (+) GL50803_ kda 100% 1 MCT 1 protein like protein location=glchr05: (+) length=237 GL50803_ kda 99% 1 Kinase, NEK location=glchr05: ( ) length=349 GL50803_ kda 96% 1 Nucleoside diphosphate kinase location=glchr03: ( ) length=387 GL50803_ kda 96% 1 Hypothetical protein location=glchr05: ( ) length=198 GL50803_ kda 96% 1 Phosphatase location=glchr02: ( ) length=385 GL50803_ kda 96% 1 Transglutaminase/protease, putative location=glchr05: ( ) GL50803_ kda 96% 1 Phosphatidate cytidylyltransferase location=glchr03: ( ) length=376 GL50803_ kda 95% 1 Phosphorylated CTD interacting factor PCIF1 location=glchr04: ( ) GL50803_ kda 96% 1 IFT complex B location=glchr03: ( ) length=672 GL50803_ kda 96% 1 Hypothetical protein location=glchr04: ( ) length=605 GL50803_ kda 99% 1 Clathrin heavy chain location=glchr04: (+) length=1871 GL50803_ kda 100% 1 Hypothetical protein location=glchr03: ( ) length=848 GL50803_ kda 96% 1 Spindle pole protein, putative location=glchr05: ( ) length=554 GL50803_ kda 96% 1 L asparaginase location=glchr04: (+) length=400 GL50803_ kda 96% 1 Kinase, NEK location=glchr04: (+) length=386 GL50803_ kda 96% 1 Translation initiation factor 2 gamma subunit location=glchr04: (+) GL50803_ kda 96% 1 Translation initiation factor eif 4A, putative location=glchr01: ( ) GL50803_ kda 96% 95% 1 1 Protein location=glchr05: (+) length=748 GL50803_ kda 99% 100% 100% Ser/Thr phosphatase 2C, putative location=glchr02: ( ) GL50803_ kda 96% 1 La ribonucleoprotein, putative location=glchr05: ( ) length=348 GL50803_ kda 100% 1 Protein 21.1 location=glchr03: (+) length=1457 GL50803_ kda 100% 100% 7 1 Trichohyalin location=glchr05: ( ) length=1414 GL50803_ kda 100% 1 Kinase, NEK location=glchr03: (+) length=650 GL50803_ kda 100% 100% 1 1 Protein 21.1 location=glchr04: ( ) length=1500 GL50803_ kda 100% 99% 6 1 Hypothetical protein location=glchr05: ( ) length=342 GL50803_ kda 99% 100% 95% Alcohol dehydrogenase lateral transfer candidate location=glchr04: GL50803_ kda 96% 1 Beta giardin location=glchr04: (+) length=272 GL50803_ kda 100% 95% 2 1 Hypothetical protein location=glchr02: (+) length=254 GL50803_ kda 100% 97% S proteasome regulatory subunit, putative location=glchr01: (+) GL50803_ kda 96% 1 Type II inositol 1,4,5 trisphosphate 5 phosphatase precursor GL50803_ kda 100% 1 Hypothetical protein location=glchr02: ( ) length=493 GL50803_ kda 96% 1 Uridine kinase location=glchr05: ( ) length=682 GL50803_ kda 96% 1 Hypothetical protein location=glchr04: (+) length=480 GL50803_ kda 96% 1 hypothetical protein location=glchr04: (+) length=853 GL50803_ kda 96% 1 Hypothetical protein location=glchr04: ( ) length=308 GL50803_ kda 95% 1 Uridine kinase location=glchr03: (+) length=582 GL50803_ kda 96% 1 Kinase, NEK location=glchr05: ( ) length=493 GL50803_ kda 100% 2 Long chain fatty acid CoA ligase 5 location=glchr05: (+) GL50803_ kda 100% 2 Histone H2B location=glchr05: (+) length=130 GL50803_ kda 95% 1 Kinase, CMGC DYRK location=glchr01: ( ) length=860 GL50803_ kda 96% 1 DNA topoisomerase III location=glchr04: (+) length=973 GL50803_ kda 96% 1 Phosphoenolpyruvate carboxykinase location=glchr03: ( ) GL50803_ kda 100% 3 Hypothetical protein location=glchr04: (+) length=125 GL50803_ kda 95% 1 Peptide methionine sulfoxide reductase msrb location=glchr05: (+) GL50803_ kda 95% 1 Dynein intermediate chain location=glchr02: (+) length=757 GL50803_ kda 100% 1 CAMP dependent protein kinase regulatory chain location=glchr05: GL50803_ kda 100% 3 Ribosome biogenesis protein BMS1 location=glchr03: (+) GL50803_ kda 100% 1 hypothetical protein location=glchr04: ( ) length=872 GL50803_ kda 100% 100% 99% 95%
9 Histidyl trna synthetase location=glchr01: ( ) length=961 GL50803_ kda 96% 1 70 kda peptidylprolyl isomerase, putative location=glchr04: ( ) GL50803_ kda 100% 2 Nitroreductase Fd NR2 location=glchr01: (+) length=264 GL50803_ kda 96% 1 Hypothetical protein location=glchr02: ( ) length=319 GL50803_ kda 99% 1 Aminoacyl histidine dipeptidase location=glchr05: (+) length=506 GL50803_ kda 100% 1 Kinase, STE STE20 location=ch991761: (+) length=423 GL50803_ kda 96% 1 Farnesyl diphosphate synthase location=glchr04: ( ) length=407 GL50803_ kda 96% 1 Protein 21.1 location=glchr05: ( ) length=681 GL50803_ kda 100% 2 A type flavoprotein lateral transfer candidate location=glchr04: ( ) GL50803_ kda 100% 2 Protein 21.1 location=glchr03: ( ) length=760 GL50803_ kda 100% 100% 100% Protein phosphatase 2A regulatory subunit, putative location=glchr04: GL50803_ kda 100% 2 Rab2a location=glchr04: ( ) length=214 GL50803_ kda 100% 100% 1 1 Hypothetical protein location=glchr02: ( ) length=431 GL50803_ kda 100% 99% 1 1 Kinesin 13 location=glchr03: ( ) length=714 GL50803_ kda 100% 1 Hypothetical protein location=glchr05: (+) length=588 GL50803_ kda 100% 100% 1 1 Kinase, NEK frag location=glchr02: (+) length=706 GL50803_ kda 100% 2 hypothetical protein location=glchr05: ( ) length=1529 GL50803_ kda 100% 1 Kinase, CAMK CAMKL location=glchr05: (+) length=403 GL50803_ kda 96% 1 Phosphorylase B kinase gamma catalytic chain location=glchr04: ( ) GL50803_ kda 96% 1 Hypothetical protein location=glchr04: (+) length=875 GL50803_ kda 95% 1 Protein 21.1 location=glchr04: ( ) length=785 GL50803_ kda 95% 1 Ribosomal protein S29A location=glchr05: (+) length=137 GL50803_ kda 100% 1 TCP 1 chaperonin subunit alpha location=glchr04: (+) length=550 GL50803_ kda 99% 1 Coiled coil protein location=ch991767: (+) length=1685 GL50803_ kda 100% 2 TCP 1 chaperonin subunit beta location=glchr02: ( ) length=525 GL50803_ kda 100% 2 Hypothetical protein location=glchr01: (+) length=528 GL50803_ kda 100% 1 Protein F10G7.1 location=glchr04: (+) length=826 GL50803_ kda 100% 2 Dual specificity phosphatase Cdc25, putative location=glchr03: (+) GL50803_ kda 95% 100% 1 1 Dipeptidyl peptidase III location=glchr01: (+) length=694 GL50803_ kda 100% 1 Hypothetical protein location=ch991767: ( ) length=1262 GL50803_ kda 96% 1 Prefoldin subunit 3, putative location=glchr01: (+) length=192 GL50803_ kda 100% 1 Nucleolar protein NOP5 location=glchr05: (+) length=613 GL50803_ kda 100% 100% 100% Ubiquitin conjugating enzyme E1 location=glchr05: ( ) GL50803_ kda 100% 2 DEAD box RNA helicase Vasa location=glchr05: (+) length=449 GL50803_ kda 100% 1 TCP 1 chaperonin subunit theta location=glchr02: ( ) length=563 GL50803_ kda 100% 1 Alcohol dehydrogenase lateral transfer candidate location=glchr03: GL50803_ kda 100% 1 Ribosomal protein L7 location=glchr04: (+) length=235 GL50803_ kda 100% 100% 1 1 Kinase, NEK location=glchr05: ( ) length=288 GL50803_ kda 100% 3 Hypothetical protein location=glchr05: ( ) length=262 GL50803_ kda 100% 95% 3 1 Proteasome subunit beta type 1 location=glchr05: ( ) length=254 GL50803_ kda 100% 2 Nucleolar protein NOP5 location=glchr01: ( ) length=498 GL50803_ kda 100% 2 Coiled coil protein location=glchr05: ( ) length=1652 GL50803_ kda 100% 1 Metalloprotease, insulinase family location=glchr02: (+) GL50803_ kda 100% 2 Hypothetical protein location=glchr02: ( ) length=237 GL50803_ kda 100% 1 Translation elongation factor location=glchr01: ( ) length=220 GL50803_ kda 100% 2 Hypothetical protein location=glchr05: ( ) length=250 GL50803_ kda 100% 2 Hypothetical protein location=glchr03: (+) length=213 GL50803_ kda 100% 1 hypothetical protein location=glchr03: ( ) length=4360 GL50803_ kda 96% 1 20S proteasome alpha subunit 5 location=glchr02: ( ) length=245 GL50803_ kda 100% 2 Serine/threonine protein phosphatase PP X isozyme 2 location=glchr02: GL50803_ kda 100% 1 Ubiquitin carboxyl terminal hydrolase 4 location=glchr05: ( ) GL50803_ kda 100% 2 Hypothetical protein location=glchr03: ( ) length=204 GL50803_ kda 100% 1 Kinase, NEK location=glchr04: ( ) length=584 GL50803_ kda 100% 2 Nucleolar GTP binding protein 2 location=glchr05: ( ) length=737 GL50803_ kda 100% 2 Hypothetical protein location=glchr03: (+) length=330 GL50803_ kda 100% 100% 1 1
10 Glutaminyl trna synthetase location=glchr05: ( ) length=702 GL50803_ kda 100% 1 Rab11 location=glchr05: ( ) length=216 GL50803_ kda 96% 100% 1 3 Alanine aminotransferase, putative location=glchr03: (+) length=521 GL50803_ kda 100% 3 Hypothetical protein location=glchr04: ( ) length=845 GL50803_ kda 100% 2 Prolyl trna synthetase location=glchr04: (+) length=542 GL50803_ kda 100% 1 Hypothetical protein location=glchr05: (+) length=959 GL50803_ kda 99% 100% 1 1 TBP interacting protein TIP49 location=glchr02: (+) length=468 GL50803_ kda 100% 3 Hypothetical protein location=glchr05: (+) length=1236 GL50803_ kda 100% 100% 99% 2 2 Aldose reductase location=glchr02: (+) length=313 GL50803_ kda 100% 2 Hypothetical protein location=glchr02: (+) length=271 GL50803_ kda 100% 2 Serine/threonine protein phosphatase 7 location=glchr05: (+) GL50803_ kda 100% 2 Hypothetical protein location=glchr05: (+) length=276 GL50803_ kda 100% 100% 100% hypothetical protein location=glchr02: ( ) length=423 GL50803_ kda 100% 2 Kinase, NEK location=glchr02: (+) length=285 GL50803_ kda 100% 2 Hypothetical protein location=glchr05: (+) length=888 GL50803_ kda 100% 100% 100% Dual specificity protein phosphatase CDC14A location=glchr04: (+) GL50803_ kda 100% 99% 1 1 Hypothetical protein location=glchr04: (+) length=460 GL50803_ kda 96% 100% 1 2 Hypothetical protein location=glchr05: ( ) length=123 GL50803_ kda 100% 1 Hypothetical protein location=glchr02: (+) length=260 GL50803_ kda 100% 3 Fibrillarin like pre rrna processing protein Narcisi et al location=glchr05: GL50803_ kda 100% 3 hypothetical protein location=glchr04: (+) length=1296 GL50803_ kda 100% 3 Kinase, CMGC MAPK location=glchr05: ( ) length=360 GL50803_ kda 100% 3 TCP 1 chaperonin subunit eta location=glchr05: ( ) length=595 GL50803_ kda 100% 1 S adenosylmethionine synthetase location=glchr01: (+) GL50803_ kda 100% 2 Protein location=glchr05: ( ) length=1044 GL50803_ kda 100% 100% 4 1 Kinase, NEK location=glchr03: (+) length=376 GL50803_ kda 100% 100% 1 1 Kinase, NEK location=glchr04: (+) length=405 GL50803_ kda 100% 1 Threonyl trna synthetase location=glchr05: (+) length=695 GL50803_ kda 100% 3 Kinesin 14 location=glchr01: ( ) length=1172 GL50803_ kda 100% 5 hypothetical protein location=glchr04: ( ) length=1003 GL50803_ kda 100% 100% 100% Bip location=glchr04: (+) length=677 GL50803_ kda 100% 100% 100% Hypothetical protein location=glchr05: (+) length=1904 GL50803_ kda 100% 3 Ribosomal protein S7 location=glchr05: ( ) length=190 GL50803_ kda 100% 2 Coiled coil protein location=ch991776: (+) length=1086 GL50803_ kda 100% 5 Plasma membrane calcium transporting ATPase 2 location=glchr05: GL50803_ kda 100% 100% 3 5 NADH oxidase lateral transfer candidate location=glchr01: ( ) GL50803_ kda 100% 100% 1 3 Hypothetical protein location=glchr04: (+) length=567 GL50803_ kda 100% 4 Phosphomannomutase 2 location=glchr04: ( ) length=655 GL50803_ kda 100% 3 FKBP type peptidyl prolyl cis trans isomerase location=glchr01: ( ) GL50803_ kda 100% 3 Alcohol dehydrogenase 3 lateral transfer candidate location=glchr03: GL50803_ kda 100% 1 Hexose transporter location=glchr03: ( ) length=512 GL50803_ kda 95% 95% 1 1 Protein 21.1 location=glchr04: (+) length=723 GL50803_ kda 95% 1 Hypothetical protein location=glchr01: ( ) length=257 GL50803_ kda 95% 96% 96% 97% Glucokinase location=glchr05: (+) length=343 GL50803_ kda 100% 2 U3 small nucleolar RNA associated protein 11, putative location=glchr04: GL50803_ kda 96% 1 Kinase, NEK location=glchr03: ( ) length=733 GL50803_ kda 96% 1 Cysteinyl trna synthetase location=glchr04: ( ) length=645 GL50803_ kda 96% 1 Vacuolar ATP synthase subunit C location=glchr02: (+) length=494 GL50803_ kda 96% 1 Phosphoglycolate phosphatase location=glchr05: (+) length=228 GL50803_ kda 95% 1 Ubiquitin conjugating enzyme E2 17 kda location=glchr02: (+) GL50803_ kda 95% 1 Hypothetical protein location=glchr04: ( ) length=141 GL50803_ kda 95% 95% 1 1 FKBP type peptidyl prolyl cis trans isomerase location=glchr01: ( ) GL50803_ kda 100% 1 Hypothetical protein location=glchr02: (+) length=81 GL50803_ kda 95% 1 GTP binding protein location=glchr05: ( ) length=507 GL50803_ kda 100% 1 Hypothetical protein location=glchr05: (+) length=445 GL50803_ kda 99% 1
11 Spindle pole protein, putative location=glchr03: ( ) length=693 GL50803_ kda 99% 1 Hypothetical protein location=glchr02: (+) length=153 GL50803_ kda 100% 1 Arsenical pump driving ATPase location=glchr03: ( ) length=354 GL50803_ kda 96% 1 Hypothetical protein location=glchr05: (+) length=284 GL50803_ kda 98% 96% 95% 100% Hypothetical protein location=glchr01: ( ) length=140 GL50803_ kda 100% 1 Alanyl trna synthetase location=glchr04: ( ) length=969 GL50803_ kda 100% 1 Protein 21.1 location=glchr05: (+) length=925 GL50803_ kda 99% 1 L serine dehydratase location=glchr03: (+) length=516 GL50803_ kda 96% 1 TBP interacting protein TIP49 location=glchr04: ( ) length=483 GL50803_ kda 98% 1 Protein 21.1 location=glchr05: ( ) length=1873 GL50803_ kda 100% 1 LEK1 location=glchr05: (+) length=716 GL50803_ kda 100% 1 Hypothetical protein location=glchr05: ( ) length=483 GL50803_ kda 96% 1 Vacuolar protein sorting 26, putative location=glchr05: (+) GL50803_ kda 100% 1 Ubiquitin fusion degradation protein 1 location=glchr01: ( ) GL50803_ kda 96% 1 Spindle pole protein, putative location=glchr05: ( ) length=1032 GL50803_ kda 100% 1 organism=crap product=crap_aldoa_rabit GL50803_ kda 95% 1 Hypothetical protein location=glchr01: ( ) length=695 GL50803_ kda 100% 96% 100% Hypothetical protein location=glchr03: (+) length=599 GL50803_ kda 95% 1 Ripening regulated protein DDTFR19 location=glchr05: (+) GL50803_ kda 98% 100% 1 1 SnRNP Sm like protein, putative location=glchr04: (+) length=125 GL50803_ kda 100% 1 Hypothetical protein location=glchr03: ( ) length=223 GL50803_ kda 100% 1 Protein 21.1 location=glchr05: ( ) length=886 GL50803_ kda 100% 1 Hypothetical protein location=glchr05: (+) length=990 GL50803_ kda 100% 1 Hypothetical protein location=glchr05: ( ) length=1086 GL50803_ kda 100% 2 Polyadenylate binding protein, putative location=glchr01: ( ) GL50803_ kda 100% 1 ATP dependent RNA helicase location=glchr05: ( ) length=616 GL50803_ kda 100% 2 ATP dependent RNA helicase p47, putative location=glchr05: (+) GL50803_ kda 100% 2 ACTIN location=glchr02: ( ) length=375 GL50803_ kda 99% 100% 95% Ribosomal protein L7Ae location=glchr04: ( ) length=122 GL50803_ kda 100% 1 ARF GAP location=glchr05: ( ) length=398 GL50803_ kda Hypothetical protein location=glchr04: (+) length=331 GL50803_ kda 100% 2 Ribose 5 phosphate isomerase location=glchr03: ( ) length=145 GL50803_ kda 100% 1 Eukaryotic translation initiation factor 2 beta subunit location=glchr05: GL50803_ kda 100% 2 Hypothetical protein location=glchr03: (+) length=275 GL50803_ kda 95% 100% 95% NADH oxidase location=glchr05: (+) length=429 GL50803_ kda 100% 1 Low molecular weight protein tyrosine phosphatase location=glchr05: GL50803_ kda 100% 2 NADPH oxidoreductase, putative location=glchr05: ( ) length=164 GL50803_ kda 100% 1 Hypothetical protein location=glchr03: ( ) length=137 GL50803_ kda 100% 95% 1 1 Ribosomal protein L5 location=glchr02: (+) length=297 GL50803_ kda 100% 2 Ribosomal protein S27 location=glchr04: (+) length=124 GL50803_ kda 100% 2 Cathepsin B precursor location=glchr04: ( ) length=300 GL50803_ kda 95% 97% 1 1 Translation initiation inhibitor location=glchr02: (+) length=120 GL50803_ kda 100% 2 Ser/Thr protein kinase location=glchr04: ( ) length=869 GL50803_ kda 100% 100% Zinc finger domain location=glchr02: (+) length=128 GL50803_ kda 100% 100% 1 1 Hypothetical protein location=glchr05: (+) length=668 GL50803_ kda 100% 1 Hypothetical protein location=glchr01: (+) length=192 GL50803_ kda 100% 1 RNA binding protein, putative location=glchr05: (+) length=273 GL50803_ kda 100% 2 Heat shock protein, putative location=glchr04: (+) length=845 GL50803_ kda 100% 3 Hypothetical protein location=glchr05: ( ) length=288 GL50803_ kda 100% 2 Hypothetical protein location=glchr03: ( ) length=373 GL50803_ kda 100% 2 Cluster of SKD1 protein location=glchr05: (+) length=454 GL50803_ [2] 50 kda 100% 2 Topoisomerase II location=glchr05: (+) length=519 GL50803_ kda 100% 2 Threonine dehydratase lateral transfer candidate location=glchr04: ( ) GL50803_ kda 100% 1 CGI 35 protein, putative location=glchr01: (+) length=190 GL50803_ kda 100% 1 Kinesin 3 location=glchr04: (+) length=1026 GL50803_ kda 90% 100% 1 2
12 hypothetical protein location=glchr03: (+) length=144 GL50803_ kda 100% 2 Kinesin like protein location=glchr01: ( ) length=796 GL50803_ kda 100% 1 Hypothetical protein location=glchr04: ( ) length=177 GL50803_ kda 100% 1 Phosphatidylinositol 4 phosphate 5 kinase, putative location=glchr02: GL50803_ kda 100% 100% 6 1 Branched chain amino acid aminotransferase lateral transfer candidate GL50803_ kda 100% 100% 1 5 Hypothetical protein location=glchr04: ( ) length=150 GL50803_ kda 100% 1 Ribosomal protein L30 location=glchr05: (+) length=109 GL50803_ kda 100% 2 WD 40 repeat protein family location=glchr01: ( ) length=342 GL50803_ kda 96% 100% 1 2 Ribosomal protein L15 location=glchr02: (+) length=204 GL50803_ kda 100% 100% 100% 100% 100% Eukaryotic translation initiation factor 5A location=glchr05: (+) GL50803_ kda 100% 1 Heat shock protein HSP 90 alpha location=glchr05: (+) length=324 GL50803_ kda 100% 3 Ribosomal protein L44 location=glchr05: ( ) length=106 GL50803_ kda 100% 100% 96% 100% 100% Ribosomal protein L7a location=glchr03: (+) length=225 GL50803_ kda 99% 95% 100% 100% 95% Hypothetical protein location=glchr01: ( ) length=971 GL50803_ kda 100% 100% 1 1 Pyrophosphate fructose 6 phosphate 1 phosphotransferase alpha subunit GL50803_ kda 100% 2 Hypothetical protein location=glchr02: (+) length=479 GL50803_ kda 100% 100% 2 1 Hypothetical protein location=glchr04: ( ) length=680 GL50803_ kda 100% 3 Hypothetical protein location=glchr02: ( ) length=88 GL50803_ kda 100% 2 Liver stage antigen like protein location=glchr03: (+) length=1811 GL50803_ kda 100% 100% 100% 100% Ribosomal protein L9 location=glchr05: ( ) length=185 GL50803_ kda 100% 100% 100% Aminoacyl histidine dipeptidase location=glchr05: (+) length=524 GL50803_ kda 100% 8 Glutamate synthase location=glchr05: ( ) length=910 GL50803_ kda 100% 6 Ribosomal protein L13a location=glchr04: (+) length=197 GL50803_ kda 95% 100% 92% Ribosomal protein L32 location=glchr04: (+) length=136 GL50803_ kda 100% 2 Heat shock protein HSP 90 alpha location=glchr05: (+) length=376 GL50803_ kda 100% 4 Kinase, STE STE20 location=glchr03: ( ) length=527 GL50803_ kda 100% 3 Protein 21.1 location=glchr04: ( ) length=343 GL50803_ kda 100% 4 Chaperone protein DnaJ location=glchr05: ( ) length=409 GL50803_ kda 100% 4 Hypothetical protein location=glchr05: (+) length=367 GL50803_ kda 100% 1 Transketolase location=glchr03: ( ) length=719 GL50803_ kda 100% 3 Glucosamine 6 phosphate N acetyltransferase location=glchr05: ( ) GL50803_ kda 100% 2 Deoxyribose phosphate aldolase lateral transfer candidate location=glchr04: GL50803_ kda 96% 1 Dynamin location=glchr03: (+) length=732 GL50803_ kda 100% 100% 100% 100% 100% Hypothetical protein location=glchr04: (+) length=77 GL50803_ kda 100% 100% 100% methyl 5 thiazole monophosphate biosynthesis enzyme location=glchr02: GL50803_ kda 100% 2 Glycogen phosphorylase location=glchr05: (+) length=924 GL50803_ kda 100% 3 Ribokinase location=glchr01: (+) length=344 GL50803_ kda 100% 2 Ser/Thr protein kinase location=glchr02: (+) length=285 GL50803_ kda 100% 100% 1 2 Protein 21.1 location=glchr05: (+) length=1251 GL50803_ kda 100% 100% 2 1 Hypothetical protein location=glchr02: (+) length=782 GL50803_ kda 100% 2 Hypothetical protein location=glchr03: ( ) length=1025 GL50803_ kda 100% 100% 100% FixW protein, putative location=glchr05: ( ) length=133 GL50803_ kda 100% 2 Adenylate kinase location=glchr01: ( ) length=190 GL50803_ kda 100% 2 Giardia trophozoite antigen GTA 2 location=glchr03: ( ) length=236 GL50803_ kda 100% 96% 99% Ribosomal protein L37 location=glchr02: ( ) length=89 GL50803_ kda 100% 100% 100% 100% 100% Dynein light chain location=glchr05: ( ) length=144 GL50803_ kda 100% 2 Hypothetical protein location=glchr03: ( ) length=1437 GL50803_ kda 100% 2 Hypothetical protein location=glchr04: ( ) length=131 GL50803_ kda 100% 100% 1 1 Ubiquitin conjugating enzyme E2 17 kda 3 location=ch991761: (+) GL50803_ kda 100% 1 Hypothetical protein, similar to AMMECR1 location=glchr05: ( ) GL50803_ kda 100% 1 Ribosomal protein L36 1 location=glchr05: (+) length=90 GL50803_ kda 100% 100% 1 2 Ribosomal protein L35 location=glchr05: ( ) length=152 GL50803_ kda 100% 100% 100% 100% 100% Hypothetical protein location=glchr01: ( ) length=973 GL50803_ kda 100% 100% 100% 100% Hypothetical protein location=glchr05: (+) length=1330 GL50803_ kda 100% 100% 100% Hypothetical protein location=glchr01: (+) length=569 GL50803_ kda 100% 100% 100% 6 4 1
13 Ribosomal protein L35a location=glchr01: (+) length=123 GL50803_ kda 96% 100% 100% Hypothetical protein location=ch991767: (+) length=386 GL50803_ kda 100% 100% 100% Hypothetical protein location=glchr03: ( ) length=383 GL50803_ kda 100% 7 Protein 21.1 location=glchr05: ( ) length=1052 GL50803_ kda 100% 100% 100% 100% Spindle pole protein, putative location=glchr04: ( ) length=623 GL50803_ kda 100% 100% 2 1 hypothetical protein location=glchr03: (+) length=491 GL50803_ kda 95% 100% 1 7 Hypothetical protein location=glchr05: (+) length=507 GL50803_ kda 100% 100% 100% Pyruvate flavodoxin oxidoreductase location=glchr05: ( ) GL50803_ kda 100% 6 Hypothetical protein location=glchr04: (+) length=766 GL50803_ kda 100% 100% 2 1 Protein 21.1 location=glchr05: (+) length=623 GL50803_ kda 100% 100% 1 6 Ribosomal protein S15 location=glchr05: ( ) length=145 GL50803_ kda 100% 100% 2 1 Phosphatidylinositol transfer protein alpha isoform location=glchr02: GL50803_ kda 100% 8 Ribosomal protein L11 location=glchr05: ( ) length=173 GL50803_ kda 95% 95% 100% 100% 97% Pyruvate kinase location=glchr03: ( ) length=654 GL50803_ kda 100% 100% 1 3 Elongation factor 1 gamma location=glchr04: ( ) length=402 GL50803_ kda 98% 100% 1 4 Vacuolar ATP synthase subunit B location=glchr04: (+) length=497 GL50803_ kda 100% 6 ATP dependent RNA helicase like protein location=glchr05: (+) GL50803_ kda 100% 6 Vacuolar ATP synthase catalytic subunit A location=glchr03: (+) GL50803_ kda 100% 6 Protein 21.1 location=glchr03: ( ) length=1156 GL50803_ kda 100% 2 Ubiquitin location=glchr05: (+) length=82 GL50803_ kda 100% 100% 4 1 Hypothetical protein location=glchr01: (+) length=164 GL50803_ kda 100% 3 Hypothetical protein location=glchr04: ( ) length=739 GL50803_ kda 100% 3 Ribosomal protein L24 location=glchr05: (+) length=189 GL50803_ kda 100% 100% 2 1 2,3 bisphosphoglycerate independent phosphoglycerate mutase GL50803_ kda 100% 4 Dynein light chain location=glchr01: ( ) length=89 GL50803_ kda 100% 3 20S proteasome alpha subunit 4 location=glchr05: (+) length=214 GL50803_ kda 100% 2 Cytidine deaminase location=glchr01: (+) length=134 GL50803_ kda 100% 3 Kinase, NEK location=glchr02: (+) length=777 GL50803_ kda 100% 4 Hypothetical protein location=glchr05: (+) length=171 GL50803_ kda 100% 2 Hypothetical protein location=glchr01: ( ) length=621 GL50803_ kda 100% 5 Hypothetical protein location=glchr01: (+) length=176 GL50803_ kda 100% 96% 2 1 GTP binding protein Sar1 location=glchr02: ( ) length=191 GL50803_ kda 100% 1 TCP 1 chaperonin subunit epsilon location=glchr01: ( ) GL50803_ kda 100% 3 Proteasome subunit beta type 5 precursor location=glchr05: (+) GL50803_ kda 100% 3 Ribosomal protein S2 location=glchr05: ( ) length=242 GL50803_ kda 100% 100% 95% 100% 100% 97% Ribosomal protein L24A location=glchr05: (+) length=102 GL50803_ kda 100% 100% 100% Hypothetical protein location=glchr01: ( ) length=993 GL50803_ kda 100% 100% 100% Ribosomal protein S3a location=glchr05: ( ) length=248 GL50803_ kda 99% 100% 100% 100% 100% Ribosomal protein S4 location=glchr04: ( ) length=268 GL50803_ kda 99% 100% 100% 100% 100% Malic enzyme location=glchr05: ( ) length=557 GL50803_ kda 100% 100% 100% Histone H2A location=glchr03: ( ) length=124 GL50803_ kda 95% 95% 100% 100% 100% Hypothetical protein location=glchr04: ( ) length=886 GL50803_ kda 100% 100% 100% 100% Median body protein location=glchr05: (+) length=857 GL50803_ kda 100% 100% 3 2 AAA family ATPase location=glchr04: (+) length=870 GL50803_ kda 100% 9 Alcohol dehydrogenase location=glchr03: (+) length=888 GL50803_ kda 100% 7 Aspartyl trna synthetase location=glchr03: ( ) length=555 GL50803_ kda 100% 100% 2 5 Protein 21.1 location=glchr04: (+) length=786 GL50803_ kda 100% 100% 100% Ribosomal protein L12 location=glchr02: (+) length=182 GL50803_ kda 100% 4 Ribosomal protein S3 location=glchr02: (+) length=217 GL50803_ kda 100% 100% 100% Hypothetical protein location=glchr05: (+) length=985 GL50803_ kda 100% 3 Macrophage migration inhibitory factor location=glchr04: ( ) GL50803_ kda 100% 5 Ribosomal protein S11 location=glchr02: ( ) length=154 GL50803_ kda 99% 100% 96% Histone H3 location=glchr03: ( ) length=159 GL50803_ kda 100% 2 ADP ribosylation factor location=glchr05: (+) length=191 GL50803_ kda 96% 100% 1 3 ATP dependent RNA helicase location=glchr05: (+) length=900 GL50803_ kda 100% 6
14 Hypothetical protein location=glchr05: (+) length=403 GL50803_ kda 100% 5 Hypothetical protein location=glchr05: ( ) length=178 GL50803_ kda 100% 1 Ribosomal protein S21 location=glchr04: (+) length=89 GL50803_ kda 100% 1 Protein F17L21.10 location=glchr05: (+) length=122 GL50803_ kda 100% 2 Spastin location=glchr01: ( ) length=1118 GL50803_ kda 100% 100% 100% 100% 100% Ribosomal protein L10 location=glchr04: ( ) length=210 GL50803_ kda 100% 100% 99% 100% 100% 100% Pyruvate flavodoxin oxidoreductase location=glchr02: (+) GL50803_ kda 100% 100% 1 15 Hypothetical protein location=glchr04: (+) length=1216 GL50803_ kda 100% 100% 100% 100% 100% Ribosomal protein S23 location=glchr05: ( ) length=143 GL50803_ kda 100% 99% 100% 100% Ribosomal protein L23 location=glchr05: ( ) length=142 GL50803_ kda 100% 100% 100% 100% 100% 100% Histone H4 location=ch991767: ( ) length=99 GL50803_ kda 100% 100% 99% 100% Protein 21.6 location=glchr04: ( ) length=854 GL50803_ kda 95% 100% 100% 100% Ribosomal protein L21 location=glchr03: (+) length=159 GL50803_ kda 100% 100% 95% Acetyl CoA synthetase location=glchr03: ( ) length=726 GL50803_ kda 100% 11 Ribosomal protein L34 location=glchr04: ( ) length=120 GL50803_ kda 95% 100% 100% 100% Hypothetical protein location=glchr05: ( ) length=159 GL50803_ kda 100% 99% 9 1 Elongation factor 2 location=glchr04: (+) length=898 GL50803_ kda 100% 100% 2 7 Axoneme associated protein GASP 180 location=glchr05: ( ) GL50803_ kda 100% 11 Peptidyl prolyl cis trans isomerase B precursor location=glchr01: (+) GL50803_ kda 100% 100% 5 1 Nucleoside diphosphate kinase location=glchr04: (+) length=151 GL50803_ kda 100% 6 ATP dependent RNA helicase location=glchr04: ( ) length=625 GL50803_ kda 100% 100% 100% Ribosomal protein L18a location=glchr05: ( ) length=173 GL50803_ kda 100% 3 Wos2 protein location=glchr04: (+) length=185 GL50803_ kda 100% 95% 3 1 Nucleolar GTPase location=glchr04: (+) length=541 GL50803_ kda 100% 10 Acyl CoA synthetase location=glchr04: ( ) length=905 GL50803_ kda 100% 8 Hypothetical protein location=glchr04: ( ) length=281 GL50803_ kda 100% 96% 100% Adenine phosphoribosyltransferase location=glchr01: (+) GL50803_ kda 100% 4 Histone H3 location=glchr04: ( ) length=189 GL50803_ kda 100% 100% 100% 100% 100% 100% 100% 100% Ribosomal protein L3 location=glchr04: (+) length=379 GL50803_ kda 100% 100% 100% 100% 100% Hypothetical protein location=glchr05: ( ) length=367 GL50803_ kda 100% 100% 100% 100% 100% Hypothetical protein location=glchr02: (+) length=281 GL50803_ kda 100% 100% 100% 100% 100% 100% Hypothetical protein location=glchr05: (+) length=137 GL50803_ kda 100% 100% 100% 100% 100% 100% 100% Ribosomal protein L2 location=glchr05: (+) length=251 GL50803_ kda 100% 100% 1 5 BirA GL50803_ kda 100% 100% 100% 99% 100% 100% Giardia trophozoite antigen GTA 1 location=glchr03: ( ) GL50803_ kda 100% 100% 100% 100% 100% Protein 21.1 location=glchr05: (+) length=716 GL50803_ kda 100% 100% 100% Ribosomal protein S14 location=glchr04: (+) length=145 GL50803_ kda 100% 100% 100% Pyruvate, phosphate dikinase location=glchr04: ( ) length=884 GL50803_ kda 100% 12 Uracil phosphoribosyltransferase location=glchr05: ( ) length=211 GL50803_ kda 100% 100% 5 2 Ribosomal protein L27 location=glchr03: (+) length=135 GL50803_ kda 100% 95% 100% Hypothetical protein location=glchr05: (+) length=1330 GL50803_ kda 100% 12 Ribosomal protein S15A location=glchr04: ( ) length=130 GL50803_ kda 100% 100% 3 5 Acyl CoA synthetase location=glchr03: (+) length=970 GL50803_ kda 100% 9 Ribosomal protein S26 location=glchr04: ( ) length=109 GL50803_ kda 96% 100% 100% 100% Acidic ribosomal protein P0 location=glchr05: (+) length=326 GL50803_ kda 100% 98% 100% GTP binding nuclear protein RAN/TC4 location=glchr05: (+) GL50803_ kda 100% 100% 100% Ribosomal protein L37a location=glchr05: (+) length=94 GL50803_ kda 100% 100% 100% 100% 100% 100% 100% Hypothetical protein location=glchr01: (+) length=562 GL50803_ kda 100% 100% 100% 100% 100% Hypothetical protein location=glchr05: (+) length=826 GL50803_ kda 100% 100% 100% 100% Ribosomal protein S18 location=glchr01: ( ) length=154 GL50803_ kda 100% 100% 100% Cytosolic HSP70 location=glchr01: (+) length=664 GL50803_ kda 100% 92% 12 1 Carbamate kinase location=glchr02: (+) length=316 GL50803_ kda 100% 100% 100% Phosphoglycerate kinase location=glchr02: ( ) length=409 GL50803_ kda 100% 100% 11 1 Fructose bisphosphate aldolase location=glchr03: (+) length=323 GL50803_ kda 100% 10 Malate dehydrogenase location=glchr02: (+) length=331 GL50803_ kda 100% 100% 95% 2 4 1
15 Thioredoxin reductase location=glchr02: ( ) length=314 GL50803_ kda 99% 100% 100% Nucleolar GAR1 like protein, putative location=glchr02: (+) GL50803_ kda 100% 95% 4 1 Protein 21.1 location=glchr05: ( ) length=1024 GL50803_ kda 100% 100% 100% 100% 100% Ribosomal protein L4 location=glchr05: ( ) length=316 GL50803_ kda 100% 100% 100% 100% 100% 100% NADP specific glutamate dehydrogenase location=glchr04: ( ) GL50803_ kda 100% 100% 1 20 Ribosomal protein L23A location=glchr04: ( ) length=141 GL50803_ kda 100% 100% 100% 100% 100% 100% Hypothetical protein location=glchr05: (+) length=606 GL50803_ kda 100% 100% 100% 100% 100% Kinase, NEK location=glchr02: (+) length=515 GL50803_ kda 95% 100% 99% Protein 21.1 location=glchr05: (+) length=836 GL50803_ kda 100% 100% 100% 100% Kelch repeat containing protein location=glchr05: ( ) length=1514 GL50803_ kda 100% 7 Hypothetical protein location=glchr04: (+) length=1230 GL50803_ kda 100% 100% 100% 100% 100% Ribosomal protein S24 location=glchr04: (+) length=132 GL50803_ kda 100% 100% 100% 100% 100% Glyceraldehyde 3 phosphate dehydrogenase location=glchr05: ( ) GL50803_ kda 100% 100% 100% Peroxiredoxin 1 location=glchr03: ( ) length=201 GL50803_ kda 100% 100% 100% 100% 96% 100% 100% Ribosomal protein S5 location=ch991769: (+) length=190 GL50803_ kda 96% 99% 100% 100% Ribosomal protein S8 location=glchr03: ( ) length=174 GL50803_ kda 100% 100% 100% 100% 100% 100% Ribosomal protein S9 location=glchr01: ( ) length=189 GL50803_ kda 100% 100% 96% 100% Ribosomal protein S6 location=glchr01: (+) length=248 GL50803_ kda 100% 100% 100% 100% 100% 100% Ribosomal protein S13 location=glchr05: ( ) length=154 GL50803_ kda 100% 100% 100% 100% Ribosomal protein L17 location=glchr05: (+) length=164 GL50803_ kda 100% 100% 100% Ribosomal protein L18 location=glchr04: (+) length=179 GL50803_ kda 95% 100% 100% 100% 100% 100% Protein 21.1 location=glchr03: ( ) length=1146 GL50803_ kda 100% 100% 10 2 Enolase location=glchr05: (+) length=445 GL50803_ kda 100% 100% 100% Xaa Pro dipeptidase location=glchr01: (+) length=444 GL50803_ kda 100% 100% 95% Glycerol 3 phosphate dehydrogenase location=glchr05: ( ) GL50803_ kda 100% 17 Ribosomal protein L26 location=glchr04: (+) length=135 GL50803_ kda 100% 100% 100% 100% 100% 100% Spindle pole protein, putative location=glchr04: (+) length=780 GL50803_ kda 100% 100% 100% 100% 100% Ribosomal protein S17 location=glchr05: ( ) length=137 GL50803_ kda 100% 100% 9 6 UPL 1 location=glchr04: (+) length=310 GL50803_ kda 100% 100% 100% Ribosomal protein S16 location=glchr01: (+) length=158 GL50803_ kda 100% 95% 100% 100% 100% Ribosomal protein L19 location=glchr02: ( ) length=196 GL50803_ kda 100% 100% 100% 100% 100% 100% Ribosomal protein L13 location=glchr01: (+) length=234 GL50803_ kda 100% 100% 100% 100% 100% 100% 100% Alpha tubulin location=glchr03: ( ) length=454 GL50803_ kda 100% 100% 100% 100% 100% 100% 100% 100% Arginine deiminase location=glchr03: ( ) length=580 GL50803_ kda 100% 100% 100% Beta tubulin location=glchr05: ( ) length=447 GL50803_ kda 100% 100% 100% 100% 100% 100% 100% Kinase, NEK location=glchr02: (+) length=1117 GL50803_ kda 100% 100% 100% 100% 100% Hypothetical protein location=glchr05: (+) length=487 GL50803_ kda 100% 100% 100% 100% 100% Hypothetical protein location=glchr05: (+) length=988 GL50803_ kda 100% 100% 100% 100% 100% Elongation factor 1 alpha location=glchr02: (+) length=442 GL50803_ kda 100% 100% 100% 100% 100% 100% 100% Trichohyalin location=glchr04: ( ) length=1026 GL50803_ kda 100% 100% 100% 100% 100% Ribosomal protein L27a location=glchr05: (+) length=149 GL50803_ kda 100% 100% 100% 100% 96% 100% 100% 100% FtsJ cell division protein, putative location=glchr05: (+) GL50803_ kda 100% 100% 100% Ornithine carbamoyltransferase location=glchr03: (+) length=327 GL50803_ kda 100% 100% 100% 100% ATP/GTP binding protein, putative location=glchr04: (+) GL50803_ kda 100% 100% 100% 100% 100% 100% 100% 95% Coiled coil protein location=glchr02: (+) length=1374 GL50803_ kda 100% 100% 100% 100% Spindle pole protein, putative location=glchr01: ( ) length=995 GL50803_ kda 100% 100% 100% 100% 100% Hypothetical protein location=glchr01: ( ) length=385 GL50803_ kda 100% 100% 100% 100% 100% 100% GiOR1 G. intestinalis strain expressing cytosolic biotine ligase (cbira) and GiOR1 fused with biotin acceptor peptide Cia2 G. intestinalis strain expressing cytosolic biotine ligase (cbira) and Cia2 fused with biotin acceptor peptide cbira G. intestinalis strain expressing cbira (control) wt parent G. intestinalis strain (control)
Overlapping sets of transcripts from host and non-host interactions of tomato are expressed early during non-host resistance
 POJ 7(1):19-27 (2014) ISSN:1836-3644 Overlapping sets of transcripts from host and non-host interactions of tomato are expressed early during non-host resistance Battepati Uma and Appa Rao Podile* Supplementary
POJ 7(1):19-27 (2014) ISSN:1836-3644 Overlapping sets of transcripts from host and non-host interactions of tomato are expressed early during non-host resistance Battepati Uma and Appa Rao Podile* Supplementary
Human angiogenin is a potent cytotoxin in the absence of ribonuclease inhibitor
 Human angiogenin is a potent cytotoxin in the absence of ribonuclease inhibitor SYDNEY P. THOMAS, 1 TRISH T. HOANG, 2 VALERIE T. RESSLER, 4 and RONALD T. RAINES 2,3,4 1 Graduate Program in Cell & Molecular
Human angiogenin is a potent cytotoxin in the absence of ribonuclease inhibitor SYDNEY P. THOMAS, 1 TRISH T. HOANG, 2 VALERIE T. RESSLER, 4 and RONALD T. RAINES 2,3,4 1 Graduate Program in Cell & Molecular
JCVI Role Category_1 JCVI Role Category_2. histidinol phosphatase-related protein Unknown function NT01ST0303 AAL19211 STM0248
 Additional Table 3. Detailed information on all 81 genes that showed evidence for positive selection from initial positive selection analysis, in Protein Name Gene name JCVI Role Category_1 JCVI Role Category_2
Additional Table 3. Detailed information on all 81 genes that showed evidence for positive selection from initial positive selection analysis, in Protein Name Gene name JCVI Role Category_1 JCVI Role Category_2
Subject Index. 2D, analyses, 173, electrophoresis, 166
 Subject Index 2D, analyses, 173, electrophoresis, 166 acetate/succinate CoA transferase (ASCT), 8, 9, 104, 115, 116, 118, 119, 126, 128 acetyl, CoA, 104, 119, 121, CoA hydrolase, 126, CoA synthase, 102
Subject Index 2D, analyses, 173, electrophoresis, 166 acetate/succinate CoA transferase (ASCT), 8, 9, 104, 115, 116, 118, 119, 126, 128 acetyl, CoA, 104, 119, 121, CoA hydrolase, 126, CoA synthase, 102
JCVI Locus Protein Name. JCVI Role Category JCVI Locus (LT2) Annotation name
 Additional File 4. Detailed information on all 41 genes that showed evidence for positive selection from positive selection analysis, in which gene Alignment Primary Gene Genbank Locus JCVI Locus Protein
Additional File 4. Detailed information on all 41 genes that showed evidence for positive selection from positive selection analysis, in which gene Alignment Primary Gene Genbank Locus JCVI Locus Protein
Guo_Fig. S1. Atg7 +/+ Atg7 -/- FBP_M+6 DHAP_M+3. Glucose Uptake Rate G6P_M+6. nmol/ul cell/hr
 Guo_Fig. S1 A C D nmol/ul cell/hr nmol/ul cell/hr Glucose Uptake Rate.3.2.1 *. Lactate Secretion Rate.4.3.2.1. Atg7-/- B Glucose G3P Lac Glucose G6P FBP 3-PG Pyr DHAP cycle Carbon from labeled glucose
Guo_Fig. S1 A C D nmol/ul cell/hr nmol/ul cell/hr Glucose Uptake Rate.3.2.1 *. Lactate Secretion Rate.4.3.2.1. Atg7-/- B Glucose G3P Lac Glucose G6P FBP 3-PG Pyr DHAP cycle Carbon from labeled glucose
Α ν α σ κ ό π η σ η. Περίληψη. Dientamoeba fragilis: ένα παραγνωρισμένο παράσιτο του εντέρου
 Α ν α σ κ ό π η σ η Dientamoeba fragilis: ένα παραγνωρισμένο παράσιτο του εντέρου Ε. Βασσάλου 1,2, Κ.Μ. Βασάλος 2, Α. Βακάλη 2, Ν. Βακάλης 2, Ε.-Θ. Πιπεράκη 1, Γ. Βρυώνη 1, Α. Τσακρής 1 1 Εργαστήριο Μικροβιολογίας,
Α ν α σ κ ό π η σ η Dientamoeba fragilis: ένα παραγνωρισμένο παράσιτο του εντέρου Ε. Βασσάλου 1,2, Κ.Μ. Βασάλος 2, Α. Βακάλη 2, Ν. Βακάλης 2, Ε.-Θ. Πιπεράκη 1, Γ. Βρυώνη 1, Α. Τσακρής 1 1 Εργαστήριο Μικροβιολογίας,
Αντιδράσεις οξειδωτικού μέρους. Μη οξειδωτικές αντιδράσεις ΔΡΟΜΟΣ ΤΩΝ ΦΩΣΦΟΤΙΚΩΝ ΠΕΝΤΟΖΩΝ. 6-Ρ γλυκόζη. 5-Ρ ριβουλόζη
 Αντιδράσεις οξειδωτικού μέρους 6-Ρ γλυκόζη 5-Ρ ριβουλόζη ΔΡΟΜΟΣ ΤΩΝ ΦΩΣΦΟΤΙΚΩΝ ΠΕΝΤΟΖΩΝ 5-Ρ ριβόζη (C 5 ) 5-Ρ ξυλουλόζη (C 5 ) GAP (C 3 ) 7-Ρ σεδοεπτουλόζη (C 7 ) 6-Ρ φρουκτόζη(c 6 ) 4-Ρ ερυθρόζη (C 5
Αντιδράσεις οξειδωτικού μέρους 6-Ρ γλυκόζη 5-Ρ ριβουλόζη ΔΡΟΜΟΣ ΤΩΝ ΦΩΣΦΟΤΙΚΩΝ ΠΕΝΤΟΖΩΝ 5-Ρ ριβόζη (C 5 ) 5-Ρ ξυλουλόζη (C 5 ) GAP (C 3 ) 7-Ρ σεδοεπτουλόζη (C 7 ) 6-Ρ φρουκτόζη(c 6 ) 4-Ρ ερυθρόζη (C 5
Ειδικά Κεφάλαια Βιοχηµείας
 Ειδικά Κεφάλαια Βιοχηµείας ΑΠΟΙΚΟΔΟΜΗΣΗ ΠΡΩΤΕΙΝΩΝ-ΠΡΩΤΕΟΣΩΜΑ Δρ. Κ. Ε. Βοργιάς Καθηγητής Βιοχηµείας Τµήµα Βιολογίας 2009 Περιεχόµενα Σελίδα 1. Εισαγωγή 2 2. Τα πρωτεολυτικά ένζυµα συµµετέχουν στην ανακύκλωση
Ειδικά Κεφάλαια Βιοχηµείας ΑΠΟΙΚΟΔΟΜΗΣΗ ΠΡΩΤΕΙΝΩΝ-ΠΡΩΤΕΟΣΩΜΑ Δρ. Κ. Ε. Βοργιάς Καθηγητής Βιοχηµείας Τµήµα Βιολογίας 2009 Περιεχόµενα Σελίδα 1. Εισαγωγή 2 2. Τα πρωτεολυτικά ένζυµα συµµετέχουν στην ανακύκλωση
encouraged to use the Version of Record that, when published, will replace this version. The most /BCJ BIOCHEMICAL JOURNAL
 Biochemical Journal: this is an Accepted Manuscript, not the final Version of Record. You are encouraged to use the Version of Record that, when published, will replace this version. The most up-to-date
Biochemical Journal: this is an Accepted Manuscript, not the final Version of Record. You are encouraged to use the Version of Record that, when published, will replace this version. The most up-to-date
encouraged to use the Version of Record that, when published, will replace this version. The most /BCJ
 Biochemical Journal: this is an Accepted Manuscript, not the final Version of Record. You are encouraged to use the Version of Record that, when published, will replace this version. The most up-to-date
Biochemical Journal: this is an Accepted Manuscript, not the final Version of Record. You are encouraged to use the Version of Record that, when published, will replace this version. The most up-to-date
Supporting Information
 Tissue Level Diet and Sex-by-Diet Interactions Reveal Unique Metabolite and Clustering Profiles Using Untargeted Liquid Chromatography-Mass Spectrometry on Adipose, Skeletal Muscle, and Liver Tissue in
Tissue Level Diet and Sex-by-Diet Interactions Reveal Unique Metabolite and Clustering Profiles Using Untargeted Liquid Chromatography-Mass Spectrometry on Adipose, Skeletal Muscle, and Liver Tissue in
Cellular Physiology and Biochemistry
 Original Paper 2016 The Author(s). 2016 Published The Author(s) by S. Karger AG, Basel Published online: November 25, 2016 www.karger.com/cpb Published by S. Karger AG, Basel 486 www.karger.com/cpb Accepted:
Original Paper 2016 The Author(s). 2016 Published The Author(s) by S. Karger AG, Basel Published online: November 25, 2016 www.karger.com/cpb Published by S. Karger AG, Basel 486 www.karger.com/cpb Accepted:
Μελέτη της αντιμεταλλαξιγόνου δράσης φλαβονοειδών του φυτού Lotus Edulis με τη μέθοδο Ames test
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΙΑΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΒΙΟΧΗΜΕΙΑΣ & ΒΙΟΤΕΧΝΟΛΟΓΙΑΣ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Μελέτη της αντιμεταλλαξιγόνου δράσης φλαβονοειδών του φυτού Lotus Edulis με τη μέθοδο Ames test Επιμέλεια
ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΙΑΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΒΙΟΧΗΜΕΙΑΣ & ΒΙΟΤΕΧΝΟΛΟΓΙΑΣ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Μελέτη της αντιμεταλλαξιγόνου δράσης φλαβονοειδών του φυτού Lotus Edulis με τη μέθοδο Ames test Επιμέλεια
Μελέτη της έκφρασης του ογκοκατασταλτικού γονιδίου Cyld στον καρκίνο του μαστού
 Σχολή Θετικών Επιστημών Τμήμα Βιολογίας Πρόγραμμα Μεταπτυχιακών Σπουδών Κατεύθυνση: Εφαρμοσμένη γενετική και βιοτεχνολογία ΜΕΤΑΠΤΥΧΙΑΚΗ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Μελέτη της έκφρασης του ογκοκατασταλτικού γονιδίου
Σχολή Θετικών Επιστημών Τμήμα Βιολογίας Πρόγραμμα Μεταπτυχιακών Σπουδών Κατεύθυνση: Εφαρμοσμένη γενετική και βιοτεχνολογία ΜΕΤΑΠΤΥΧΙΑΚΗ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Μελέτη της έκφρασης του ογκοκατασταλτικού γονιδίου
encouraged to use the Version of Record that, when published, will replace this version. The most /BCJ
 Biochemical Journal: this is an Accepted Manuscript, not the final Version of Record. You are encouraged to use the Version of Record that, when published, will replace this version. The most up-to-date
Biochemical Journal: this is an Accepted Manuscript, not the final Version of Record. You are encouraged to use the Version of Record that, when published, will replace this version. The most up-to-date
Acknowledgements... 3 Contents... I Figures... VII Tables... IX Abbreviations... X
 I Acknowledgements... 3... I Figures... VII Tables... IX Abbreviations... X Amino acids... XII Nucleobases... XII 1 Introduction... 1 1.1 Polyketides and non-ribosomal peptides... 1 1.2 Polyketide synthases...
I Acknowledgements... 3... I Figures... VII Tables... IX Abbreviations... X Amino acids... XII Nucleobases... XII 1 Introduction... 1 1.1 Polyketides and non-ribosomal peptides... 1 1.2 Polyketide synthases...
ΠΡΟΣΚΛΗΣΗ ΕΚΔΗΛΩΣΗΣ ΕΝΔΙΑΦΕΡΟΝΤΟΣ
 1 ΕΛΛΗΝΙΚΗ ΔΗΜΟΚΡΑΤΙΑ ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΡΗΤΗΣ ΕΙΔΙΚΟΣ ΛΟΓΑΡΙΑΣΜΟΣ ΑΝΑΡΤΗΤΕΑ ΣΤΟ ΔΙΑΔΙΚΤΥΟ ΠΡΟΣΚΛΗΣΗ ΕΚΔΗΛΩΣΗΣ ΕΝΔΙΑΦΕΡΟΝΤΟΣ Ηράκλειο, 04.02.2014 Αρ. πρωτ. 1054 Ο Ειδικός Λογαριασμός του Πανεπιστημίου Κρήτης
1 ΕΛΛΗΝΙΚΗ ΔΗΜΟΚΡΑΤΙΑ ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΡΗΤΗΣ ΕΙΔΙΚΟΣ ΛΟΓΑΡΙΑΣΜΟΣ ΑΝΑΡΤΗΤΕΑ ΣΤΟ ΔΙΑΔΙΚΤΥΟ ΠΡΟΣΚΛΗΣΗ ΕΚΔΗΛΩΣΗΣ ΕΝΔΙΑΦΕΡΟΝΤΟΣ Ηράκλειο, 04.02.2014 Αρ. πρωτ. 1054 Ο Ειδικός Λογαριασμός του Πανεπιστημίου Κρήτης
Supplementary figures for MED13 dependent signaling from the heart confers leanness by enhancing metabolism in adipose tissue and liver.
 Supplementary figures for MED13 dependent signaling from the heart confers leanness by enhancing metabolism in adipose tissue and liver. Content: Figure S1. Cardiac overexpression of MED13 increases metabolic
Supplementary figures for MED13 dependent signaling from the heart confers leanness by enhancing metabolism in adipose tissue and liver. Content: Figure S1. Cardiac overexpression of MED13 increases metabolic
The effect of curcumin on the stability of Aβ. dimers
 The effect of curcumin on the stability of Aβ dimers Li Na Zhao, See-Wing Chiu, Jérôme Benoit, Lock Yue Chew,, and Yuguang Mu, School of Physical and Mathematical Sciences, Nanyang Technological University,
The effect of curcumin on the stability of Aβ dimers Li Na Zhao, See-Wing Chiu, Jérôme Benoit, Lock Yue Chew,, and Yuguang Mu, School of Physical and Mathematical Sciences, Nanyang Technological University,
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΓΕΩΠΟΝΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΕΠΙΣΤΗΜΗΣ ΚΑΙ ΤΕΧΝΟΛΟΓΙΑΣ ΤΡΟΦΙΜΩΝ ΜΑΡΙΑΣ ΦΩΤΙΟΥ ΠΤΥΧΙΟΥΧΟΥ ΓΕΩΠΟΝΟΥ
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΓΕΩΠΟΝΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΕΠΙΣΤΗΜΗΣ ΚΑΙ ΤΕΧΝΟΛΟΓΙΑΣ ΤΡΟΦΙΜΩΝ ΜΑΡΙΑΣ ΦΩΤΙΟΥ ΠΤΥΧΙΟΥΧΟΥ ΓΕΩΠΟΝΟΥ Συγκέντρωση των ελεύθερων αµινοξέων στο αµνιακό υγρό σε σχέση µε την εβδοµάδα
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΓΕΩΠΟΝΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΕΠΙΣΤΗΜΗΣ ΚΑΙ ΤΕΧΝΟΛΟΓΙΑΣ ΤΡΟΦΙΜΩΝ ΜΑΡΙΑΣ ΦΩΤΙΟΥ ΠΤΥΧΙΟΥΧΟΥ ΓΕΩΠΟΝΟΥ Συγκέντρωση των ελεύθερων αµινοξέων στο αµνιακό υγρό σε σχέση µε την εβδοµάδα
Supporting Information
 Supporting Information Figure S1. The functionality of the tagged Arp6 and Swr1 was confirmed by monitoring cell growth and sensitivity to hydeoxyurea (HU). Five-fold serial dilutions of each strain were
Supporting Information Figure S1. The functionality of the tagged Arp6 and Swr1 was confirmed by monitoring cell growth and sensitivity to hydeoxyurea (HU). Five-fold serial dilutions of each strain were
ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΣΤΙΣ «ΚΛΙΝΙΚΕΣ ΚΑΙ ΚΛΙΝΙΚΟΕΡΓΑΣΤΗΡΙΑΚΕΣ ΙΑΤΡΙΚΕΣ ΕΙΔΙΚΟΤΗΤΕΣ»
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΣΤΙΣ «ΚΛΙΝΙΚΕΣ ΚΑΙ ΚΛΙΝΙΚΟΕΡΓΑΣΤΗΡΙΑΚΕΣ ΙΑΤΡΙΚΕΣ ΕΙΔΙΚΟΤΗΤΕΣ» ΑΡΝΗΤΙΚΗ ΡΥΘΜΙΣΗ ΤΗΣ ΜΕΤΑΒΙΒΑΣΗΣ ΤΟΥ ΣΗΜΑΤΟΣ ΤΗΣ
ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΣΤΙΣ «ΚΛΙΝΙΚΕΣ ΚΑΙ ΚΛΙΝΙΚΟΕΡΓΑΣΤΗΡΙΑΚΕΣ ΙΑΤΡΙΚΕΣ ΕΙΔΙΚΟΤΗΤΕΣ» ΑΡΝΗΤΙΚΗ ΡΥΘΜΙΣΗ ΤΗΣ ΜΕΤΑΒΙΒΑΣΗΣ ΤΟΥ ΣΗΜΑΤΟΣ ΤΗΣ
Microcosm Setup and Biomass Sampling. Seawater for microcosm incubation. August 16, 2007 during the CMORE Bloomer Cruise. Hydrocasts for sampling were
 SUPPORTING INFORMATION SUPPORTING TEXT METHODS Microcosm Setup and Biomass Sampling. Seawater for microcosm incubation experiments was collected at 23 12.88 N, 159 8.17 W, from 75-m depth, pre-dawn on
SUPPORTING INFORMATION SUPPORTING TEXT METHODS Microcosm Setup and Biomass Sampling. Seawater for microcosm incubation experiments was collected at 23 12.88 N, 159 8.17 W, from 75-m depth, pre-dawn on
«ΑΓΡΟΤΟΥΡΙΣΜΟΣ ΚΑΙ ΤΟΠΙΚΗ ΑΝΑΠΤΥΞΗ: Ο ΡΟΛΟΣ ΤΩΝ ΝΕΩΝ ΤΕΧΝΟΛΟΓΙΩΝ ΣΤΗΝ ΠΡΟΩΘΗΣΗ ΤΩΝ ΓΥΝΑΙΚΕΙΩΝ ΣΥΝΕΤΑΙΡΙΣΜΩΝ»
 I ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΝΟΜΙΚΩΝ ΟΙΚΟΝΟΜΙΚΩΝ ΚΑΙ ΠΟΛΙΤΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΤΜΗΜΑ ΟΙΚΟΝΟΜΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΣΤΗΝ «ΔΙΟΙΚΗΣΗ ΚΑΙ ΟΙΚΟΝΟΜΙΑ» ΚΑΤΕΥΘΥΝΣΗ: ΟΙΚΟΝΟΜΙΚΗ
I ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΝΟΜΙΚΩΝ ΟΙΚΟΝΟΜΙΚΩΝ ΚΑΙ ΠΟΛΙΤΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΤΜΗΜΑ ΟΙΚΟΝΟΜΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΣΤΗΝ «ΔΙΟΙΚΗΣΗ ΚΑΙ ΟΙΚΟΝΟΜΙΑ» ΚΑΤΕΥΘΥΝΣΗ: ΟΙΚΟΝΟΜΙΚΗ
Figures Supplementary Figure 1. Tandem MS of yeast eif2b.
 Figures Supplementary Figure 1. Tandem MS of yeast eif2b. Tandem MS of the 56+ charge state (shaded blue) of the eif2b dimer showing that two α subunits ( at 2000 m/z) dissociate from the dimer when collision
Figures Supplementary Figure 1. Tandem MS of yeast eif2b. Tandem MS of the 56+ charge state (shaded blue) of the eif2b dimer showing that two α subunits ( at 2000 m/z) dissociate from the dimer when collision
Γλυκόζη. Ανασκόπηση μεταβολισμού υδατανθρακών ΗΠΑΡ. ΤΡΟΦΗ Γλυκόζη. Κυκλοφορία. Κυτταρόπλασμα ΓΛΥΚΟΓΟΝΟ. Οδός Φωσφ. Πεντοζών.
 ΓΛΥΚΟΛΥΣΗ ΙΙ Ανασκόπηση μεταβολισμού υδατανθρακών ΗΠΑΡ ΤΡΟΦΗ Γλυκόζη Κυκλοφορία ΓΛΥΚΟΓΟΝΟ Γλυκόζη ΗΠΑΡ Κυτταρόπλασμα ΗΠΑΡ (Οξέωση) NADH ΟΞΕΙΔ. ΦΩΣΦ. ATP CO 2 Φρουκτόζη Γαλακτόζη 2ATP+2NADH Αναερόβια Γαλακτικό
ΓΛΥΚΟΛΥΣΗ ΙΙ Ανασκόπηση μεταβολισμού υδατανθρακών ΗΠΑΡ ΤΡΟΦΗ Γλυκόζη Κυκλοφορία ΓΛΥΚΟΓΟΝΟ Γλυκόζη ΗΠΑΡ Κυτταρόπλασμα ΗΠΑΡ (Οξέωση) NADH ΟΞΕΙΔ. ΦΩΣΦ. ATP CO 2 Φρουκτόζη Γαλακτόζη 2ATP+2NADH Αναερόβια Γαλακτικό
Table S2. Sequences immunoprecipitating with anti-hif-1α
 Genome-wide analysis of HIF DNA-bdg-supplemental material Table S2. Sequences immunoprecipitatg with anti-α The table gives the chromosomal co-ordates of each anti-α immunoprecipitated locus meetg criteria
Genome-wide analysis of HIF DNA-bdg-supplemental material Table S2. Sequences immunoprecipitatg with anti-α The table gives the chromosomal co-ordates of each anti-α immunoprecipitated locus meetg criteria
Supporting Information
 Supporting Information Copyright Wiley-VCH Verlag GmbH & Co. KGaA, 69451 Weinheim, 2013 Structure Activity Relationship Study Reveals ML240 and ML241 as Potent and Selective Inhibitors of p97 ATPase Tsui-Fen
Supporting Information Copyright Wiley-VCH Verlag GmbH & Co. KGaA, 69451 Weinheim, 2013 Structure Activity Relationship Study Reveals ML240 and ML241 as Potent and Selective Inhibitors of p97 ATPase Tsui-Fen
5- CACGAAACTACCTTCAACTCC-3 beta actin-r 5- CATACTCCTGCTTGCTGATC-3 GAPDH-F GAPDH-R
 Table S1 Primers used in this work LncPPARδ-F 5- AGGATCCCAAGGCAGAAGTT -3 LncPPARδ-R 5- GAATAGAAAGGCGGGAAAGG -3 PPARδ-F 5- CACCCAGTGTCCTTCCATCT -3 PPARδ-R 5- CAGAATTGGGTGTGCTGCTT -3 ADRP-F 5- ACAACCGAGTGTGGTGACTC
Table S1 Primers used in this work LncPPARδ-F 5- AGGATCCCAAGGCAGAAGTT -3 LncPPARδ-R 5- GAATAGAAAGGCGGGAAAGG -3 PPARδ-F 5- CACCCAGTGTCCTTCCATCT -3 PPARδ-R 5- CAGAATTGGGTGTGCTGCTT -3 ADRP-F 5- ACAACCGAGTGTGGTGACTC
Molecular evolutionary dynamics of respiratory syncytial virus group A in
 Molecular evolutionary dynamics of respiratory syncytial virus group A in recurrent epidemics in coastal Kenya James R. Otieno 1#, Charles N. Agoti 1, 2, Caroline W. Gitahi 1, Ann Bett 1, Mwanajuma Ngama
Molecular evolutionary dynamics of respiratory syncytial virus group A in recurrent epidemics in coastal Kenya James R. Otieno 1#, Charles N. Agoti 1, 2, Caroline W. Gitahi 1, Ann Bett 1, Mwanajuma Ngama
Μαρία Κατσιφοδήμου. Ο ρόλος της έκκρισης HLA-G από τα ανθρώπινα έμβρυα στην επιτυχία της εξωσωματικής γονιμοποίησης. Μεταπτυχιακή Διπλωματική Εργασία
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΘΕΤΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΤΜΗΜΑ ΒΙΟΛΟΓΙΑΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΚΑΤΕΥΘΥΝΣΗ: ΕΦΑΡΜΟΣΜΕΝΗ ΓΕΝΕΤΙΚΗ ΚΑΙ ΒΙΟΤΕΧΝΟΛΟΓΙΑ Ο ρόλος της έκκρισης HLA-G από τα ανθρώπινα
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΘΕΤΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΤΜΗΜΑ ΒΙΟΛΟΓΙΑΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΚΑΤΕΥΘΥΝΣΗ: ΕΦΑΡΜΟΣΜΕΝΗ ΓΕΝΕΤΙΚΗ ΚΑΙ ΒΙΟΤΕΧΝΟΛΟΓΙΑ Ο ρόλος της έκκρισης HLA-G από τα ανθρώπινα
High mobility group 1 HMG1
 Vol. 29, pp.705 ~ 711, 2001 High mobility group 1 HMG1 13 12 20 anti-neutrophil cytoplasmic antibodies, ANCA ANCA 1982 Davies 1980 1 high mobility group HMG1 HMG2 30 kd high mobility group HMGHMG HMG1
Vol. 29, pp.705 ~ 711, 2001 High mobility group 1 HMG1 13 12 20 anti-neutrophil cytoplasmic antibodies, ANCA ANCA 1982 Davies 1980 1 high mobility group HMG1 HMG2 30 kd high mobility group HMGHMG HMG1
abs acces acces Sample test results. Actual results may vary. Antioxidants B-Vitamins Minerals Order Today At Results Overview
 Order Today At www.accesalabs. Antioxidants B-Vitamins Minerals Vitamin C Pyridoxine - Biotin Vitamin A / Carotenoids Vitamin E / Tocopher α-lipoic A Co T ami B1 cer R bo a n - B2 N acin - B3 Results Overview
Order Today At www.accesalabs. Antioxidants B-Vitamins Minerals Vitamin C Pyridoxine - Biotin Vitamin A / Carotenoids Vitamin E / Tocopher α-lipoic A Co T ami B1 cer R bo a n - B2 N acin - B3 Results Overview
Ε.Φ.Ο.Α. - Βαθμολογία 2014 (βδ.24) - Αγόρια U18 (best4) κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ Βαθμ b18 1 1 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998
 b18 1 1 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 820.0 b18 2 2 25438 ΤΣΙΤΣΙΠΑΣ ΣΤΕΦΑΝΟΣ 1998 Ο.Α.ΓΛΥΦΑΔΑΣ ΙΑ 770.0 b18 3 3 24845 ΗΛΙΟΠΟΥΛΟΣ ΒΑΣΙΛΗΣ 1998 Α.Ο.Α.ΦΙΛΟΘΕΗΣ Η 750.0 b18 4 4 21565 ΘΕΟΔΩΡΟΥ
b18 1 1 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 820.0 b18 2 2 25438 ΤΣΙΤΣΙΠΑΣ ΣΤΕΦΑΝΟΣ 1998 Ο.Α.ΓΛΥΦΑΔΑΣ ΙΑ 770.0 b18 3 3 24845 ΗΛΙΟΠΟΥΛΟΣ ΒΑΣΙΛΗΣ 1998 Α.Ο.Α.ΦΙΛΟΘΕΗΣ Η 750.0 b18 4 4 21565 ΘΕΟΔΩΡΟΥ
Η ΦΛΕΓΜΟΝΩ ΗΣ ΑΝΤΙ ΡΑΣΗ ΤΟΥ ΓΑΣΤΡΙΚΟΥ ΒΛΕΝΝΟΓΟΝΟΥ ΣΤΗ ΛΟΙΜΩΞΗ ΜΕ ΕΛΙΚΟΒΑΚΤΗΡΙ ΙΟ ΤΟΥ ΠΥΛΩΡΟΥ ΠΡΙΝ ΚΑΙ ΜΕΤΑ ΤΗ ΘΕΡΑΠΕΙΑ
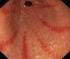 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ ΤΟΜΕΑΣ ΥΓΕΙΑΣ ΕΡΓΑΣΤΗΡΙΑΚΟΣ ΙΕΥΘΥΝΤΗΣ: Ο ΚΑΘΗΓΗΤΗΣ ΓΕΩΡΓΙΟΣ ΚΑΡΚΑΒΕΛΑΣ ΠΑΝΕΠ. ΕΤΟΣ 2008-2009 Αριθµ. 2084 Η ΦΛΕΓΜΟΝΩ ΗΣ ΑΝΤΙ
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ ΤΟΜΕΑΣ ΥΓΕΙΑΣ ΕΡΓΑΣΤΗΡΙΑΚΟΣ ΙΕΥΘΥΝΤΗΣ: Ο ΚΑΘΗΓΗΤΗΣ ΓΕΩΡΓΙΟΣ ΚΑΡΚΑΒΕΛΑΣ ΠΑΝΕΠ. ΕΤΟΣ 2008-2009 Αριθµ. 2084 Η ΦΛΕΓΜΟΝΩ ΗΣ ΑΝΤΙ
SUPPLEMENTARY INFORMATION
 Sensitivity of [Ru(phen) 2 dppz] 2+ Light Switch Emission to Ionic Strength, Temperature, and DNA Sequence and Conformation Andrew W. McKinley, Per Lincoln and Eimer M. Tuite* SUPPLEMENTARY INFORMATION
Sensitivity of [Ru(phen) 2 dppz] 2+ Light Switch Emission to Ionic Strength, Temperature, and DNA Sequence and Conformation Andrew W. McKinley, Per Lincoln and Eimer M. Tuite* SUPPLEMENTARY INFORMATION
Data-Driven Discovery of Extravasation Pathway in Circulating Tumor Cells
 Data-Driven Discovery of Extravasation Pathway in Circulating Tumor Cells Yadavalli S. 1, Jayaram S. 1,2, Manda SS. 1,3, Madugundu AK. 1,3, Nayakanti DS. 1, Tuan TZ. 6, Bhat R. 4, Rangarajan A. 4, Chatterjee
Data-Driven Discovery of Extravasation Pathway in Circulating Tumor Cells Yadavalli S. 1, Jayaram S. 1,2, Manda SS. 1,3, Madugundu AK. 1,3, Nayakanti DS. 1, Tuan TZ. 6, Bhat R. 4, Rangarajan A. 4, Chatterjee
ΚΥΤΤΑΡΙΚΟΣ ΘΑΝΑΤΟΣ ΚΑΙ ΑΝΑΓΕΝΝΗΣΗ Β-ΚΥΤΤΑΡΟΥ ΝΕΟΤΕΡΑ ΔΕΔΟΜΕΝΑ ΓΙΑ ΤΗ ΘΕΡΑΠΕΙΑ ΤΟΥ ΣΑΚΧΑΡΩΔΗ ΔΙΑΒΗΤΗ
 ΚΥΤΤΑΡΙΚΟΣ ΘΑΝΑΤΟΣ ΚΑΙ ΑΝΑΓΕΝΝΗΣΗ Β-ΚΥΤΤΑΡΟΥ ΝΕΟΤΕΡΑ ΔΕΔΟΜΕΝΑ ΓΙΑ ΤΗ ΘΕΡΑΠΕΙΑ ΤΟΥ ΣΑΚΧΑΡΩΔΗ ΔΙΑΒΗΤΗ ΚΑΛΛΙΟΠΗ ΚΩΤΣΑ ΕΠΙΚ. ΚΑΘΗΓΗΤΡΙΑ ΕΝΔΟΚΡΙΝΟΛΟΓΙΑΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ Α.Π.Θ ΤΜΗΜΑ ΕΝΔΟΚΡΙΝΟΛΟΓΙΑΣ-ΔΙΑΒΗΤΟΛΟΓΙΚΟ
ΚΥΤΤΑΡΙΚΟΣ ΘΑΝΑΤΟΣ ΚΑΙ ΑΝΑΓΕΝΝΗΣΗ Β-ΚΥΤΤΑΡΟΥ ΝΕΟΤΕΡΑ ΔΕΔΟΜΕΝΑ ΓΙΑ ΤΗ ΘΕΡΑΠΕΙΑ ΤΟΥ ΣΑΚΧΑΡΩΔΗ ΔΙΑΒΗΤΗ ΚΑΛΛΙΟΠΗ ΚΩΤΣΑ ΕΠΙΚ. ΚΑΘΗΓΗΤΡΙΑ ΕΝΔΟΚΡΙΝΟΛΟΓΙΑΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ Α.Π.Θ ΤΜΗΜΑ ΕΝΔΟΚΡΙΝΟΛΟΓΙΑΣ-ΔΙΑΒΗΤΟΛΟΓΙΚΟ
ΕΦΑΡΜΟΓΗ ΕΥΤΕΡΟΒΑΘΜΙΑ ΕΠΕΞΕΡΓΑΣΜΕΝΩΝ ΥΓΡΩΝ ΑΠΟΒΛΗΤΩΝ ΣΕ ΦΥΣΙΚΑ ΣΥΣΤΗΜΑΤΑ ΚΛΙΝΗΣ ΚΑΛΑΜΙΩΝ
 ΤΕΧΝΟΛΟΓΙΚΟ ΕΚΠΑΙ ΕΥΤΙΚΟ Ι ΡΥΜΑ ΚΡΗΤΗΣ ΤΜΗΜΑ ΦΥΣΙΚΩΝ ΠΟΡΩΝ ΚΑΙ ΠΕΡΙΒΑΛΛΟΝΤΟΣ ΕΦΑΡΜΟΓΗ ΕΥΤΕΡΟΒΑΘΜΙΑ ΕΠΕΞΕΡΓΑΣΜΕΝΩΝ ΥΓΡΩΝ ΑΠΟΒΛΗΤΩΝ ΣΕ ΦΥΣΙΚΑ ΣΥΣΤΗΜΑΤΑ ΚΛΙΝΗΣ ΚΑΛΑΜΙΩΝ ΕΠΙΜΕΛΕΙΑ: ΑΡΜΕΝΑΚΑΣ ΜΑΡΙΝΟΣ ΧΑΝΙΑ
ΤΕΧΝΟΛΟΓΙΚΟ ΕΚΠΑΙ ΕΥΤΙΚΟ Ι ΡΥΜΑ ΚΡΗΤΗΣ ΤΜΗΜΑ ΦΥΣΙΚΩΝ ΠΟΡΩΝ ΚΑΙ ΠΕΡΙΒΑΛΛΟΝΤΟΣ ΕΦΑΡΜΟΓΗ ΕΥΤΕΡΟΒΑΘΜΙΑ ΕΠΕΞΕΡΓΑΣΜΕΝΩΝ ΥΓΡΩΝ ΑΠΟΒΛΗΤΩΝ ΣΕ ΦΥΣΙΚΑ ΣΥΣΤΗΜΑΤΑ ΚΛΙΝΗΣ ΚΑΛΑΜΙΩΝ ΕΠΙΜΕΛΕΙΑ: ΑΡΜΕΝΑΚΑΣ ΜΑΡΙΝΟΣ ΧΑΝΙΑ
Ενδοκυττάρια ιαµερίσµατα, ιαλογή και µεταφορά πρωτεινών
 Ενδοκυττάρια ιαµερίσµατα, ιαλογή και µεταφορά πρωτεινών Ένα ηπατικό κύτταρο σε διατοµή Η.Μ Compartment Main Function Cytosol contains many metabolic pathways (Chapters 3 and 4); protein synthesis (Chapter
Ενδοκυττάρια ιαµερίσµατα, ιαλογή και µεταφορά πρωτεινών Ένα ηπατικό κύτταρο σε διατοµή Η.Μ Compartment Main Function Cytosol contains many metabolic pathways (Chapters 3 and 4); protein synthesis (Chapter
ΜΕΛΕΤΗ ΤΗΣ ΕΠΙΔΗΜΙΟΛΟΓΙΑΣ ΚΑΙ ΤΗΣ ΚΛΙΝΙΚΗΣ ΣΗΜΑΣΙΑΣ ΤΗΣ ΛΟΙΜΩΞΗΣ ΤΗΣ ΓΑΤΑΣ ΑΠΟ LEISHMANIA SPP.
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΙΑΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΚΤΗΝΙΑΤΡΙΚΗΣ ΜΕΛΕΤΗ ΤΗΣ ΕΠΙΔΗΜΙΟΛΟΓΙΑΣ ΚΑΙ ΤΗΣ ΚΛΙΝΙΚΗΣ ΣΗΜΑΣΙΑΣ ΤΗΣ ΛΟΙΜΩΞΗΣ ΤΗΣ ΓΑΤΑΣ ΑΠΟ LEISHMANIA SPP. ΕΜΜΑΝΟΥΗΛ Κ. ΧΑΤΖΗΣ, ΚΤΗΝΙΑΤΡΟΣ ΔΙΔΑΚΤΟΡΙΚΗ
ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΙΑΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΚΤΗΝΙΑΤΡΙΚΗΣ ΜΕΛΕΤΗ ΤΗΣ ΕΠΙΔΗΜΙΟΛΟΓΙΑΣ ΚΑΙ ΤΗΣ ΚΛΙΝΙΚΗΣ ΣΗΜΑΣΙΑΣ ΤΗΣ ΛΟΙΜΩΞΗΣ ΤΗΣ ΓΑΤΑΣ ΑΠΟ LEISHMANIA SPP. ΕΜΜΑΝΟΥΗΛ Κ. ΧΑΤΖΗΣ, ΚΤΗΝΙΑΤΡΟΣ ΔΙΔΑΚΤΟΡΙΚΗ
ΤΑΥΤΟΠΟΙΗΣΗ ΝΕΩΝ ΑΥΤΟΑΝΤΙΓΟΝΩΝ ΣΤΟΧΩΝ ΤΗΣ ΝΟΣΟΕΙΔΙΚΗΣ ΑΝΟΣΙΑΚΗΣ ΑΠΟΚΡΙΣΗΣ ΜΕ ΜΕΘΟΔΟΥΣ ΑΝΟΣΟΑΠΟΤΥΠΩΣΗΣ ΚΑΙ MALDI-TOF ΑΝΑΛΥΣΗΣ
 ΕΡΕΥΝΗΤΙΚΗ ΜΟΝΑΔΑ: Κ.Α.Μ.Α ΥΠΕΥΘΥΝΟΣ: Δ. Π. ΜΠΟΓΔΑΝΟΣ ΤΑΥΤΟΠΟΙΗΣΗ ΝΕΩΝ ΑΥΤΟΑΝΤΙΓΟΝΩΝ ΣΤΟΧΩΝ ΤΗΣ ΝΟΣΟΕΙΔΙΚΗΣ ΑΝΟΣΙΑΚΗΣ ΑΠΟΚΡΙΣΗΣ ΜΕ ΜΕΘΟΔΟΥΣ ΑΝΟΣΟΑΠΟΤΥΠΩΣΗΣ ΚΑΙ MALDI-TOF ΑΝΑΛΥΣΗΣ Μαρία Μυτιληναίου ΑΥΤΟΑΝΟΣΑ
ΕΡΕΥΝΗΤΙΚΗ ΜΟΝΑΔΑ: Κ.Α.Μ.Α ΥΠΕΥΘΥΝΟΣ: Δ. Π. ΜΠΟΓΔΑΝΟΣ ΤΑΥΤΟΠΟΙΗΣΗ ΝΕΩΝ ΑΥΤΟΑΝΤΙΓΟΝΩΝ ΣΤΟΧΩΝ ΤΗΣ ΝΟΣΟΕΙΔΙΚΗΣ ΑΝΟΣΙΑΚΗΣ ΑΠΟΚΡΙΣΗΣ ΜΕ ΜΕΘΟΔΟΥΣ ΑΝΟΣΟΑΠΟΤΥΠΩΣΗΣ ΚΑΙ MALDI-TOF ΑΝΑΛΥΣΗΣ Μαρία Μυτιληναίου ΑΥΤΟΑΝΟΣΑ
ΥΠΕΡΙΝΣΟΥΛΙΝΑΙΜΙΑ ΚΑΙ ΑΝΤΙΣΤΑΣΗ ΣΤΗΝ ΙΝΣΟΥΛΙΝΗ ΣΕ ΠΑΧΥΣΑΡΚΑ ΠΑΙΔΙΑ ΚΑΙ ΕΦΗΒΟΥΣ ΜΕ ΠΡΩΙΜΗ ΑΔΡΕΝΑΡΧΗ
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΚΑΙ ΠΡΟΛΗΠΤΙΚΗΣ ΙΑΤΡΙΚΗΣ ΕΡΓΑΣΤΗΡΙΟ ΒΙΟΧΗΜΕΙΑΣ ΔΙΕΥΘΥΝΤΡΙΑ: Η ΚΑΘΗΓΗΤΡΙΑ Ν. ΒΑΒΑΤΣΗ-ΧΡΙΣΤΑΚΗ ΠΑΝΕΠ. ΕΤΟΣ 2009-2010 Αριθμ.2456
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΚΑΙ ΠΡΟΛΗΠΤΙΚΗΣ ΙΑΤΡΙΚΗΣ ΕΡΓΑΣΤΗΡΙΟ ΒΙΟΧΗΜΕΙΑΣ ΔΙΕΥΘΥΝΤΡΙΑ: Η ΚΑΘΗΓΗΤΡΙΑ Ν. ΒΑΒΑΤΣΗ-ΧΡΙΣΤΑΚΗ ΠΑΝΕΠ. ΕΤΟΣ 2009-2010 Αριθμ.2456
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ. ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥ ΩΝ «Ιατρική Ερευνητική Μεθοδολογία» ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥ ΩΝ «Ιατρική Ερευνητική Μεθοδολογία» ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Νόσος Pompe: Νεότερα κλινικά, γενετικά, διαγνωστικά και θεραπευτικά
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥ ΩΝ «Ιατρική Ερευνητική Μεθοδολογία» ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Νόσος Pompe: Νεότερα κλινικά, γενετικά, διαγνωστικά και θεραπευτικά
± 20% ± 5% ± 10% RENCO ELECTRONICS, INC.
 RL15 RL16, RL17, RL18 MINIINDUCTORS CONFORMALLY COATED MARKING The nominal inductance is marked by a color code as listed in the table below. Color Black Brown Red Orange Yellow Green Blue Purple Grey
RL15 RL16, RL17, RL18 MINIINDUCTORS CONFORMALLY COATED MARKING The nominal inductance is marked by a color code as listed in the table below. Color Black Brown Red Orange Yellow Green Blue Purple Grey
ΙΑΤΑΡΑΧΕΣ ΜΕΤΑΒΟΛΙΣΜΟΥ ΛΙΠΑΡΩΝ ΟΞΕΩΝ
 Ηµ/νία Εξέτασης : ΙΑΤΑΡΑΧΕΣ ΜΕΤΑΒΟΛΙΣΜΟΥ ΛΙΠΑΡΩΝ ΟΞΕΩΝ Medium Chain Acyl CoA Dehydrogenase C6< 0.24 C8< 0.35 C10< 0.3 C10:1 < 0.22 C8/C2< 0.02 C8/C10 < 3 Long Chain 3 Hydroxyacyl Coa Dehydrogenase C16OH
Ηµ/νία Εξέτασης : ΙΑΤΑΡΑΧΕΣ ΜΕΤΑΒΟΛΙΣΜΟΥ ΛΙΠΑΡΩΝ ΟΞΕΩΝ Medium Chain Acyl CoA Dehydrogenase C6< 0.24 C8< 0.35 C10< 0.3 C10:1 < 0.22 C8/C2< 0.02 C8/C10 < 3 Long Chain 3 Hydroxyacyl Coa Dehydrogenase C16OH
Strain gauge and rosettes
 Strain gauge and rosettes Introduction A strain gauge is a device which is used to measure strain (deformation) on an object subjected to forces. Strain can be measured using various types of devices classified
Strain gauge and rosettes Introduction A strain gauge is a device which is used to measure strain (deformation) on an object subjected to forces. Strain can be measured using various types of devices classified
Ε.Φ.Ο.Α. - Βαθμολογία 2015 (βδ.47) - Κορίτσια U16 (best 8μ+3δ) κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ tours Βαθμ g16 1 1 22833 ΑΔΑΛΟΓΛΟΥ
 g16 1 1 22833 ΑΔΑΛΟΓΛΟΥ ΜΑΓΔΑΛΗΝΗ 1999 Ε.Σ.Ο.ΕΠΙΚΟΥΡΟΣ ΠΟΛΙΧΝΗΣ Β 10 917.5 g16 2 2 90069 ΜΤΣΕΝΤΛΙΤΖΕ ΕΛΕΝΗ 2000 Α.Ο.Α.ΣΤΑΥΡΟΥΠΟΛΗΣ ΙΦΙΤΟΣ Β 7 666.0 g16 3 3 28688 ΣΤΑΜΑΤΟΓΙΑΝΝΟΠΟΥΛΟΥ ΒΑΣΙΛΙΚΗ 2001 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ
g16 1 1 22833 ΑΔΑΛΟΓΛΟΥ ΜΑΓΔΑΛΗΝΗ 1999 Ε.Σ.Ο.ΕΠΙΚΟΥΡΟΣ ΠΟΛΙΧΝΗΣ Β 10 917.5 g16 2 2 90069 ΜΤΣΕΝΤΛΙΤΖΕ ΕΛΕΝΗ 2000 Α.Ο.Α.ΣΤΑΥΡΟΥΠΟΛΗΣ ΙΦΙΤΟΣ Β 7 666.0 g16 3 3 28688 ΣΤΑΜΑΤΟΓΙΑΝΝΟΠΟΥΛΟΥ ΒΑΣΙΛΙΚΗ 2001 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ
Ε.Φ.Ο.Α. - Βαθμολογία 2014 (βδ.31) - Αγόρια U18 (best4) κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ Βαθμ b18 1 1 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998
 b18 1 1 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 810.0 b18 2 2 24845 ΗΛΙΟΠΟΥΛΟΣ ΒΑΣΙΛΗΣ 1998 Α.Ο.Α.ΦΙΛΟΘΕΗΣ Η 690.0 b18 3 3 23517 ΤΣΙΡΑΝΙΔΗΣ ΕΥΣΤΑΘΙΟΣ 1998 Ο.Α.ΚΑΣΤΟΡΙΑΣ ΚΕΛΕΤΡΟΝ Γ 680.0 b18 4
b18 1 1 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 810.0 b18 2 2 24845 ΗΛΙΟΠΟΥΛΟΣ ΒΑΣΙΛΗΣ 1998 Α.Ο.Α.ΦΙΛΟΘΕΗΣ Η 690.0 b18 3 3 23517 ΤΣΙΡΑΝΙΔΗΣ ΕΥΣΤΑΘΙΟΣ 1998 Ο.Α.ΚΑΣΤΟΡΙΑΣ ΚΕΛΕΤΡΟΝ Γ 680.0 b18 4
Ε.Φ.Ο.Α. - Βαθμολογία 2015 (βδ.12) - Αγόρια U18 (best4) κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ Βαθμ b18 1 1 23517 ΤΣΙΡΑΝΙΔΗΣ ΕΥΣΤΑΘΙΟΣ 1998
 b18 1 1 23517 ΤΣΙΡΑΝΙΔΗΣ ΕΥΣΤΑΘΙΟΣ 1998 Ο.Α.ΘΕΣΣΑΛΟΝΙΚΗΣ Β 797.5 b18 2 2 22969 ΚΑΝΕΛΛΟΠΟΥΛΟΣ ΑΛΕΞΑΝΔΡΟΣ 1997 Α.Ο.ΤΑΤΟΪΟΥ Η 747.0 b18 3 3 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 692.5 b18 4 4
b18 1 1 23517 ΤΣΙΡΑΝΙΔΗΣ ΕΥΣΤΑΘΙΟΣ 1998 Ο.Α.ΘΕΣΣΑΛΟΝΙΚΗΣ Β 797.5 b18 2 2 22969 ΚΑΝΕΛΛΟΠΟΥΛΟΣ ΑΛΕΞΑΝΔΡΟΣ 1997 Α.Ο.ΤΑΤΟΪΟΥ Η 747.0 b18 3 3 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 692.5 b18 4 4
ΩΡΟΛΟΓΙΟ ΠΡΟΓΡΑΜΜΑ ΜΑΘΗΜΑΤΩΝ
 ΤΜΗΜΑ Α1 1η ΩΡΑ ΠΑΠΟΥΤΣΗ ΣΟΦΟΥΛΗ ΣΟΦΟΥΛΗ ΟΞΟΠΟΥΛΟΥ ΣΟΦΟΥΛΗ 2η ΩΡΑ ΝΙΚΟΛΟΠΟΥΛΟΥ ΣΠΑΝΟΣ ΣΟΦΟΥΛΗ ΓΙΑΝΝΑ ΑΚΗΣ ΣΠΑΝΟΣ 3η ΩΡΑ ΜΑΡΚΑΝΤΩΝΗΣ ΜΑΡΚΑΝΤΩΝΗΣ ΚΑΛΑΒΡΙΖΙΩΤΗΣ ΧΡΙΣΤΟ ΟΥΛΗ ΝΙΚΟΛΟΠΟΥΛΟΥ 4η ΩΡΑ ΚΑΛΑΒΡΙΖΙΩΤΗΣ
ΤΜΗΜΑ Α1 1η ΩΡΑ ΠΑΠΟΥΤΣΗ ΣΟΦΟΥΛΗ ΣΟΦΟΥΛΗ ΟΞΟΠΟΥΛΟΥ ΣΟΦΟΥΛΗ 2η ΩΡΑ ΝΙΚΟΛΟΠΟΥΛΟΥ ΣΠΑΝΟΣ ΣΟΦΟΥΛΗ ΓΙΑΝΝΑ ΑΚΗΣ ΣΠΑΝΟΣ 3η ΩΡΑ ΜΑΡΚΑΝΤΩΝΗΣ ΜΑΡΚΑΝΤΩΝΗΣ ΚΑΛΑΒΡΙΖΙΩΤΗΣ ΧΡΙΣΤΟ ΟΥΛΗ ΝΙΚΟΛΟΠΟΥΛΟΥ 4η ΩΡΑ ΚΑΛΑΒΡΙΖΙΩΤΗΣ
Proteomics. Laurent Gatto 1 CSAMA 27 June
 Proteomics Laurent Gatto 1 http://cpu.sysbiol.cam.ac.uk CSAMA 27 June 2014 1 lg390@cam.ac.uk Outline Proteomics and MS data Ranges infrastructure Application: spatial proteomics Mass-spectrometry (LC-MSMS)
Proteomics Laurent Gatto 1 http://cpu.sysbiol.cam.ac.uk CSAMA 27 June 2014 1 lg390@cam.ac.uk Outline Proteomics and MS data Ranges infrastructure Application: spatial proteomics Mass-spectrometry (LC-MSMS)
Page 1 of 14. α/α Α.Μ. Ονοματεπώνυμο Σύλλογος Έτος Βαθμοί Κατηγορία ΑΓΟΡΙΑ 10
 ΑΓΟΡΙΑ 10 1 36030 ΤΟΥΝΤΑΣ ΜΑΡΙΟΣ ΡΗΓΑΣ Α.Ο.Α.ΑΡΓΟΛΙΔΑΣ 2005 b10 2 35955 ΖΑΧΑΡΑΚΗΣ ΚΩΝΣΤΑΝΤΙΝΟΣ Ο.Α.ΣΑΛΑΜΙΝΑΣ 2005 b10 3 34580 ΠΑΠΑΔΟΠΟΥΛΟΣ ΙΑΣΟΝΑΣ Α.Ο.ΤΑΤΟΪΟΥ 2005 b10 4 35959 ΚΟΚΚΙΝΟΣ ΙΑΣΩΝ-ΝΙΚΟΛΑΟΣ Α.Ο.Α.ΠΑΠΑΓΟΥ
ΑΓΟΡΙΑ 10 1 36030 ΤΟΥΝΤΑΣ ΜΑΡΙΟΣ ΡΗΓΑΣ Α.Ο.Α.ΑΡΓΟΛΙΔΑΣ 2005 b10 2 35955 ΖΑΧΑΡΑΚΗΣ ΚΩΝΣΤΑΝΤΙΝΟΣ Ο.Α.ΣΑΛΑΜΙΝΑΣ 2005 b10 3 34580 ΠΑΠΑΔΟΠΟΥΛΟΣ ΙΑΣΟΝΑΣ Α.Ο.ΤΑΤΟΪΟΥ 2005 b10 4 35959 ΚΟΚΚΙΝΟΣ ΙΑΣΩΝ-ΝΙΚΟΛΑΟΣ Α.Ο.Α.ΠΑΠΑΓΟΥ
Ε.Φ.Ο.Α. - Βαθμολογία 2015 (βδ.12) - Αγόρια U12 (best8) κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ Βαθμ b12 1 1 32605 ΚΥΠΡΙΩΤΗΣ ΕΥΑΓΓΕΛΟΣ 2003
 b12 1 1 32605 ΚΥΠΡΙΩΤΗΣ ΕΥΑΓΓΕΛΟΣ 2003 Ο.Α.ΑΘΗΝΩΝ Η 194.0 b12 2 2 31353 ΜΗΤΣΑΚΟΣ ΘΕΟΔΩΡΟΣ 2004 ΡΗΓΑΣ Α.Ο.Α.ΑΡΓΟΛΙΔΑΣ ΣΤ 74.5 b12 3 3 32680 ΦΩΤΕΙΝΟΠΟΥΛΟΣ ΑΘΑΝΑΣΙΟΣ 2003 Α.Ο.Α.ΗΛΙΟΥΠΟΛΗΣ ΙΑ 68.5 b12 4 4
b12 1 1 32605 ΚΥΠΡΙΩΤΗΣ ΕΥΑΓΓΕΛΟΣ 2003 Ο.Α.ΑΘΗΝΩΝ Η 194.0 b12 2 2 31353 ΜΗΤΣΑΚΟΣ ΘΕΟΔΩΡΟΣ 2004 ΡΗΓΑΣ Α.Ο.Α.ΑΡΓΟΛΙΔΑΣ ΣΤ 74.5 b12 3 3 32680 ΦΩΤΕΙΝΟΠΟΥΛΟΣ ΑΘΑΝΑΣΙΟΣ 2003 Α.Ο.Α.ΗΛΙΟΥΠΟΛΗΣ ΙΑ 68.5 b12 4 4
Ε.Φ.Ο.Α. - Βαθμολογία 2015 (βδ.12) - Αγόρια U16 (best8) κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ Βαθμ b16 1 1 30186 ΠΙΤΣΙΝΗΣ ΔΗΜΗΤΡΙΟΣ 1999
 b16 1 1 30186 ΠΙΤΣΙΝΗΣ ΔΗΜΗΤΡΙΟΣ 1999 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 810.0 b16 2 2 26317 ΚΩΣΤΑΡΑΣ ΠΑΝΑΓΙΩΤΗΣ 1999 Ο.Α.ΠΕΤΡΟΥΠΟΛΗΣ Θ 804.0 b16 3 3 25297 ΚΑΠΙΡΗΣ ΣΤΑΜΑΤΗΣ 1999 Α.Ο.Α.ΗΛΙΟΥΠΟΛΗΣ ΙΑ 682.0 b16 4 4 29817
b16 1 1 30186 ΠΙΤΣΙΝΗΣ ΔΗΜΗΤΡΙΟΣ 1999 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 810.0 b16 2 2 26317 ΚΩΣΤΑΡΑΣ ΠΑΝΑΓΙΩΤΗΣ 1999 Ο.Α.ΠΕΤΡΟΥΠΟΛΗΣ Θ 804.0 b16 3 3 25297 ΚΑΠΙΡΗΣ ΣΤΑΜΑΤΗΣ 1999 Α.Ο.Α.ΗΛΙΟΥΠΟΛΗΣ ΙΑ 682.0 b16 4 4 29817
Supporting Information
 Chloromethylhalicyclamine B, a Marine-Derived Protein Kinase CK1δ/ε Inhibitor Germana Esposito, Marie-Lise Bourguet-Kondracki, * Linh H. Mai, Arlette Longeon, Roberta Teta, Laurent Meijer, Rob Van Soest,
Chloromethylhalicyclamine B, a Marine-Derived Protein Kinase CK1δ/ε Inhibitor Germana Esposito, Marie-Lise Bourguet-Kondracki, * Linh H. Mai, Arlette Longeon, Roberta Teta, Laurent Meijer, Rob Van Soest,
Ο Παιδίατρος μπροστά στην επιλογή βρεφικού γάλακτος
 Ο Παιδίατρος μπροστά στην επιλογή βρεφικού γάλακτος Κ. Σαραφίδης Επίκουρος Καθηγητής Α.Π.Θ. Α Νεογνολογική Κλινική και ΜΕΝΝ Α.Π.Θ. Μητρικό γάλα Καλύτερη πηγή τροφής για τα βρέφη τον 1 ο χρόνο ζωής Αποτελεί
Ο Παιδίατρος μπροστά στην επιλογή βρεφικού γάλακτος Κ. Σαραφίδης Επίκουρος Καθηγητής Α.Π.Θ. Α Νεογνολογική Κλινική και ΜΕΝΝ Α.Π.Θ. Μητρικό γάλα Καλύτερη πηγή τροφής για τα βρέφη τον 1 ο χρόνο ζωής Αποτελεί
Supplemental Table 1. Oligonucleotides used to identify H-RAS, K-RAS and N-RAS mutations and PTEN gene expression.
 Supplemental Table 1. Oligonucleotides used to identify H-RAS, K-RAS and N-RAS mutations and PTEN gene expression. Gene Exon Forward primer Reverse primer Temp ( C) HRAS 2-3 ATGACGGAATATAAGCTGGT ATGGCAAACACACACAGGAA
Supplemental Table 1. Oligonucleotides used to identify H-RAS, K-RAS and N-RAS mutations and PTEN gene expression. Gene Exon Forward primer Reverse primer Temp ( C) HRAS 2-3 ATGACGGAATATAAGCTGGT ATGGCAAACACACACAGGAA
ΜΟΡΙΑΚΕΣ ΜΕΘΟΔΟΙ ΚΡΙΤΗΡΙΑ ΕΠΙΛΟΓΗΣ ΑΞΙΟΛΟΓΗΣΗ
 - - ό ό : Ω ί ό ί ώ.. ά ή ί ός ή ς ύ ί ί ή έ ώ ό. ή ς ά ς ής ό ός ά ς ή έ ός ή ς ί ής ί ς. ό έ ώ - ώ ή ής ή ς- ί ά ά ς ί ς. ά ί ί έ έ ά ύ ή, ό ί ά, ό ό ά έ ά ής ί ύ George Wald ή ί έ ς ί ύ ό ς ί ς ά έ
- - ό ό : Ω ί ό ί ώ.. ά ή ί ός ή ς ύ ί ί ή έ ώ ό. ή ς ά ς ής ό ός ά ς ή έ ός ή ς ί ής ί ς. ό έ ώ - ώ ή ής ή ς- ί ά ά ς ί ς. ά ί ί έ έ ά ύ ή, ό ί ά, ό ό ά έ ά ής ί ύ George Wald ή ί έ ς ί ύ ό ς ί ς ά έ
ΠΕΡΙΛΗΨΗ ΧΑΡΑΚΤΗΡΙΣΤΙΚΩΝ ΤΟΥ ΠΡΟΙΟΝΤΟΣ (SPC) (AMINOVEN 3.5% Glucose / Electrolytes)
 ΠΕΡΙΛΗΨΗ ΧΑΡΑΚΤΗΡΙΣΤΙΚΩΝ ΤΟΥ ΠΡΟΙΟΝΤΟΣ (SPC) (AMINOVEN 3.5% Glucose / Electrolytes) 1. ΕΜΠΟΡΙΚΗ ΟΝΟΜΑΣΙΑ ΤΟΥ ΦΑΡΜΑΚΕΥΤΙΚΟΥ ΠΡΟΙΟΝΤΟΣ: AMINOVEN 3.5% Glucose / Electrolytes 2. ΠΟΙΟΤΙΚΗ ΚΑΙ ΠΟΣΟΤΙΚΗ ΣΥΝΘΕΣΗ
ΠΕΡΙΛΗΨΗ ΧΑΡΑΚΤΗΡΙΣΤΙΚΩΝ ΤΟΥ ΠΡΟΙΟΝΤΟΣ (SPC) (AMINOVEN 3.5% Glucose / Electrolytes) 1. ΕΜΠΟΡΙΚΗ ΟΝΟΜΑΣΙΑ ΤΟΥ ΦΑΡΜΑΚΕΥΤΙΚΟΥ ΠΡΟΙΟΝΤΟΣ: AMINOVEN 3.5% Glucose / Electrolytes 2. ΠΟΙΟΤΙΚΗ ΚΑΙ ΠΟΣΟΤΙΚΗ ΣΥΝΘΕΣΗ
Supplementary Materials
 Supplementary Materials Figure S1. Chemical structures of phloracetophenone intermediates (2a, 2c 2g). 2a 2c 2d 2e 2f 2g Figure S2. Chemical structures of geranylacetophenone analogues (3a, 3c, 3e g).
Supplementary Materials Figure S1. Chemical structures of phloracetophenone intermediates (2a, 2c 2g). 2a 2c 2d 2e 2f 2g Figure S2. Chemical structures of geranylacetophenone analogues (3a, 3c, 3e g).
ΠΑΤΡΩΝΥΜΟ / ΟΝΟΜΑ ΣΥΖΥΓΟΥ 1 ΑΓΟΡΑΣΤΟΥ ΜΑΡΙΑ ΤΟΥ ΔΗΜΗΤΡΙΟΥ 2 ΑΘΑΝΑΣΙΑΔΗΣ ΙΩΑΝΝΗΣ ΤΟΥ ΠΑΥΛΟΥ 3 ΑΚΤΣΟΓΛΟΥ ΣΩΚΡΑΤΗΣ ΤΟΥ ΓΕΩΡΓΙΟΥ
 Υποψήφιοι ημοτικοί Σύμβουλοι: ΠΑΤΡΩΝΥΜΟ / ΣΥΖΥΓΟΥ 1 ΑΓΟΡΑΣΤΟΥ ΜΑΡΙΑ ΤΟΥ ΔΗΜΗΤΡΙΟΥ 2 ΑΘΑΝΑΣΙΑΔΗΣ ΙΩΑΝΝΗΣ ΤΟΥ ΠΑΥΛΟΥ 3 ΑΚΤΣΟΓΛΟΥ ΣΩΚΡΑΤΗΣ ΤΟΥ ΓΕΩΡΓΙΟΥ 4 ΑΛΦΑΤΖΗΣ ΙΩΑΝΝΗΣ ΤΟΥ ΓΕΩΡΓΙΟΥ 5 ΑΜΟΡΓΙΑΝΟΣ ΝΙΚΟΛΑΟΣ
Υποψήφιοι ημοτικοί Σύμβουλοι: ΠΑΤΡΩΝΥΜΟ / ΣΥΖΥΓΟΥ 1 ΑΓΟΡΑΣΤΟΥ ΜΑΡΙΑ ΤΟΥ ΔΗΜΗΤΡΙΟΥ 2 ΑΘΑΝΑΣΙΑΔΗΣ ΙΩΑΝΝΗΣ ΤΟΥ ΠΑΥΛΟΥ 3 ΑΚΤΣΟΓΛΟΥ ΣΩΚΡΑΤΗΣ ΤΟΥ ΓΕΩΡΓΙΟΥ 4 ΑΛΦΑΤΖΗΣ ΙΩΑΝΝΗΣ ΤΟΥ ΓΕΩΡΓΙΟΥ 5 ΑΜΟΡΓΙΑΝΟΣ ΝΙΚΟΛΑΟΣ
Fixed Inductors / AL TYPE
 .Features: 1.Coating epoxy resin that ensures the humidity resistance to be long life. 2.Contribute to be high Q and selfresonant frequencies.applications: 1.Electronics products. 2.Communication equipment.
.Features: 1.Coating epoxy resin that ensures the humidity resistance to be long life. 2.Contribute to be high Q and selfresonant frequencies.applications: 1.Electronics products. 2.Communication equipment.
Βιολογία Γενικής Παιδείας Β Λυκείου
 Απρίλιος Μάιος 12 Βιολογία Γενικής Παιδείας Β Λυκείου Δημοσθένης Καρυοφύλλης Βιολογία Γενικής Παιδείας Β Λυκείου (Ερωτήσεις που παρουσιάζουν ενδιαφέρον) 1. Τι είναι τα βιομόρια και ποια είναι τα βασικά
Απρίλιος Μάιος 12 Βιολογία Γενικής Παιδείας Β Λυκείου Δημοσθένης Καρυοφύλλης Βιολογία Γενικής Παιδείας Β Λυκείου (Ερωτήσεις που παρουσιάζουν ενδιαφέρον) 1. Τι είναι τα βιομόρια και ποια είναι τα βασικά
Inverse trigonometric functions & General Solution of Trigonometric Equations. ------------------ ----------------------------- -----------------
 Inverse trigonometric functions & General Solution of Trigonometric Equations. 1. Sin ( ) = a) b) c) d) Ans b. Solution : Method 1. Ans a: 17 > 1 a) is rejected. w.k.t Sin ( sin ) = d is rejected. If sin
Inverse trigonometric functions & General Solution of Trigonometric Equations. 1. Sin ( ) = a) b) c) d) Ans b. Solution : Method 1. Ans a: 17 > 1 a) is rejected. w.k.t Sin ( sin ) = d is rejected. If sin
CRC Croce Tili List of patients for mir125b Metabolomic study Clinical data update 13/01/12
 AGGRESSIVE CRC Croce Tili List of patients for mir125b Metabolomic study Clinical data update 13/01/12 INDOLENT Table S1. Clinical data of the patients used for the study ID ZAP %VH WBC Dx date First TX
AGGRESSIVE CRC Croce Tili List of patients for mir125b Metabolomic study Clinical data update 13/01/12 INDOLENT Table S1. Clinical data of the patients used for the study ID ZAP %VH WBC Dx date First TX
Εισαγωγή στις πρωτεΐνες Δομή πρωτεϊνών Ταξινόμηση βάσει δομής Βάσεις με δομές πρωτεϊνών Ευθυγράμμιση δομών Πρόβλεψη 2D δομής Πρόβλεψη 3D δομής
 Εισαγωγή στις πρωτεΐνες Δομή πρωτεϊνών Ταξινόμηση βάσει δομής Βάσεις με δομές πρωτεϊνών Ευθυγράμμιση δομών Πρόβλεψη 2D δομής Πρόβλεψη 3D δομής Τι είναι η πρωτεΐνη Τι εννοούμε με δομή πρωτεϊνών Οικογένειες
Εισαγωγή στις πρωτεΐνες Δομή πρωτεϊνών Ταξινόμηση βάσει δομής Βάσεις με δομές πρωτεϊνών Ευθυγράμμιση δομών Πρόβλεψη 2D δομής Πρόβλεψη 3D δομής Τι είναι η πρωτεΐνη Τι εννοούμε με δομή πρωτεϊνών Οικογένειες
Th, Ra, Rn, Po, Pb, Bi, & Tl K x-rays. Rn Kα1. Rn Kα2. 93( 227 Th)/Rn Kβ3. Ra Kα2. Po Kα2 /Bi K α1 79( 227 Th)/Po Kα1. Ra Kα1 /Bi K β1.
 Page -1-10 8 10 7 10 6 10 5 10 4 334 ( Th) Counts/Channel 10 3 10 2 10 1 49 ( Th)/ 50 ( Th)/ 50 ( Fr) 0 100 200 300 400 500 600 700 800 900 1000 1000 1100 1200 1300 1400 1500 1600 1700 1800 1900 2000 Channel
Page -1-10 8 10 7 10 6 10 5 10 4 334 ( Th) Counts/Channel 10 3 10 2 10 1 49 ( Th)/ 50 ( Th)/ 50 ( Fr) 0 100 200 300 400 500 600 700 800 900 1000 1000 1100 1200 1300 1400 1500 1600 1700 1800 1900 2000 Channel
ΕΚΤΙΜΗΣΗ ΤΟΥ ΚΟΣΤΟΥΣ ΤΩΝ ΟΔΙΚΩΝ ΑΤΥΧΗΜΑΤΩΝ ΚΑΙ ΔΙΕΡΕΥΝΗΣΗ ΤΩΝ ΠΑΡΑΓΟΝΤΩΝ ΕΠΙΡΡΟΗΣ ΤΟΥ
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΠΟΛΥΤΕΧΝΙΚΗ ΣΧΟΛΗ ΤΜΗΜΑ ΠΟΛΙΤΙΚΩΝ ΜΗΧΑΝΙΚΩΝ ΕΚΤΙΜΗΣΗ ΤΟΥ ΚΟΣΤΟΥΣ ΤΩΝ ΟΔΙΚΩΝ ΑΤΥΧΗΜΑΤΩΝ ΚΑΙ ΔΙΕΡΕΥΝΗΣΗ ΤΩΝ ΠΑΡΑΓΟΝΤΩΝ ΕΠΙΡΡΟΗΣ ΤΟΥ ΔΙΑΤΡΙΒΗ ΔΙΠΛΩΜΑΤΟΣ ΕΙΔΙΚΕΥΣΗΣ ΔΗΜΗΤΡΙΟΥ Ν. ΠΙΤΕΡΟΥ
ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΠΟΛΥΤΕΧΝΙΚΗ ΣΧΟΛΗ ΤΜΗΜΑ ΠΟΛΙΤΙΚΩΝ ΜΗΧΑΝΙΚΩΝ ΕΚΤΙΜΗΣΗ ΤΟΥ ΚΟΣΤΟΥΣ ΤΩΝ ΟΔΙΚΩΝ ΑΤΥΧΗΜΑΤΩΝ ΚΑΙ ΔΙΕΡΕΥΝΗΣΗ ΤΩΝ ΠΑΡΑΓΟΝΤΩΝ ΕΠΙΡΡΟΗΣ ΤΟΥ ΔΙΑΤΡΙΒΗ ΔΙΠΛΩΜΑΤΟΣ ΕΙΔΙΚΕΥΣΗΣ ΔΗΜΗΤΡΙΟΥ Ν. ΠΙΤΕΡΟΥ
Ε.Φ.Ο.Α. - Βαθμολογία 2014 (βδ.01) - Αγόρια U16 κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ Βαθμ b16 1 1 25438 ΤΣΙΤΣΙΠΑΣ ΣΤΕΦΑΝΟΣ 1998 Ο.Α.
 b16 1 1 25438 ΤΣΙΤΣΙΠΑΣ ΣΤΕΦΑΝΟΣ 1998 Ο.Α.ΓΛΥΦΑΔΑΣ ΙΑ 1270,0 b16 2 2 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 1180,0 b16 3 3 24845 ΗΛΙΟΠΟΥΛΟΣ ΒΑΣΙΛΗΣ 1998 Α.Ο.Α.ΦΙΛΟΘΕΗΣ Η 1030,0 b16 4 4 23517
b16 1 1 25438 ΤΣΙΤΣΙΠΑΣ ΣΤΕΦΑΝΟΣ 1998 Ο.Α.ΓΛΥΦΑΔΑΣ ΙΑ 1270,0 b16 2 2 23775 ΑΝΤΩΝΟΠΟΥΛΟΣ ΧΡΗΣΤΟΣ 1998 Ο.Α.ΚΕΡΑΤΣΙΝΙΟΥ Θ 1180,0 b16 3 3 24845 ΗΛΙΟΠΟΥΛΟΣ ΒΑΣΙΛΗΣ 1998 Α.Ο.Α.ΦΙΛΟΘΕΗΣ Η 1030,0 b16 4 4 23517
Ε.Φ.Ο.Α. - Βαθμολογία 2014 (βδ.43) - Κορίτσια U12 (best8) κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ Βαθμ g12 1 1 31873 ΓΡΙΒΑ ΒΑΣΙΛΕΙΑ 2002 ΑΙΟΛΟΣ
 g12 1 1 31873 ΓΡΙΒΑ ΒΑΣΙΛΕΙΑ 2002 ΑΙΟΛΟΣ Α.Λ.ΙΛΙΟΥ Θ 226.0 g12 2 2 29169 ΝΤΑΝΟΥ ΧΡΙΣΤΙΝΑ 2002 Ο.Α.ΓΟΥΔΙΟΥ ΙΑ 186.0 g12 3 3 31551 ΠΑΠΑΚΩΝΣΤΑΝΤΙΝΟΥ ΕΛΕΝΑ-ΜΑΡΙΑ 2002 ΦΘΙΩΤΙΚΟΣ Ο.Α. Ε 184.5 g12 4 4 30176 ΓΙΑΝΝΑΚΟΥ
g12 1 1 31873 ΓΡΙΒΑ ΒΑΣΙΛΕΙΑ 2002 ΑΙΟΛΟΣ Α.Λ.ΙΛΙΟΥ Θ 226.0 g12 2 2 29169 ΝΤΑΝΟΥ ΧΡΙΣΤΙΝΑ 2002 Ο.Α.ΓΟΥΔΙΟΥ ΙΑ 186.0 g12 3 3 31551 ΠΑΠΑΚΩΝΣΤΑΝΤΙΝΟΥ ΕΛΕΝΑ-ΜΑΡΙΑ 2002 ΦΘΙΩΤΙΚΟΣ Ο.Α. Ε 184.5 g12 4 4 30176 ΓΙΑΝΝΑΚΟΥ
Ε.Φ.Ο.Α. - Βαθμολογία 2014 (βδ.31) - Κορίτσια U12 (best8) κτγρ # αα ΑΜ Ονοματεπώνυμο Έτος Σύλλογος ΕΝ Βαθμ g12 1 1 31873 ΓΡΙΒΑ ΒΑΣΙΛΕΙΑ 2002 ΑΙΟΛΟΣ
 g12 1 1 31873 ΓΡΙΒΑ ΒΑΣΙΛΕΙΑ 2002 ΑΙΟΛΟΣ Α.Λ.ΙΛΙΟΥ Θ 210.0 g12 2 2 30176 ΓΙΑΝΝΑΚΟΥ ΙΩΑΝΝΑ 2002 Α.Ο.Α.ΠΟΣΕΙΔΩΝ ΘΕΣΣΑΛΟΝΙΚΗΣ Β 161.0 g12 3 3 31551 ΠΑΠΑΚΩΝΣΤΑΝΤΙΝΟΥ ΕΛΕΝΑ-ΜΑΡΙΑ 2002 ΦΘΙΩΤΙΚΟΣ Ο.Α. Ε 155.0
g12 1 1 31873 ΓΡΙΒΑ ΒΑΣΙΛΕΙΑ 2002 ΑΙΟΛΟΣ Α.Λ.ΙΛΙΟΥ Θ 210.0 g12 2 2 30176 ΓΙΑΝΝΑΚΟΥ ΙΩΑΝΝΑ 2002 Α.Ο.Α.ΠΟΣΕΙΔΩΝ ΘΕΣΣΑΛΟΝΙΚΗΣ Β 161.0 g12 3 3 31551 ΠΑΠΑΚΩΝΣΤΑΝΤΙΝΟΥ ΕΛΕΝΑ-ΜΑΡΙΑ 2002 ΦΘΙΩΤΙΚΟΣ Ο.Α. Ε 155.0
Ο ΡΟΛΟΣ ΤΩΝ ΚΑΝΝΑΒΙΝΟΕΙ ΩΝ ΚΑΤΑ ΤΗΝ ΕΜΒΡΥΪΚΗ ΑΝΑΠΤΥΞΗ
 ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ Πρόγραµµα Μεταπτυχιακών Σπουδών Εφαρµογές στις Βασικές Ιατρικές Επιστήµες Εργαστήριο Ανατοµικής-Ιστολογίας-Εµβρυολογίας ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Ο ΡΟΛΟΣ ΤΩΝ ΚΑΝΝΑΒΙΝΟΕΙ
ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ Πρόγραµµα Μεταπτυχιακών Σπουδών Εφαρµογές στις Βασικές Ιατρικές Επιστήµες Εργαστήριο Ανατοµικής-Ιστολογίας-Εµβρυολογίας ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Ο ΡΟΛΟΣ ΤΩΝ ΚΑΝΝΑΒΙΝΟΕΙ
ΙΩΑΝΝΗ ΑΘ. ΠΑΠΑΪΩΑΝΝΟΥ
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΓΕΩΠΟΝΙΑΣ, ΔΑΣΟΛΟΓΙΑΣ ΚΑΙ ΦΥΣΙΚΟΥ ΠΕΡΙΒΑΛΛΟΝΤΟΣ ΤΜΗΜΑ ΓΕΩΠΟΝΙΑΣ ΤΟΜΕΑΣ ΑΓΡΟΤΙΚΗΣ ΟΙΚΟΝΟΜΙΑΣ ΙΩΑΝΝΗ ΑΘ. ΠΑΠΑΪΩΑΝΝΟΥ Πτυχιούχου Γεωπόνου Κατόχου Μεταπτυχιακού
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΓΕΩΠΟΝΙΑΣ, ΔΑΣΟΛΟΓΙΑΣ ΚΑΙ ΦΥΣΙΚΟΥ ΠΕΡΙΒΑΛΛΟΝΤΟΣ ΤΜΗΜΑ ΓΕΩΠΟΝΙΑΣ ΤΟΜΕΑΣ ΑΓΡΟΤΙΚΗΣ ΟΙΚΟΝΟΜΙΑΣ ΙΩΑΝΝΗ ΑΘ. ΠΑΠΑΪΩΑΝΝΟΥ Πτυχιούχου Γεωπόνου Κατόχου Μεταπτυχιακού
SABiosciences PCR Array Catalog #: PAHS-021 SA+ SCF 4h stimulaton AVG(Ct) Position Unigene Refseq Symbol Description shcontr shcontr-4h shgskβ A01
 SABiosciences PCR Array Catalog #: PAHS-021 SA+ SCF 4h stimulaton AVG(Ct) Position Unigene Refseq Symbol Description shcontr shcontr-4h shgskβ A01 Hs.1274 NM_006129 BMP1 Bone morphogenetic protein 1 25.25
SABiosciences PCR Array Catalog #: PAHS-021 SA+ SCF 4h stimulaton AVG(Ct) Position Unigene Refseq Symbol Description shcontr shcontr-4h shgskβ A01 Hs.1274 NM_006129 BMP1 Bone morphogenetic protein 1 25.25
Fixed Inductors / AL TYPE
 .Features: 1.Coating epoxy resin that ensures the humidity resistance to be long life. 2.Contribute to be high Q and selfresonant frequencies.applications: 1.Electronics products. 2.Communication equipment.
.Features: 1.Coating epoxy resin that ensures the humidity resistance to be long life. 2.Contribute to be high Q and selfresonant frequencies.applications: 1.Electronics products. 2.Communication equipment.
Manuscript submitted to the Journal of the American Society for Mass Spectrometry, September 2011.
 The Early Life of a Peptide Cation-Radical. Ground and Excited-State Trajectories of Electron-Based Peptide Dissociations During the First 330 Femtoseconds Christopher L. Moss, Wenkel Liang, Xiaosong Li,*
The Early Life of a Peptide Cation-Radical. Ground and Excited-State Trajectories of Electron-Based Peptide Dissociations During the First 330 Femtoseconds Christopher L. Moss, Wenkel Liang, Xiaosong Li,*
ΚΥΠΡΙΑΚΗ ΕΤΑΙΡΕΙΑ ΠΛΗΡΟΦΟΡΙΚΗΣ CYPRUS COMPUTER SOCIETY ΠΑΓΚΥΠΡΙΟΣ ΜΑΘΗΤΙΚΟΣ ΔΙΑΓΩΝΙΣΜΟΣ ΠΛΗΡΟΦΟΡΙΚΗΣ 19/5/2007
 Οδηγίες: Να απαντηθούν όλες οι ερωτήσεις. Αν κάπου κάνετε κάποιες υποθέσεις να αναφερθούν στη σχετική ερώτηση. Όλα τα αρχεία που αναφέρονται στα προβλήματα βρίσκονται στον ίδιο φάκελο με το εκτελέσιμο
Οδηγίες: Να απαντηθούν όλες οι ερωτήσεις. Αν κάπου κάνετε κάποιες υποθέσεις να αναφερθούν στη σχετική ερώτηση. Όλα τα αρχεία που αναφέρονται στα προβλήματα βρίσκονται στον ίδιο φάκελο με το εκτελέσιμο
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΤΜΗΜΑ ΟΔΟΝΤΙΑΤΡΙΚΗΣ ΕΡΓΑΣΤΗΡΙΟ ΟΔΟΝΤΙΚΗΣ ΚΑΙ ΑΝΩΤΕΡΑΣ ΠΡΟΣΘΕΤΙΚΗΣ
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΤΜΗΜΑ ΟΔΟΝΤΙΑΤΡΙΚΗΣ ΕΡΓΑΣΤΗΡΙΟ ΟΔΟΝΤΙΚΗΣ ΚΑΙ ΑΝΩΤΕΡΑΣ ΠΡΟΣΘΕΤΙΚΗΣ ΣΥΓΚΡΙΤΙΚΗ ΜΕΛΕΤΗ ΤΗΣ ΣΥΓΚΡΑΤΗΤΙΚΗΣ ΙΚΑΝΟΤΗΤΑΣ ΟΡΙΣΜΕΝΩΝ ΠΡΟΚΑΤΑΣΚΕΥΑΣΜΕΝΩΝ ΣΥΝΔΕΣΜΩΝ ΑΚΡΙΒΕΙΑΣ
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΤΜΗΜΑ ΟΔΟΝΤΙΑΤΡΙΚΗΣ ΕΡΓΑΣΤΗΡΙΟ ΟΔΟΝΤΙΚΗΣ ΚΑΙ ΑΝΩΤΕΡΑΣ ΠΡΟΣΘΕΤΙΚΗΣ ΣΥΓΚΡΙΤΙΚΗ ΜΕΛΕΤΗ ΤΗΣ ΣΥΓΚΡΑΤΗΤΙΚΗΣ ΙΚΑΝΟΤΗΤΑΣ ΟΡΙΣΜΕΝΩΝ ΠΡΟΚΑΤΑΣΚΕΥΑΣΜΕΝΩΝ ΣΥΝΔΕΣΜΩΝ ΑΚΡΙΒΕΙΑΣ
Supplementary Table 1. Construct List with key Biophysical Properties of the expression
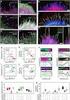 SPINE Benchmark Target ID Well Code Tag (N or C) Fusion MW (kda) Fusion pi Cleavage site Prot MW (Da) Prot pi 1 A1 OPPF 2585 N-His6 21.75 6.41 Protease 3C 19.77 5.43 2 B1 OPPF 2586 N-His6 15.6 6.27 Protease
SPINE Benchmark Target ID Well Code Tag (N or C) Fusion MW (kda) Fusion pi Cleavage site Prot MW (Da) Prot pi 1 A1 OPPF 2585 N-His6 21.75 6.41 Protease 3C 19.77 5.43 2 B1 OPPF 2586 N-His6 15.6 6.27 Protease
ΜΕΛΕΤΗ ΤΗΣ ΗΛΕΚΤΡΟΝΙΚΗΣ ΣΥΝΤΑΓΟΓΡΑΦΗΣΗΣ ΚΑΙ Η ΔΙΕΡΕΥΝΗΣΗ ΤΗΣ ΕΦΑΡΜΟΓΗΣ ΤΗΣ ΣΤΗΝ ΕΛΛΑΔΑ: Ο.Α.Ε.Ε. ΠΕΡΙΦΕΡΕΙΑ ΠΕΛΟΠΟΝΝΗΣΟΥ ΚΑΣΚΑΦΕΤΟΥ ΣΩΤΗΡΙΑ
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΕΙΡΑΙΩΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΔΙΟΙΚΗΣΗ ΤΗΣ ΥΓΕΙΑΣ ΤΕΙ ΠΕΙΡΑΙΑ ΜΕΛΕΤΗ ΤΗΣ ΗΛΕΚΤΡΟΝΙΚΗΣ ΣΥΝΤΑΓΟΓΡΑΦΗΣΗΣ ΚΑΙ Η ΔΙΕΡΕΥΝΗΣΗ ΤΗΣ ΕΦΑΡΜΟΓΗΣ ΤΗΣ ΣΤΗΝ ΕΛΛΑΔΑ: Ο.Α.Ε.Ε. ΠΕΡΙΦΕΡΕΙΑ ΠΕΛΟΠΟΝΝΗΣΟΥ
ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΕΙΡΑΙΩΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΔΙΟΙΚΗΣΗ ΤΗΣ ΥΓΕΙΑΣ ΤΕΙ ΠΕΙΡΑΙΑ ΜΕΛΕΤΗ ΤΗΣ ΗΛΕΚΤΡΟΝΙΚΗΣ ΣΥΝΤΑΓΟΓΡΑΦΗΣΗΣ ΚΑΙ Η ΔΙΕΡΕΥΝΗΣΗ ΤΗΣ ΕΦΑΡΜΟΓΗΣ ΤΗΣ ΣΤΗΝ ΕΛΛΑΔΑ: Ο.Α.Ε.Ε. ΠΕΡΙΦΕΡΕΙΑ ΠΕΛΟΠΟΝΝΗΣΟΥ
Hvordan påvirker CO 2 post-smolt i brakkvann RAS?
 Hvordan påvirker CO 2 post-smolt i brakkvann RAS? Tom O. Nilsen,. Mota, V.C., Kolarevic, J., Ytterborg, E., Ebbesson, Lars O.E., Handeland, S.O., Stiller, K., Terjesen, B.F SUNNDALSØRA, 12. NOVEMBER 2018
Hvordan påvirker CO 2 post-smolt i brakkvann RAS? Tom O. Nilsen,. Mota, V.C., Kolarevic, J., Ytterborg, E., Ebbesson, Lars O.E., Handeland, S.O., Stiller, K., Terjesen, B.F SUNNDALSØRA, 12. NOVEMBER 2018
I ΙΑΙΤΕΡΟΤΗΤΕΣ ΤΗΣ ΙΑΠΙΣΤΕΥΣΗΣ ΤΩΝ ΜΙΚΡΟΒΙΟΛΟΓΙΚΩΝ ΕΡΓΑΣΤΗΡΙΩΝ ΝΕΡΟΥ ΚΑΤΑ ISO 17025. ΠΗΓΕΣ ΑΝΟΜΟΙΟΓΕΝΕΙΑΣ ΚΑΙ ΛΑΘΩΝ
 I ΙΑΙΤΕΡΟΤΗΤΕΣ ΤΗΣ ΙΑΠΙΣΤΕΥΣΗΣ ΤΩΝ ΜΙΚΡΟΒΙΟΛΟΓΙΚΩΝ ΕΡΓΑΣΤΗΡΙΩΝ ΝΕΡΟΥ ΚΑΤΑ ISO 17025. Ρ. Αθηνά Μαυρίδου Καθ. Μικροβιολογίας ΤΕΙ Αθήνας ΠΗΓΕΣ ΑΝΟΜΟΙΟΓΕΝΕΙΑΣ ΚΑΙ ΛΑΘΩΝ Φυσική ανοµοιογένεια νερού στην προέλευση
I ΙΑΙΤΕΡΟΤΗΤΕΣ ΤΗΣ ΙΑΠΙΣΤΕΥΣΗΣ ΤΩΝ ΜΙΚΡΟΒΙΟΛΟΓΙΚΩΝ ΕΡΓΑΣΤΗΡΙΩΝ ΝΕΡΟΥ ΚΑΤΑ ISO 17025. Ρ. Αθηνά Μαυρίδου Καθ. Μικροβιολογίας ΤΕΙ Αθήνας ΠΗΓΕΣ ΑΝΟΜΟΙΟΓΕΝΕΙΑΣ ΚΑΙ ΛΑΘΩΝ Φυσική ανοµοιογένεια νερού στην προέλευση
Structure-Metabolism-Relationships in the microsomal clearance of. piperazin-1-ylpyridazines
 Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2017 Structure-Metabolism-Relationships in the microsomal clearance of piperazin-1-ylpyridazines
Electronic Supplementary Material (ESI) for MedChemComm. This journal is The Royal Society of Chemistry 2017 Structure-Metabolism-Relationships in the microsomal clearance of piperazin-1-ylpyridazines
ΟΒΑΡΗ ΔΙΕΓΧΕΙΡΗΣΙΚΗ ΑΙΜΟΡΡΑΓΙΑ- ΑΙΜΟΡΡΑΓΙΚΗ ΚΑΣΑΠΛΗΞΙΑ. Βαςιλειάδθσ Μιχάλθσ Επιμ.Β ΜΕΘ «Θεαγζνειου» Αντικαρκινικοφ Νοςοκομείου Θεςςαλονίκθσ
 ΟΒΑΡΗ ΔΙΕΓΧΕΙΡΗΣΙΚΗ ΑΙΜΟΡΡΑΓΙΑ- ΑΙΜΟΡΡΑΓΙΚΗ ΚΑΣΑΠΛΗΞΙΑ Βαςιλειάδθσ Μιχάλθσ Επιμ.Β ΜΕΘ «Θεαγζνειου» Αντικαρκινικοφ Νοςοκομείου Θεςςαλονίκθσ Σι ζπεται; 1. ΑΙΣΙΑ-ΔΙΑΓΝΩΗ ΑΙΜΟΡΡΑΓΙΑ/ΚΑΣΑΠΛΗΞΙΑ 2. ΠΑΘΟΦΤΙΟΛΟΓΙΑ
ΟΒΑΡΗ ΔΙΕΓΧΕΙΡΗΣΙΚΗ ΑΙΜΟΡΡΑΓΙΑ- ΑΙΜΟΡΡΑΓΙΚΗ ΚΑΣΑΠΛΗΞΙΑ Βαςιλειάδθσ Μιχάλθσ Επιμ.Β ΜΕΘ «Θεαγζνειου» Αντικαρκινικοφ Νοςοκομείου Θεςςαλονίκθσ Σι ζπεται; 1. ΑΙΣΙΑ-ΔΙΑΓΝΩΗ ΑΙΜΟΡΡΑΓΙΑ/ΚΑΣΑΠΛΗΞΙΑ 2. ΠΑΘΟΦΤΙΟΛΟΓΙΑ
Νανοσύνθετα πολυαιθυλενίου υψηλής πυκνότητας (HDPE) / νανοϊνών χαλκού (Cu-nanofibers) με βελτιωμένη σταθερότητα στην υπεριώδη ακτινοβολία
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΘΕΤΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΤΜΗΜΑ ΦΥΣΙΚΗΣ Πρόγραμμα Μεταπτυχιακών Σπουδών ΦΥΣΙΚΗΣ ΚΑΙ ΤΕΧΝΟΛΟΓΙΑΣ ΥΛΙΚΩΝ Νανοσύνθετα πολυαιθυλενίου υψηλής πυκνότητας (HDPE) / νανοϊνών
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΘΕΤΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΤΜΗΜΑ ΦΥΣΙΚΗΣ Πρόγραμμα Μεταπτυχιακών Σπουδών ΦΥΣΙΚΗΣ ΚΑΙ ΤΕΧΝΟΛΟΓΙΑΣ ΥΛΙΚΩΝ Νανοσύνθετα πολυαιθυλενίου υψηλής πυκνότητας (HDPE) / νανοϊνών
Μελέτη της επίδρασης αντικαρκινικών χημειοθεραπευτικών φαρμάκων στη ρύθμιση του Cdt1
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ Πρόγραμμα Μεταπτυχιακών Σπουδών Εφαρμογές Στις Βασικές Επιστήμες Κατεύθυνση: Φαρμακοκινητική Τοξικολογία Διπλωματική Εργασία Μελέτη της επίδρασης
ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ Πρόγραμμα Μεταπτυχιακών Σπουδών Εφαρμογές Στις Βασικές Επιστήμες Κατεύθυνση: Φαρμακοκινητική Τοξικολογία Διπλωματική Εργασία Μελέτη της επίδρασης
BioVision Catalog Description Pack Size Supplier Price (RS)
 Catalog Description Pack Size Supplier Price (RS) K101-100 Annexin V-FITC Apoptosis Kit 100 assays BioVision 60991 K101-25 Annexin V-FITC Apoptosis Kit 25 assays BioVision 28373 K101-400 Annexin V-FITC
Catalog Description Pack Size Supplier Price (RS) K101-100 Annexin V-FITC Apoptosis Kit 100 assays BioVision 60991 K101-25 Annexin V-FITC Apoptosis Kit 25 assays BioVision 28373 K101-400 Annexin V-FITC
ΠΕΡΙΕΧΟΜΕΝΑ. Κεφάλαιο 1: Κεφάλαιο 2: Κεφάλαιο 3:
 4 Πρόλογος Η παρούσα διπλωµατική εργασία µε τίτλο «ιερεύνηση χωρικής κατανοµής µετεωρολογικών µεταβλητών. Εφαρµογή στον ελληνικό χώρο», ανατέθηκε από το ιεπιστηµονικό ιατµηµατικό Πρόγραµµα Μεταπτυχιακών
4 Πρόλογος Η παρούσα διπλωµατική εργασία µε τίτλο «ιερεύνηση χωρικής κατανοµής µετεωρολογικών µεταβλητών. Εφαρµογή στον ελληνικό χώρο», ανατέθηκε από το ιεπιστηµονικό ιατµηµατικό Πρόγραµµα Μεταπτυχιακών
 22 .5 Real consumption.5 Real residential investment.5.5.5 965 975 985 995 25.5 965 975 985 995 25.5 Real house prices.5 Real fixed investment.5.5.5 965 975 985 995 25.5 965 975 985 995 25.3 Inflation
22 .5 Real consumption.5 Real residential investment.5.5.5 965 975 985 995 25.5 965 975 985 995 25.5 Real house prices.5 Real fixed investment.5.5.5 965 975 985 995 25.5 965 975 985 995 25.3 Inflation
Πρόσκληση υποβολής προσφοράς
 Αθήνα, 5 Μαρτίου 2014 Πρόσκληση υποβολής προσφοράς από φυσικά ή νομικά πρόσωπα για την προμήθεια υλικών στην Πράξη «ΘΑΛΗΣ Εθνικό Ίδρυμα Ερευνών Βιοσύνθεση και Γενετική Επιλογή Κυκλικών Πεπτιδίων με Εν
Αθήνα, 5 Μαρτίου 2014 Πρόσκληση υποβολής προσφοράς από φυσικά ή νομικά πρόσωπα για την προμήθεια υλικών στην Πράξη «ΘΑΛΗΣ Εθνικό Ίδρυμα Ερευνών Βιοσύνθεση και Γενετική Επιλογή Κυκλικών Πεπτιδίων με Εν
Supplementary Table 1. Primer sequences used for RT-qPCR validation of the microarray data. Primer. direction. Forward
 Supplementary Table 1. Primer sequences used for RT-qPCR validation of the microarray data Gene Glyceraldehyde 3-phosphate dehydrogenase (Gapdh) Cell death-inducing DFFAlike effector a (Cidea) Peroxisome
Supplementary Table 1. Primer sequences used for RT-qPCR validation of the microarray data Gene Glyceraldehyde 3-phosphate dehydrogenase (Gapdh) Cell death-inducing DFFAlike effector a (Cidea) Peroxisome
VBA Microsoft Excel. J. Comput. Chem. Jpn., Vol. 5, No. 1, pp (2006)
 J. Comput. Chem. Jpn., Vol. 5, No. 1, pp. 29 38 (2006) Microsoft Excel, 184-8588 2-24-16 e-mail: yosimura@cc.tuat.ac.jp (Received: July 28, 2005; Accepted for publication: October 24, 2005; Published on
J. Comput. Chem. Jpn., Vol. 5, No. 1, pp. 29 38 (2006) Microsoft Excel, 184-8588 2-24-16 e-mail: yosimura@cc.tuat.ac.jp (Received: July 28, 2005; Accepted for publication: October 24, 2005; Published on
ST5224: Advanced Statistical Theory II
 ST5224: Advanced Statistical Theory II 2014/2015: Semester II Tutorial 7 1. Let X be a sample from a population P and consider testing hypotheses H 0 : P = P 0 versus H 1 : P = P 1, where P j is a known
ST5224: Advanced Statistical Theory II 2014/2015: Semester II Tutorial 7 1. Let X be a sample from a population P and consider testing hypotheses H 0 : P = P 0 versus H 1 : P = P 1, where P j is a known
4.6 Autoregressive Moving Average Model ARMA(1,1)
 84 CHAPTER 4. STATIONARY TS MODELS 4.6 Autoregressive Moving Average Model ARMA(,) This section is an introduction to a wide class of models ARMA(p,q) which we will consider in more detail later in this
84 CHAPTER 4. STATIONARY TS MODELS 4.6 Autoregressive Moving Average Model ARMA(,) This section is an introduction to a wide class of models ARMA(p,q) which we will consider in more detail later in this
Other Test Constructions: Likelihood Ratio & Bayes Tests
 Other Test Constructions: Likelihood Ratio & Bayes Tests Side-Note: So far we have seen a few approaches for creating tests such as Neyman-Pearson Lemma ( most powerful tests of H 0 : θ = θ 0 vs H 1 :
Other Test Constructions: Likelihood Ratio & Bayes Tests Side-Note: So far we have seen a few approaches for creating tests such as Neyman-Pearson Lemma ( most powerful tests of H 0 : θ = θ 0 vs H 1 :
περιβάλλονται από δύο µεµβράνες
 Οργανίδια οργανίδια και ενέργεια µιτοχόνδρια χλωροπλάστες οργανίδια και γενετική πληροφορία πυρήνας Τα µιτοχόνδρια, οι χλωροπλάστες και ο πυρήνας αν και περιβάλλονται από µεµβράνη δεν αποτελούν µέρος του
Οργανίδια οργανίδια και ενέργεια µιτοχόνδρια χλωροπλάστες οργανίδια και γενετική πληροφορία πυρήνας Τα µιτοχόνδρια, οι χλωροπλάστες και ο πυρήνας αν και περιβάλλονται από µεµβράνη δεν αποτελούν µέρος του
Bayesian statistics. DS GA 1002 Probability and Statistics for Data Science.
 Bayesian statistics DS GA 1002 Probability and Statistics for Data Science http://www.cims.nyu.edu/~cfgranda/pages/dsga1002_fall17 Carlos Fernandez-Granda Frequentist vs Bayesian statistics In frequentist
Bayesian statistics DS GA 1002 Probability and Statistics for Data Science http://www.cims.nyu.edu/~cfgranda/pages/dsga1002_fall17 Carlos Fernandez-Granda Frequentist vs Bayesian statistics In frequentist
Dietary success of a new key fish in an overfished ecosystem: evidence from fatty acid and stable isotope signatures
 The following supplement accompanies the article Dietary success of a new key fish in an overfished ecosystem: evidence from fatty acid and stable isotope signatures M. G. van der Bank 1, A. C. Utne-Palm
The following supplement accompanies the article Dietary success of a new key fish in an overfished ecosystem: evidence from fatty acid and stable isotope signatures M. G. van der Bank 1, A. C. Utne-Palm
