Legends for Supplemental Data
|
|
|
- Πάρις Μεταξάς
- 6 χρόνια πριν
- Προβολές:
Transcript
1 Legends for Supplemental Data Supplemental Fig. 1. Top molecular and cellular functions identified by Ingenuity Pathway Analysis after 6 h (left) and 24 h (right) of exposure of mouse pancreatic islets to IL-1β and IFN-γ (p<0.05 by Fischer s exact test). Supplemental Fig. 2. Canonical pathways identified by Ingenuity Pathway Analysis after 6 h (left) and 24 h (right) of exposure of mouse pancreatic islets to IL-1β and IFN-γ (p<0.05 by Fischer s exact test). Supplemental Fig. 3. Top molecular and cellular functions identified by Ingenuity Pathway Analysis after 6 h (left) and 24 h (right) of co-treatment (1,25(OH) 2 D 3 in combination with IL-1β and IFN-γ) as compared to cytokine exposure alone (IL-1β and IFN-γ) of mouse pancreatic islets (p<0.05 by Fischer s exact test). Supplemental Fig. 4. Effect of 1,25(OH) 2 D 3 on qrt-pcr mrna expression of chemokines and cytokines in IL-1β- and IFN-γ-treated human islets. Islets were cultured for 24 h with ) control medium; ) 10-8 M 1,25(OH) 2 D 3 ; ) 50 U/ml IL-1β and 100 U/ml IFN-γ; and ) 10-8 M 1,25(OH) 2 D 3, 50 U/ml IL-1β and 100 U/ml IFN-γ. Data are presented as three individual islet isolations (HI 1, HI 2 and HI 3). Supplemental Table 1. Manual classification of selected genes differentially regulated by IL-1β and IFN-γ vs vehicle in islets after 6 and 24 h of in vitro exposure (n=4 independent experiments expressed as fold change vs respective control. *p<0.05; **p<0.02; ***p<0.01; ****p<0.001 vs vehicle by Welch s t-test. Con: Control. 1,25: 1,25(OH) 2 D 3. Cyt: IL-1β and IFN-γ). Supplemental Table 2. Manual classification of selected genes differentially regulated by cotreatment (1,25(OH) 2 D 3 in combination with IL-1β and IFN-γ) as compared to cytokine exposure alone (IL-1β and IFN-γ) in pancreatic islets (n=4 independent experiments expressed as fold change vs respective control. *p<0.05; **p<0.02; ***p<0.01; ****p<0.001 Welch s t-test. Con: Control. 1,25: 1,25(OH) 2 D 3. Cyt: IL-1β and IFN-γ).
2 1 Supplemental materials and methods Chemotaxis assay C57BL/6 islets were cultured in BioWhittaker UltraCULTURE Serum-Free Medium (Lonza, Basel, Switzerland) supplemented with Glutamax -I (Life Technologies, Carlsbad, CA) and 5 µg/ml geneticin. Exposure to 1,25(OH) 2 D 3 and cytokines was similar to that described in section 2.2. except for a prolonged culture period of 3 days. For the chemotaxis assay thioglycolate-elicited peritoneal cells from 8-12 week old C57BL/6 mice were seeded in the top chamber of Costar transwell plates ( cells per well) (5.0 µm, Corning Inc., Corning, NY), while conditioned islet medium was added to the lower chambers of the transwell plate. Positive and negative controls consisted of UltraCULTURE Serum-Free medium with or without recombinant murine MIP-1α (50ng/ml, PeproTech), respectively. Following an incubation time of 2 h at 37 C in 5% CO 2, the cells in the lower chamber were harvested and stained for monocyte specific cell surface markers. To minimize nonspecific binding, the cells were first pre-incubated with anti-cd16/cd32 (clone 2.4G2). Cells were then labeled with directly conjugated mabs: CD45, CD11b, Ly6C, F4/80 (San Diego, CA, USA). Dead cells were excluded by using the Fixable Live/Dead Yellow stain according to the manufacturer s specifications (Invitrogen, Carlsbad, CA). The absolute number of migrated monocytes was determined by CountBright absolute counting beads (Invitrogen). Data acquisition was performed on a Gallios TM flow cytometer (Beckman Coulter, Brea, CA, USA) and KaluzaTM software was used for data analysis. Monocytes were determined as Ly6C + CD11b + F4/80 - within the viable leukocyte (CD45 + ) gate Assessment of NF-κB activity Islets were cultured for 24 h in the presence of 1,25(OH) 2 D 3 or vehicle, after which they were stimulated for 30 min with IL-1β and IFN-γ at 37 C. Thereafter, islets were washed twice in ice-cold PBS supplemented with 1:100 Phosphatase Inhibitor Cocktail 2 (Sigma-Aldrich, St. Louis, MO) and lysed in Complete lysis buffer (Active Motif, Carlsbad, CA). Protein concentrations were determined
3 28 29 by the Coomassie method. Binding activity of NF-κB p52 subunits was determined by the TransAM NF-κB assay according to the manufacturer s instructions (Active Motif).
4
5
6
7
8 6 h 24 h Cyt Cyt Accession Symbol Gene name / functional group vs Con vs Con Arginine metabolism, Nitric oxide formation NM_ Arg1 arginase, liver **** **** NM_ Nos2 (inos) nitric oxide synthase 2, inducible **** **** Glucose metabolism, Krebs cycle NM_ Aco1 aconitase **** **** NM_ Aco2 aconitase 2, mitochondrial **** *** NM_ Aldh2 aldehyde dehydrogenase 2, mitochondrial *** *** NM_ Cs citrate synthase * NM_ Eno1 enolase 1, alpha non-neuron **** 1.38 *** NM_ Fh1 fumarate hydratase **** **** NM_ Gck glucokinase *** NM_ Glud1 glutamate dehydrogenase **** 1.01 NM_ Gpam glycerol-3-phosphate acyltransferase, mitochondrial *** 1.06 NM_ Gpd1 glycerol-3-phosphate dehydrogenase 1 (soluble) **** 1.07 NM_ Gpi1 glucose phosphate isomerase ** 1.3 ** NM_ Hk2 hexokinase **** 2.78 *** NM_ Idh1 isocitrate dehydrogenase 1 (NADP+), soluble **** **** NM_ Idh2 isocitrate dehydrogenase 2 (NADP+), mitochondrial **** 1.45 *** NM_ Idh3b isocitrate dehydrogenase 3 (NAD+), beta **** 1.14 ** NM_ Idh3g isocitrate dehydrogenase 3 (NAD+), gamma * **** NM_ Ldha lactate dehydrogenase A ** NM_ Ldhd lactate dehydrogenase D 1.38 *** NM_ Mdh1 malate dehydrogenase 1, NAD (soluble) **** -1.4 **** NM_ Mdh2 malate dehydrogenase 2, NAD (mitochondrial) * ** NM_ Ogdh oxoglutarate dehydrogenase (lipoamide) *** **** NM_ P4hb prolyl 4-hydroxylase, beta polypeptide **** * NM_ Pcx pyruvate carboxylase *** *** NM_ Pdha1 pyruvate dehydrogenase E1 alpha *** **** NM_ Pdhb pyruvate dehydrogenase (lipoamide) beta **** NM_ Pdk1 pyruvate dehydrogenase kinase, isoenzyme *** NM_ Pdk2 pyruvate dehydrogenase kinase, isoenzyme **** *** NM_ Pdk3 pyruvate dehydrogenase kinase, isoenzyme *** 1.54 ** NM_ Pdk4 pyruvate dehydrogenase kinase, isoenzyme **** **** NM_ Pfkfb2 6-phosphofructo-2-kinase/fructose-2,6-biphosphatase *** *** NM_ Pfkfb3 6-phosphofructo-2-kinase/fructose-2,6-biphosphatase 3 (glycolytic 9.8 **** 4.08 **** activator) NM_ Pfkl phosphofructokinase, liver, B-type **** 1.46 * NM_ Pfkm phosphofructokinase, muscle **** -2 **** NM_ Pfkp phosphofructokinase, platelet 6 **** 2.67 *** NM_ Pgam5 phosphoglycerate mutase family member *** 1.54 *** NM_ Pklr pyruvate kinase liver and red blood cell *** *** NM_ Pkm2 pyruvate kinase, muscle *** 1.03 NM_ Sdhb succinate dehydrogenase complex, subunit B, iron sulfur (Ip) * 1.06 NM_ Slc2a1 solute carrier family 2 (facilitated glucose transporter), member *** 4.92 *** NM_ Slc2a2 (Glut-2) solute carrier family 2 (facilitated glucose transporter), member **** **** NM_ Suclg1 succinate-coa ligase, GDP-forming, alpha subunit NM_ Tpi1 triosephosphate isomerase **** 1.38 ** Lipid metabolism NM_ Acaca acetyl-coenzyme A carboxylase alpha *** **** NM_ Acat1 acetyl-coenzyme A acetyltransferase **** **** NM_ Acly ATP citrate lyase **** **** NM_ Acsl1 acyl-coa synthetase long-chain family member *** NM_ Acsl3 acyl-coa synthetase long-chain family member *** 1.3 **** NM_ Acsl5 acyl-coa synthetase long-chain family member **** 1.52 **** NM_ Ckb creatine kinase, brain * **** NM_ Ckmt1 creatine kinase, mitochondrial 1, ubiquitous -1.2 * 1.74 * NM_ Cpt1a carnitine palmitoyltransferase 1a, liver 1.14 ** 2.14 **** NM_ Cpt2 carnitine palmitoyltransferase NM_ Dagla diacylglycerol lipase, alpha **** -1.7 * NM_ Dgkg diacylglycerol kinase, gamma 7.43 **** 2.08 *
9 NM_ Elovl2 elongation of very long chain fatty acids (FEN1/Elo2, SUR4/Elo3, *** **** yeast)-like 2 NM_ Elovl6 ELOVL family member 6, elongation of long chain fatty acids 1.18 ** ** (yeast) NM_ Fads1 fatty acid desaturase **** **** NM_ Fads2 fatty acid desaturase **** * NM_ Fasn fatty acid synthase **** **** NM_ Gyk glycerol kinase ** **** NM_ Lipc lipase, hepatic 1.51 **** 1.65 **** NM_ Lpl lipoprotein lipase *** ** NM_ Srebf2 sterol regulatory element binding factor * * NM_ Vldlr very low density lipoprotein receptor Interferon-γ signaling NM_ Ifnar2 interferon (alpha and beta) receptor **** 2.52 **** NM_ Ifngr1 interferon gamma receptor NM_ Ifngr2 interferon gamma receptor **** 3.03 **** NM_ Irf1 interferon regulatory factor **** 9.97 **** NM_ Irf1 interferon regulatory factor **** 1.64 **** NM_ Irf2 interferon regulatory factor **** 1.47 **** NM_ Irf3 interferon regulatory factor NM_ Irf7 interferon regulatory factor **** **** NM_ Irf8 interferon regulatory factor **** 2.29 **** NM_ Irf9 interferon regulatory factor **** 5.79 **** NM_ Jak1 Janus kinase **** 1.49 **** NM_ Jak2 Janus kinase **** 2.02 *** NM_ Jak3 Janus kinase **** 1.52 *** NM_ Socs1 suppressor of cytokine signaling **** 1.48 **** NM_ Socs2 suppressor of cytokine signaling U72673 Socs3 suppressor of cytokine signaling * 1.04 NM_ Socs3 suppressor of cytokine signaling **** 1.41 ** NM_ Socs6 suppressor of cytokine signaling * 1.47 * NM_ Socs7 suppressor of cytokine signaling *** NM_ Stat1 signal transducer and activator of transcription **** **** NM_ Stat2 signal transducer and activator of transcription **** 7.85 **** NM_ Stat3 signal transducer and activator of transcription **** 2.1 *** NM_ Stat5b signal transducer and activator of transcription 5B ** NM_ Stat6 signal transducer and activator of transcription **** 1.63 *** NF-κB regulation NM_ Ikbke inhibitor of kappab kinase epsilon 9.22 **** 2.99 **** NM_ Ikbkg inhibitor of kappab kinase gamma 1.38 *** 2.14 **** NM_ Irak2 interleukin-1 receptor-associated kinase **** 2.43 **** NM_ Irak3 interleukin-1 receptor-associated kinase **** 1.92 **** NM_ Nfkb1 nuclear factor of kappa light polypeptide gene enhancer in B-cells 1, 3.36 **** 1.29 ** p105 NM_ Nfkb2 nuclear factor of kappa light polypeptide gene enhancer in B-cells 2, **** 3.86 **** p49/p100 NM_ Nfkbia nuclear factor of kappa light polypeptide gene enhancer in B-cells 10.2 **** 5.43 **** inhibitor, alpha NM_ Nfkbib nuclear factor of kappa light polypeptide gene enhancer in B-cells 2.7 **** 1.95 **** inhibitor, beta NM_ Nfkbie nuclear factor of kappa light polypeptide gene enhancer in B-cells 3.26 **** 1.49 * inhibitor, epsilon NM_ Nfkbiz nuclear factor of kappa light polypeptide gene enhancer in B-cells 4.61 **** 2.69 **** inhibitor, zeta NM_ Tank TRAF family member-associated Nf-kappa B activator 1.91 **** 1.13 ** NM_ Tbk1 TANK-binding kinase **** 1.64 *** NM_ Tifa TRAF-interacting protein with forkhead-associated domain **** 7.59 **** NM_ Tnfaip3 (A20) tumor necrosis factor, alpha-induced protein **** 3.32 **** NM_ Tnip1 TNFAIP3 interacting protein 1 (obs candidate gene in psoriarisis) 6.72 **** 4.97 **** NM_ Tnip2 TNFAIP3 interacting protein *** 1.5 **** NM_ Traf2 TNF receptor-associated factor **** 1.66 ** Other transcription factors NM_ Batf2 basic leucine zipper transcription factor, ATF-like **** 3.39 **** NM_ Bcl10 B-cell leukemia/lymphoma *** 1.32 ***
10 NM_ Bcl3 B-cell leukemia/lymphoma **** 3.96 **** NM_ Bcl6 B-cell leukemia/lymphoma **** 4.2 *** NM_ Bcor BCL6 interacting corepressor *** NM_ Bmp2 bone morphogenetic protein **** NM_ Cebpb CCAAT/enhancer binding protein (C/EBP), beta 2.79 **** 1.88 **** NM_ Cebpd CCAAT/enhancer binding protein (C/EBP), delta **** 5 **** NM_ Cebpg CCAAT/enhancer binding protein (C/EBP), gamma 1.62 **** 1.63 *** NM_ Dll1 delta-like 1 (Drosophila) 3.99 **** 1.98 *** NM_ Fos FBJ osteosarcoma oncogene NM_ Foxo1 forkhead box O1-1.6 **** **** NM_ Foxo4 forkhead box O **** **** NM_ Hes6 hairy and enhancer of split 6 (Drosophila) **** *** NM_ Hif1a hypoxia inducible factor 1, alpha subunit 2.27 **** 1.24 * NM_ Ifitm1 interferon induced transmembrane protein **** 1.48 **** NM_ Isl1 ISL1 transcription factor, LIM/homeodomain **** **** NM_ Jag2 jagged *** **** NM_ Jun (c-jun) Jun oncogene 5.86 **** 4.13 *** NM_ Junb Jun-B oncogene 2.72 **** 1.9 *** NM_ Jund Jun proto-oncogene related gene d 1.53 *** 1.4 *** NM_ Klf5 Kruppel-like factor **** 1.51 *** NM_ Klf6 Kruppel-like factor **** 2.62 *** NM_ Neurod1 neurogenic differentiation *** NM_ Neurog3 neurogenin **** *** NM_ Nkx2-2 NK2 transcription factor related, locus 2 (Drosophila) **** **** NM_ Nkx6-1 NK6 homeobox **** -1.8 *** NM_ Pax6 paired box gene **** **** NM_ Pdx1 pancreatic and duodenal homeobox **** **** NM_ Ppard peroxisome proliferator activator receptor delta 1.55 **** 1.12 NM_ Sec16a SEC16 homolog A (S. cerevisiae) *** NM_ Srebf1 sterol regulatory element binding transcription factor *** **** NM_ Tcf7l2 transcription factor 7-like 2, T-cell specific, HMG-box 1.31 *** **** NM_ Tnfaip2 tumor necrosis factor, alpha-induced protein **** 5.76 **** NM_ Trib2 tribbles homolog 2 (Drosophila) **** **** NM_ Wif1 Wnt inhibitory factor **** 1.74 *** NM_ Xbp1 X-box binding protein *** *** Splicing machinery, chromatin regulation NM_ Hdac2 histone deacetylase *** NM_ Hdac3 histone deacetylase **** *** NM_ Hdac5 histone deacetylase **** *** NM_ Hdac9 histone deacetylase **** *** NM_ Pcgf2 polycomb group ring finger *** 1.06 BC Pcgf5 polycomb group ring finger *** NM_ Sfrs1 splicing factor, arginine/serine-rich 1 (ASF/SF2) **** NM_ Sfrs17b splicing factor, arginine/serine-rich 17b *** NM_ Sfrs18 splicing factor, arginine/serine-rich *** *** NM_ Sfrs18 splicing factor, arginine/serine-rich **** NM_ Sfrs2 splicing factor, arginine/serine-rich 2 (SC-35) *** NM_ Sfrs2ip splicing factor, arginine/serine-rich 2, interacting protein -1.2 * 1.3 * NM_ Sfrs3 splicing factor, arginine/serine-rich 3 (SRp20) ** NM_ Sfrs4 splicing factor, arginine/serine-rich 4 (SRp75) 1.47 **** 1.48 * NM_ Sfrs6 splicing factor, arginine/serine-rich **** NM_ Sfrs7 splicing factor, arginine/serine-rich * ** Chemokines, cytokines, adhesion molecules NM_ Ccl11 (Eotaxin) chemokine (C-C motif) ligand **** 1.77 NM_ Ccl2 (MCP-1) chemokine (C-C motif) ligand **** **** NM_ Ccl20 chemokine (C-C motif) ligand **** 1.22 * NM_ Ccl28 chemokine (C-C motif) ligand **** 1.23 ** NM_ Ccl5 (RANTES) chemokine (C-C motif) ligand **** 4.21 **** NM_ Ccl7 (MCP-3) chemokine (C-C motif) ligand *** 2.73 *** NM_ Ccl8 (MCP-2) chemokine (C-C motif) ligand **** 3 **** NM_ Ccrl2 chemokine (C-C motif) receptor-like **** 4.92 **** NM_ Csf1 colony stimulating factor 1 (macrophage) 5.13 **** 3.43 *** NM_ Cx3cl1 (fractalkine) chemokine (C-X3-C motif) ligand **** ****
11 NM_ Cxcl1 (Gro-α) chemokine (C-X-C motif) ligand **** 4.06 **** NM_ Cxcl10 (IP-10) chemokine (C-X-C motif) ligand **** **** NM_ Cxcl11 (Itac) chemokine (C-X-C motif) ligand **** **** NM_ Cxcl16 chemokine (C-X-C motif) ligand **** 8.57 **** NM_ Cxcl2 (Gro-beta) chemokine (C-X-C motif) ligand **** 2.78 * NM_ Cxcl5 (ENA-78) chemokine (C-X-C motif) ligand **** **** NM_ Cxcl9 (Mig) chemokine (C-X-C motif) ligand **** **** NM_ Icam1 intercellular adhesion molecule **** **** NM_ Il10rb interleukin 10 receptor, beta 1.84 **** 1.45 **** NM_ Il13ra1 interleukin 13 receptor, alpha **** 3.1 **** NM_ Il15 interleukin **** 1.64 ** NM_ Il15ra interleukin 15 receptor, alpha chain 2 **** 1.66 *** NM_ Il17c interleukin 17C 2.1 *** NM_ Il17rc interleukin 17 receptor C 1.34 *** 1.44 **** NM_ Il17rd interleukin 17 receptor D 1.29 *** 1.28 NM_ Il17re interleukin 17 receptor E -1.5 **** **** NM_ Il18 interleukin **** NM_ Il18bp interleukin 18 binding protein 7.71 **** 9.58 **** NM_ Il18r1 interleukin 18 receptor **** 3.51 **** NM_ Il1a interleukin 1 alpha *** NM_ Il1b interleukin 1 beta 1.74 *** 1.89 **** NM_ Il1r2 interleukin 1 receptor, type II 1.59 *** 1.28 *** NM_ Il1rap interleukin 1 receptor accessory protein 1.82 **** 1.23 *** NM_ Il1rn interleukin 1 receptor antagonist 1.48 *** 1.76 **** NM_ Il20rb interleukin 20 receptor beta 1.49 *** 1 NM_ Il21 interleukin *** 1.28 ** NM_ Il27 interleukin *** 1.22 NM_ Il28b interleukin 28B 1.79 *** 1.5 *** NM_ Il33 interleukin **** 1.28 NM_ Il34 interleukin *** 1.27 ** NM_ Il4ra interleukin 4 receptor, alpha 2 **** 1.65 *** NM_ Il6 interleukin **** **** NM_ Il6ra interleukin 6 receptor, alpha **** **** NM_ Il7 interleukin **** 1.64 **** NM_ Tnf tumor necrosis factor 3.9 **** 1.28 NM_ Vcam1 vascular cell adhesion molecule **** 2.14 **** Other innate immune response components NM_ Cfb complement factor B 59.1 **** **** NM_ Ddx58 (RIG-I) DEAD (Asp-Glu-Ala-Asp) box polypeptide **** 4.34 **** NM_ Ddx60 DEAD (Asp-Glu-Ala-Asp) box polypeptide **** 6.45 **** NM_ Defb1 defensin beta **** 3.06 *** NM_ Hamp hepcidin antimicrobial peptide **** **** NM_ Ido1 indoleamine 2,3-dioxygenase **** 5.74 **** NM_ Ido2 indoleamine 2,3-dioxygenase *** NM_ Ifih1 (MDA5) interferon induced with helicase C domain **** 1.76 **** NM_ Irg1 immunoresponsive gene **** 8.46 **** NM_ Mx1 myxovirus (influenza virus) resistance **** 4.21 **** NM_ Oas1a 2'-5' oligoadenylate synthetase 1A 9.6 **** 4.98 **** NR_ Oas1b 2'-5' oligoadenylate synthetase 1B 7.12 **** 3.92 *** NM_ Oas1c 2'-5' oligoadenylate synthetase 1C 1.43 *** * NM_ Oas2 2'-5' oligoadenylate synthetase **** 11.9 **** NM_ Oas3 2'-5' oligoadenylate synthetase **** 1.52 ** NM_ Oasl1 2'-5' oligoadenylate synthetase-like **** 1.99 **** NM_ Oasl2 2'-5' oligoadenylate synthetase-like **** **** NM_ Tirap toll-interleukin 1 receptor (TIR) domain-containing adaptor protein 1.54 *** 1.03 NM_ Tlr2 toll-like receptor **** 3.05 **** NM_ Tlr3 toll-like receptor **** 1.62 **** Proteasome, antigen presentation NM_ Cd74 CD74 antigen (invariant polypeptide of major histocompatibility **** **** complex, class II antigen-associated) NM_ H2-Ea histocompatibility 2, class II antigen E alpha NM_ H2-M3 histocompatibility 2, M region locus **** 1.62 **** NM_ H2-T23 histocompatibility 2, T region locus **** 6.39 **** NM_ Prr3 proline-rich polypeptide
12 NM_ Psmb8 proteasome (prosome, macropain) subunit, beta type 8 (large 14.3 **** **** multifunctional peptidase 7) NM_ Psme2 proteasome (prosome, macropain) 28 subunit, beta 5.16 **** 5.77 **** NM_ Ring1 ring finger protein *** * NM_ Slc39a7 solute carrier family 39 (zinc transporter), member **** NM_ Tap1 (Mtp-1) transporter 1, ATP-binding cassette, sub-family B (MDR/TAP) **** **** NM_ Tap2 (Mtp-2) transporter 2, ATP-binding cassette, sub-family B (MDR/TAP) 8.41 **** 6.9 **** NM_ Vps52 vacuolar protein sorting 52 (yeast) * (there are also a large number of up-regulated HLA-related molecules) Hormones, growth factors NM_ Adm adrenomedullin ** NM_ Chga chromogranin A *** NM_ Chgb chromogranin B 1.17 * 1.17 NM_ Fgf1 fibroblast growth factor **** ** NM_ Furin furin (paired basic amino acid cleaving enzyme) NM_ Gcg glucagon NM_ Iapp islet amyloid polypeptide NM_ Igf2bp2 insulin-like growth factor 2 mrna binding protein **** 3.34 *** NM_ Ins1 insulin I *** NM_ Ins2 insulin II NM_ Pcsk1 (Pc1) proprotein convertase subtilisin/kexin type * **** NM_ Pcsk2 (Pc2) proprotein convertase subtilisin/kexin type *** **** NM_ Pdgfb platelet derived growth factor, B polypeptide 1.61 *** 1.52 NM_ Sst somatostatin -1.5 **** * NM_ Tgfb1 transforming growth factor, beta ** 1.6 ** NM_ Vegfa vascular endothelial growth factor A **** 1.16 *** NM_ Vegfb vascular endothelial growth factor B *** Hormone and growth factor receptors NM_ Galr1 galanin receptor **** **** NM_ Gcgr glucagon receptor **** ** NM_ Ghr growth hormone receptor **** **** NM_ Gipr gastric inhibitory polypeptide receptor **** *** NM_ Glp1r glucagon-like peptide 1 receptor **** **** NM_ Igf1r insulin-like growth factor I receptor 2.04 **** 1.6 * NM_ Igf2r insulin-like growth factor 2 receptor 1.48 **** 1.06 NM_ Insr insulin receptor -1.4 **** 1.08 NM_ Insr insulin receptor **** 1.02 NM_ Oxtr oxytocin receptor **** **** NM_ Pdgfrb platelet derived growth factor receptor, beta polypeptide *** NM_ Prlhr prolactin releasing hormone receptor **** * BC Prlr prolactin receptor -2.6 **** **** NM_ Prlr prolactin receptor **** *** NM_ Tgfbr1 transforming growth factor, beta receptor I *** NM_ Vdr vitamin D receptor *** *** Free radical scavenger, DNA damage response NM_ Cat catalase 1.23 * 2.88 **** NM_ Ccnd1 cyclin D **** **** NM_ Ccnh cyclin H **** *** NM_ Cdk5 cyclin-dependent kinase **** *** NM_ Cdkl1 cyclin-dependent kinase-like 1 (CDC2-related kinase) **** ** NM_ Cnnm2 cyclin M *** NM_ Gadd45b growth arrest and DNA-damage-inducible 45 beta 6.57 **** **** NM_ Gadd45g growth arrest and DNA-damage-inducible 45 gamma 1.72 *** 1.85 **** NM_ Gpx1 glutathione peroxidase **** 2.63 **** NM_ Gpx2 glutathione peroxidase **** 1.06 NM_ Gsr glutathione reductase 1.95 **** 1.69 **** NM_ Gsta3 glutathione S-transferase, alpha **** NM_ Gstm7 glutathione S-transferase, mu *** *** NM_ Gsto1 glutathione S-transferase omega ** 2.55 *** NM_ Hmox1 heme oxygenase (decycling) **** 7 **** NM_ Mt1 metallothionein **** 4.55 **** NM_ Mt2 metallothionein **** 8.89 **** NM_ Parp12 poly (ADP-ribose) polymerase family, member **** 4.93 **** NM_ Parp2 poly (ADP-ribose) polymerase family, member *** -1.01
13 NM_ Parp3 poly (ADP-ribose) polymerase family, member **** 1.82 **** NM_ Parp9 poly (ADP-ribose) polymerase family, member **** 6.35 **** NM_ Pdrg1 p53 and DNA damage regulated * 1.2 NM_ Sod2 (MnSOD) superoxide dismutase 2, mitochondrial 4.47 **** 2.65 **** NM_ Txn1 thioredoxin *** NM_ Txnrd1 thioredoxin reductase **** 1.41 *** Cell cycle NM_ Ccnd1 cyclin D **** **** NM_ Ccnh cyclin H **** *** NM_ Cdk11b cyclin-dependent kinase 11B **** NM_ Cdk5 cyclin-dependent kinase **** *** NM_ Cnnm2 cyclin M **** **** NM_ Cnnm2 cyclin M *** Endoplasmic reticulum stress, translational regulation NM_ Atf3 activating transcription factor **** 6.89 **** NM_ Atf4 activating transcription factor ** 1.01 NR_ Atp2a2 (Serca2b) ATPase, Ca++ transporting, cardiac muscle, slow twitch **** *** NM_ Calb1 calbindin **** **** NM_ Ddit3 (Chop) DNA-damage inducible transcript *** 9.11 **** NM_ Derl1 Der1-like domain family, member * 1.51 *** NM_ Edem1 ER degradation enhancer, mannosidase alpha-like * 1.53 *** NM_ Eef1a2 eukaryotic translation elongation factor 1 alpha *** *** NM_ Eif1a eukaryotic translation initiation factor 1A 1.86 **** 1.04 * NM_ Eif2a eukaryotic translation initiation factor 2a NM_ Eif2ak2 eukaryotic translation initiation factor 2-alpha kinase **** 1.56 **** NM_ Eif2ak3 (Perk) eukaryotic translation initiation factor 2 alpha kinase *** 1.52 * NM_ Eif2c1 eukaryotic translation initiation factor 2C, **** *** NM_ Eif2c3 eukaryotic translation initiation factor 2C, **** 1.06 NM_ Eif2c4 eukaryotic translation initiation factor 2C, *** NM_ Eif4e1b eukaryotic translation initiation factor 4E family member 1B *** -1.1 NM_ Eif4ebp1 eukaryotic translation initiation factor 4E binding protein **** 1.35 * NM_ Eif5b eukaryotic translation initiation factor 5B 1.43 **** 1.56 **** NM_ Eif6 eukaryotic translation initiation factor **** 1.98 **** NM_ Ern1 (IRE1) endoplasmic reticulum (ER) to nucleus signalling *** 1.27 * AK Hspa1b (Hsp70) heat shock protein 1B NM_ Hspa5 (Bip) heat shock protein **** NM_ Hspa8 heat shock protein *** 1.43 **** NM_ Hspa9 heat shock protein **** NM_ Hspbap1 Hspb associated protein *** 1.38 **** NM_ Hspd1 heat shock protein 1 (chaperonin) *** 1.24 *** NM_ Hsph1 heat shock 105kDa/110kDa protein * 1.45 * NM_ Hyou1 (Orp150) hypoxia up-regulated **** **** NM_ Man2a1 mannosidase 2, alpha *** **** NM_ Mars methionine-trna synthetase * NM_ Psen1 presenilin **** 1.3 **** NM_ Sec61a1 Sec61 alpha 1 subunit (S. cerevisiae) -1.1 *** **** NM_ Sec61a2 Sec61, alpha subunit 2 (S. cerevisiae) **** Apoptosis related NM_ Aen apoptosis enhancing nuclease 1.58 **** 2.42 **** NM_ Apaf1 apoptotic peptidase activating factor **** 1.86 **** NM_ Bag1 BCL2-associated athanogene 1 (oncogene; increases Bcl2 act.) 1.4 **** 1.68 **** NM_ Bag2 BCL2-associated athanogene ** *** NM_ Bag3 BCL2-associated athanogene **** 1.27 NM_ Bak1 BCL2-antagonist/killer **** 1.61 **** NM_ Bax BCL2-associated X protein **** NM_ Bbc3 (Puma) BCL2 binding component * NM_ Bcl2l1 (Bcl-XL) BCL2-like **** 2.1 **** NM_ Bid BH3 interacting domain death agonist 3.73 **** 2.47 **** NM_ Bik BCL2-interacting killer 1.37 *** NM_ Casp1 caspase **** 6.54 **** NM_ Casp12 caspase *** 2.24 *** NM_ Casp2 caspase **** 1.31 ** NM_ Casp3 caspase **** 2.55 **** NM_ Casp4 caspase 4, apoptosis-related cysteine peptidase **** 8.34 ****
14 NM_ Casp7 caspase **** 5.27 **** NM_ Casp8 caspase **** 1.11 * NM_ Cflar CASP8 and FADD-like apoptosis regulator 1.57 **** *** NM_ Dap death-associated protein 1.39 *** 1.42 **** NM_ Dapl1 death associated protein-like **** **** NM_ Daxx (Fas Fas death domain-associated protein 2.64 **** 1.27 *** associated) NM_ Fas Fas (TNF receptor superfamily member 6) **** **** NM_ Hrk (Dp5) harakiri, BCL2 interacting protein (contains only BH3 domain) NM_ Moap1 modulator of apoptosis 1 (obs interacts with Bax) 1.6 **** 1.06 * NM_ Pdcd6ip programmed cell death 6 interacting protein (obs apoptosis 1.32 **** 1.61 **** NM_ Ppp1r15a protein phosphatase 1, regulatory (inhibitor) subunit 15A 1.34 *** 3.64 **** NM_ Tmbim1 transmembrane BAX inhibitor motif containing *** 1.68 **** NM_ Trib3 tribbles homolog 3 (Drosophila) 1.7 ** 4.67 **** ENSMUST Kinases, phosphatases Alpk1 alpha-kinase **** 2.46 **** Alpk1 alpha-kinase **** 2.39 **** Alpk1 alpha-kinase **** 2.14 *** NM_ Dusp1 dual specificity phosphatase 1 (obs dusps phosphatases modulate 2.18 ** 2.26 * ERK/MAPKs) NM_ Dusp10 dual specificity phosphatase **** 1.54 NM_ Dusp16 dual specificity phosphatase **** 2.53 ** NM_ Dusp18 dual specificity phosphatase **** **** NM_ Dusp19 dual specificity phosphatase **** *** NM_ Dusp3 dual specificity phosphatase 3 (vaccinia virus phosphatase VH **** 1.08 related) NM_ Dusp6 dual specificity phosphatase *** NM_ Dusp8 dual specificity phosphatase **** 3.55 ** NM_ Madd MAP-kinase activating death domain **** *** NM_ Mapk6 mitogen-activated protein kinase **** 1.66 * NM_ Mapkapk2 MAP kinase-activated protein kinase **** 2.27 **** NM_ Pak1 p21 protein (Cdc42/Rac)-activated kinase **** 1.82 **** NM_ Prkcd protein kinase C, delta 1.47 *** 1.49 **** NM_ Ptpn1 protein tyrosine phosphatase, non-receptor type **** 1.56 **** NM_ Ptpn23 protein tyrosine phosphatase, non-receptor type *** 1.61 *** Others NM_ Adcy6 adenylate cyclase **** *** NM_ Alox12 arachidonate 12-lipoxygenase 5.02 **** 2.56 **** NM_ Capn1 calpain **** 1.32 **** NM_ Gad1 (Gad67) glutamic acid decarboxylase **** * NM_ Iigp1 interferon inducible GTPase **** **** NM_ Pld1 phospholipase D **** 1.62 *** NM_ Ubd ubiquitin D **** **** NM_ Ubtd1 ubiquitin domain containing *** 1.19 ** NM_ Ucp2 uncoupling protein 2 (mitochondrial, proton carrier) *** ****
15 6 h (fold change) 24 h (fold change) Cyt 1.25+Cyt 1.25 Cyt 1.25+Cyt 1.25 Accession Symbol Gene name / functional group vs Con vs Cyt vs Con vs Con vs Cyt vs Con Cell cycle and cell growth NM_ Ccnc cyclin C * -1,15 * ** 1.3 *** NM_ Ccnh cyclin H **** ,1 ** *** 1.45 *** 1,23 * NM_ Cdk1 cyclin-dependent kinase **** *** NM_ Fmnl2 formin-like **** 1.15 ** 2,16 **** 1.51 *** 1.54 *** 2,06 **** NM_ Mlst8 MTOR associated protein, LST8 homolog (S. cerevisiae) * **** 1.48 *** 1.01 Cell death NM_ Dapl1 death associated protein-like **** ,71 *** **** *** NM_ Ggct gamma-glutamyl cyclotransferase 2.25 **** -1.4 *** ** Cellular stress, DNA damage NM_ Cat catalase 1.23 * 1.36 *** 1,57 *** 2.88 **** 1.95 *** 1,79 *** NM_ Gadd45g growth arrest and DNA-damage-inducible 45 gamma 1.72 *** ,13 *** 1.85 **** *** 1.06 NM_ Hmox1 heme oxygenase (decycling) **** 1.41 ** **** NM_ Nqo1 NAD(P)H dehydrogenase, quinone **** *** -1,16 * NF-κB signaling NM_ Ikbke (IKKε) inhibitor of kappab kinase epsilon 9.22 **** **** *** 1.05 NM_ Nfkb2 (p52) nuclear factor of kappa light polypeptide gene enhancer in B-cells 2, p49/p **** **** **** NM_ Nfkbib nuclear factor of kappa light polypeptide gene enhancer in B-cells inhibitor, beta 2.7 **** **** *** -1 (IκBβ) NM_ Nfkbie nuclear factor of kappa light polypeptide gene enhancer in B-cells inhibitor, epsilon 3.26 **** **** * *** (IκBε) NM_ Relt RELT tumor necrosis factor receptor **** 1.42 *** 1,7 **** **** 1.33 ** 1,6 **** NM_ Tnfaip8 tumor necrosis factor, alpha-induced protein **** ** 1.37 **** NM_ Tnfrsf11b tumor necrosis factor receptor superfamily, member 11b (osteoprotegerin) 6.81 **** 1.8 ** *** 2.37 **** NM_ Tnfrsf9 tumor necrosis factor receptor superfamily, member **** **** -1.6 ** -1.1 NM_ Trib3 tribbles homolog 3 (Drosophila) 1.7 ** **** 1.39 *** 1.09 Chemokines and cytokines NM_ Ccl2 chemokine (C-C motif) ligand **** *** **** ** (MCP-1) NM_ Ccl5 chemokine (C-C motif) ligand **** *** 1,13 * 4.21 **** (RANTES) NM_ Ccl8 chemokine (C-C motif) ligand **** * **** *** 1.04 (MCP-2) NM_ Cxcl1 chemokine (C-X-C motif) ligand **** -1.4 *** **** *** 1 NM_ Cxcl11 chemokine (C-X-C motif) ligand **** -1.4 *** **** -3 **** 1.05
16 NM_ Cxcl16 chemokine (C-X-C motif) ligand **** -1.3 *** **** -1.4 * -1 NM_ Cxcl2 chemokine (C-X-C motif) ligand **** -1.9 *** * NM_ Cxcl9 chemokine (C-X-C motif) ligand **** **** * 1 NM_ Il15 interleukin **** *** -1,1 *** 1.64 ** * 1.01 NM_ Il18 interleukin **** *** NM_ Il18r1 interleukin 18 receptor **** 1.9 **** **** 2.1 * 1.05 NM_ Il1rl2 interleukin 1 receptor-like *** *** 1.16 NM_ Il4i1 interleukin 4 induced **** -1.4 **** **** **** -1,17 ** NM_ Il6 interleukin **** * **** NM_ Il7 interleukin **** 1.46 *** **** 2.09 **** 1.06 Other immune response components NM_ Bat5 HLA-B associated transcript **** *** ** 1.04 NM_ C1s complement component 1, s subcomponent **** ** **** NM_ C3 complement component **** 2.29 **** **** 3.27 *** 1.08 NM_ Ceacam1 carcinoembryonic antigen-related cell adhesion molecule **** 1.54 ** 2,43 *** 2.98 **** ,55 *** NM_ Chi3l1 chitinase 3-like **** 2.36 **** **** 5.12 **** 1.07 (Ykl-40) NM_ Colec12 collectin sub-family member * 1.09 * ** **** 1.17 NM_ Defb1 defensin beta **** ,41 ** 3.06 *** 1.26 * 1.26 NM_ Hamp hepcidin antimicrobial peptide **** **** * -1.1 NM_ Icam1 intercellular adhesion molecule **** * **** -1.3 * NM_ Ifi204 interferon activated gene **** **** ** (Ifi16) NM_ Iigp1 interferon inducible GTPase **** **** 1.34 *** 1.02 NM_ Irg1 immunoresponsive gene **** 1.27 *** **** ,15 *** NM_ Irgm1 immunity-related GTPase family M member **** **** 1.44 ** NM_ Ltf lactotransferrin 8.3 **** 1.54 ** **** 1.51 *** NM_ Mx1 myxovirus (influenza virus) resistance **** **** 1,18 * 4.21 **** ,21 ** NR_ Mx2 myxovirus (influenza virus) resistance **** **** *** 1.1 NM_ Oas2 2'-5' oligoadenylate synthetase **** **** * NM_ Oasl1 2'-5' oligoadenylate synthetase-like **** **** *** 1.03 NM_ Orm1 orosomucoid ** 2.1 *** 1.03 NM_ Samhd1 SAM domain and HD domain, **** 1.63 **** **** 1.34 *** 1,08 * NM_ Tlr2 toll-like receptor **** **** 1.53 **** NM_ Tlr3 toll-like receptor **** ,1 ** 1.62 **** 1.32 * NM_ Tnfaip2 tumor necrosis factor, alpha-induced protein **** *** **** * NM_ Vcam1 vascular cell adhesion molecule **** **** **** 1.04 Ceramide, sphingomyelin, ganglioside NM_ A4galt alpha 1,4-galactosyltransferase 1.57 *** 1.33 *** 1,19 * 1.94 *** NM_ Smpdl3b sphingomyelin phosphodiesterase, acid-like 3B 2.25 **** *** ** NM_ St3gal5 ST3 beta-galactoside alpha-2,3-sialyltransferase **** 1.49 *** 2,33 **** **** 1.77 *** 1,73 ***
17 NM_ St8sia1 ST8 alpha-n-acetyl-neuraminide alpha-2,8-sialyltransferase **** 1.57 **** 1,54 **** **** 1.87 **** 1.52 Cell-to-cell contact, extracellular matrix NM_ Adam8 a disintegrin and metallopeptidase domain *** ,06 * 1.78 *** ** -1 NM_ Adamts4 a disintegrin-like and metallopeptidase (reprolysin type) with thrombospondin type 1 motif, *** *** -1.5 *** NM_ Adamts6 a disintegrin-like and metallopeptidase (reprolysin type) with thrombospondin type 1 motif, **** ** NM_ Amigo2 adhesion molecule with Ig like domain **** 2,69 **** 2.15 *** 3.09 **** 2,41 **** NM_ Frem2 Fras1 related extracellular matrix protein **** 1.61 **** ** ,25 * NM_ Itga6 integrin alpha **** 1.36 *** NM_ Lpp LIM domain containing preferred translocation partner in lipoma 1.53 *** *** **** 1.08 NM_ Mmp13 matrix metallopeptidase **** 4.01 **** 1,58 * 5.92 **** 3.95 **** 3,45 *** NM_ Mpp7 membrane protein, palmitoylated 7 (MAGUK p55 subfamily member 7) ** 2.37 **** 2,15 **** **** 2.81 **** 1,78 **** NM_ Nedd9 neural precursor cell expressed, developmentally down-regulated gene **** 1,95 **** 1.86 **** 2.41 **** 1,59 *** NM_ Pcdhb20 protocadherin beta * **** 1.51 *** NM_ Plau plasminogen activator, urokinase *** ** 1.07 NM_ Tgfbi transforming growth factor, beta induced 1.27 ** **** *** Exocytosis, intracellular trafficking, cytoskeleton NM_ Arhgap26 Rho GTPase activating protein **** **** **** 1.04 NM_ Arl6 ADP-ribosylation factor-like *** **** 1.32 *** NM_ Bet1 blocked early in transport 1 homolog (S. cerevisiae) **** * -1,11 * -3.9 **** 1.51 *** 1 NM_ Capg capping protein (actin filament), gelsolin-like *** **** 1.04 NM_ Clasp1 CLIP associating protein * **** 1.74 **** NM_ Golga1 golgi autoantigen, golgin subfamily a, **** *** 1.45 *** 1.08 NM_ Golim4 golgi integral membrane protein **** ,14 ** *** 1.33 ** NM_ Gsn gelsolin *** *** *** NM_ Mal2 mal, T-cell differentiation protein **** *** 1.34 ** -1,15 * NM_ Mylip myosin regulatory light chain interacting protein 1.48 *** **** -1,17 ** 1.62 **** *** NM_ Pls1 plastin 1 (I-isoform) **** ,1 *** **** 1.33 *** 1.06 NM_ Pstpip2 proline-serine-threonine phosphatase-interacting protein ** ** * NM_ Rab3b RAB3B, member RAS oncogene family *** *** NM_ Rab9b RAB9B, member RAS oncogene family **** 1.42 **** 1,8 **** **** 1.44 *** 1.2 NM_ Rnd3 Rho family GTPase **** *** -1,18 *** 4.42 *** NM_ Snx14 sorting nexin **** ,13 *** *** 1.34 ** NM_ Snx33 sorting nexin *** 1.31 *** 1,18 ** NM_ Snx7 sorting nexin *** *** * ** BC Stx11 syntaxin **** **** **** -1.3 *** NM_ Stx7 syntaxin **** 1.34 **** *** 1.31 **** 1.15 NM_ Stxbp6 syntaxin binding protein 6 (amisyn) **** -1.4 *** -1,31 *** **** ,2 *** NM_ Synj2 synaptojanin *** 1.83 **** 2,05 **** * 1.77 *** 1,84 **** NM_ Tmem27 transmembrane protein **** 1.23 **** 1,13 *** **** 1.69 **** 1,35 **** NM_ Trak2 trafficking protein, kinesin binding ** ,46 *** *** 1.39 *** 1,34 ****
18 Hormone and growth factor signaling NM_ Egfr epidermal growth factor receptor 1.64 **** 1.82 **** 1,15 *** 2.48 **** NM_ Ereg epiregulin 2.33 **** **** *** NM_ Fgf7 fibroblast growth factor * **** *** -1,25 * NM_ Irs2 insulin receptor substrate *** *** *** 1.08 NM_ Kit kit oncogene -1.4 * ,31 * 1.53 *** ** NM_ Pappa2 pappalysin *** *** *** NM_ Pappa2 pappalysin **** ,28 *** *** ** NM_ Pdgfa platelet derived growth factor, alpha **** 1.48 **** 1,52 **** 1.26 * 1.3 ** 1,46 **** NM_ Pdgfb platelet derived growth factor, B polypeptide 1.61 *** 1.53 **** NM_ Ralgds ral guanine nucleotide dissociation stimulator 3.2 **** 1.33 *** 1,52 *** 1.41 ** ,2 **** NM_ Vipr2 (VPAC2) vasoactive intestinal peptide receptor ,96 *** 1.92 **** 1.48 **** 1,51 * Transcription factors etc. NM_ Ankrd52 ankyrin repeat domain * *** 1.33 *** 1.13 NM_ Anks4b ankyrin repeat and sterile alpha motif domain containing 4B * *** 1.35 *** NM_ Bhlha15 basic helix-loop-helix family, member a * **** 1.46 *** 1.15 NM_ Dll1 delta-like 1 (Drosophila) 3.99 **** ** *** * -1,14 * NM_ Hes1 hairy and enhancer of split 1 (Drosophila) *** ,19 * 1.6 ** ** NM_ Id2 inhibitor of DNA binding **** *** -1,26 *** 2.01 *** NM_ Isl1 ISL1 transcription factor, LIM/homeodomain **** **** 1.3 * NM_ Lifr leukemia inhibitory factor receptor ** 1.26 * 1,35 * **** 1.39 *** 1,19 * NM_ Lmo1 LIM domain only *** *** *** 1.06 NM_ Myc myelocytomatosis oncogene 1.54 *** *** -1,33 * 4.74 *** NM_ Neurod1 neurogenic differentiation *** *** 1.43 *** 1,09 *** NM_ Nr1d1 nuclear receptor subfamily 1, group D, member **** ** NM_ Nrip1 nuclear receptor interacting protein * 1.36 ** 1,2 **** 1.88 *** 1.31 ** 1,35 *** NM_ Pax6 paired box gene **** **** 1.31 *** NM_ Snai2 snail homolog 2 (Drosophila) ,66 *** 1.81 ** 1.52 **** 1,4 * NM_ Tceal1 transcription elongation factor A (SII)-like **** ,21 * **** 1.34 ** NM_ Twist2 twist homolog 2 (Drosophila) 1.24 * *** ** 1,07 * Glucose metabolism NM_ Agl amylo-1,6-glucosidase, 4-alpha-glucanotransferase **** 1.17 **** 1,07 *** **** 1.48 *** 1.09 NM_ G6pc2 glucose-6-phosphatase, catalytic, **** 1.22 **** **** 1.54 **** 1,12 * NM_ Gck glucokinase ** *** 1.32 *** 1.07 NM_ Idh1 isocitrate dehydrogenase 1 (NADP+), soluble **** 1.37 **** 1,26 * **** 1.78 *** 1.05 NM_ Ldha lactate dehydrogenase A ** ** 1.09 NM_ Nubpl nucleotide binding protein-like ,15 *** ** 1.43 ** 1.02 NM_ Nudt14 nudix (nucleoside diphosphate linked moiety X)-type motif **** **** 1.3 *** 1.08 NM_ Pdha1 pyruvate dehydrogenase E1 alpha *** 1.3 *** **** 1.83 **** 1,25 ***
19 NM_ Pfkfb3 6-phosphofructo-2-kinase/fructose-2,6-biphosphatase **** -1.5 **** **** NM_ Pgm5 phosphoglucomutase *** 2.01 ** 1,94 **** Lipid and cholesterol metabolism NM_ Abca1 ATP-binding cassette, sub-family A (ABC1), member **** ** -1,31 **** 1.53 **** *** NM_ Abcg1 ATP-binding cassette, sub-family G (WHITE), member *** ** -1,29 **** 2.8 **** **** NM_ Acsl4 acyl-coa synthetase long-chain family member **** 1.16 * *** 1.47 **** NM_ Cyp24a1 cytochrome P450, family 24, subfamily a, polypeptide **** 56.2 **** 98,19 **** **** 88,95 **** NM_ Dgat1 diacylglycerol O-acyltransferase * **** 1.32 *** 1 NM_ Dgat2 diacylglycerol O-acyltransferase **** 1,34 **** 3.48 **** 3.1 **** 1,58 * NM_ Dhcr24 24-dehydrocholesterol reductase **** 1.19 ** **** 1.34 *** NM_ Dhrs9 dehydrogenase/reductase (SDR family) member **** 1.4 **** 1,56 **** NM_ Far1 fatty acyl CoA reductase *** **** 1.37 *** 1,2 *** NM_ Nceh1 arylacetamide deacetylase-like ** **** 1.49 **** 1,29 *** NM_ Naaa N-acylethanolamine acid amidase 1.13 *** 1.7 **** *** 1.66 **** 1.01 NM_ Pex7 peroxisomal biogenesis factor *** **** 1.37 ** 1.02 NM_ Pla2g7 phospholipase A2, group VII (platelet-activating factor acetylhydrolase, plasma) *** *** 1,8 *** NM_ Rdh9 retinol dehydrogenase *** ** ** Metabolism of amino acids and nucleic acids NM_ Bphl biphenyl hydrolase-like (serine hydrolase, breast epithelial mucin-associated antigen) **** ,7 **** ** 1.64 *** 1,55 **** NM_ Cpn1 carboxypeptidase N, polypeptide *** **** 1.46 **** 1.06 NM_ Entpd3 ectonucleoside triphosphate diphosphohydrolase *** 1.31 *** 1,34 *** 1.42 *** 1.41 *** 1,37 *** NM_ Suox sulfite oxidase *** **** 1.53 ** 1.07 NM_ Upp1 uridine phosphorylase **** **** ** Inositol phosphate metabolism NM_ Impa1 inositol (myo)-1(or 4)-monophosphatase ** 1,36 **** ** 1.67 *** 1,38 **** NM_ Inppl1 inositol polyphosphate phosphatase-like **** 1.81 **** 1,54 **** *** 1.26 *** 1,24 *** Metabolism of drugs etc. NM_ Adh1 alcohol dehydrogenase 1 (class I) -3.9 **** 1.8 **** 1,74 **** **** 2.83 **** 1,57 ** NM_ Aldh5a1 aldhehyde dehydrogenase family 5, subfamily A *** 1.31 *** NM_ Car13 carbonic anhydrase **** **** *** 1.19 NM_ Car8 carbonic anhydrase *** ** -1,31 * ** *** NM_ Sult1c2 sulfotransferase family, cytosolic, 1C, member **** 2.06 **** 2,28 **** **** 1,9 *** NM_ Ugt2b34 UDP glucuronosyltransferase 2 family, polypeptide B *** **** 1.9 **** 1.07 Splicing machinery, RNA modification NR_ Adarb1 adenosine deaminase, RNA-specific, B **** 1.44 *** 1,15 ** *** 1.67 **** 1.19 NM_ Cstf3 cleavage stimulation factor, 3' pre-rna, subunit * **** 1.33 *** 1,23 * NM_ Hdac6 histone deacetylase **** 1.34 **** **** 1.68 *** -1,1 ** NM_ Ints7 integrator complex subunit **** *** 1.31 *** -1 NM_ Nap1l2 nucleosome assembly protein 1-like **** 1.31 *** -1,26 *** NM_ Prpf4b PRP4 pre-mrna processing factor 4 homolog B (yeast) *** 1.37 *** 1,34 *** **** 1,29 ***
20 NM_ Trrap transformation/transcription domain-associated protein * **** 1.35 *** -1 Transport and homeostasis of ions etc. NM_ Abca8b ATP-binding cassette, sub-family A (ABC1), member 8b 2.06 **** ,05 ** 1.95 **** 1.38 ** NM_ Abcc2 ATP-binding cassette, sub-family C (CFTR/MRP), member ** **** 1.4 *** 1.11 NM_ Best3 bestrophin **** 1.24 **** 1,12 *** *** 1.42 * 1,44 **** NM_ Kcnh6 potassium voltage-gated channel, subfamily H (eag-related), member **** 1.32 *** 1,64 **** **** 1.66 *** 1,49 **** NM_ Mmgt2 membrane magnesium transporter * 1.46 **** 1,6 **** *** 1.74 *** 1,51 *** NM_ P2rx4 purinergic receptor P2X, ligand-gated ion channel **** 1.32 **** 1,91 **** *** 1.64 ** 1,54 **** NM_ Scn9a sodium channel, voltage-gated, type IX, alpha **** *** *** 1.4 ** NM_ Scn9a sodium channel, voltage-gated, type IX, alpha **** ** **** NM_ Scnn1g sodium channel, nonvoltage-gated 1 gamma 2.57 *** **** ,22 * NM_ Sgk1 serum/glucocorticoid regulated kinase **** ,41 **** ** *** NM_ Slc16a12 solute carrier family 16 (monocarboxylic acid transporters), member **** 1.6 *** **** 1.8 **** 1.05 NM_ Slc22a23 solute carrier family 22, member **** 1.51 **** **** 1.07 NM_ Slc25a35 solute carrier family 25, member **** 1.63 **** NM_ Slc31a2 solute carrier family 31, member **** ,8 *** ** 1.41 *** 1,56 **** NM_ Slc33a1 solute carrier family 33 (acetyl-coa transporter), member **** * -1,08 * **** 1.35 *** 1.03 NM_ Slc35a5 solute carrier family 35, member A * *** 1.32 ** NM_ Slc35d3 solute carrier family 35, member D **** * **** 1.33 **** 1.1 NM_ Slc35f2 solute carrier family 35, member F **** 2.82 **** 1,25 *** 1.51 **** NM_ Slc8a1 solute carrier family 8 (sodium/calcium exchanger), member **** **** 1.35 ** 1.07 NM_ Slco1a6 solute carrier organic anion transporter family, member 1a *** 1.56 **** 1,47 **** *** 2.18 *** 1,72 **** NM_ Stc1 stanniocalcin ** 1.54 ** **** ,03 * NM_ Steap1 six transmembrane epithelial antigen of the prostate **** 1-1,41 *** 2.61 **** 1.41 ** 1.04 NM_ Steap4 STEAP family member **** **** 2.02 *** Intracellular signaling NM_ Akap6 A kinase (PRKA) anchor protein *** *** -1,33 *** NM_ Arrb2 arrestin, beta **** 1.35 *** 1,76 **** *** 1.63 *** 1,86 *** NM_ Asb4 ankyrin repeat and SOCS box-containing **** *** **** **** NM_ Cnksr1 connector enhancer of kinase suppressor of Ras **** * **** *** 1.07 NM_ Dock8 dedicator of cytokinesis **** 1.15 * 1,14 * **** 1.77 *** 1.12 NM_ Ffar1 free fatty acid receptor **** **** -1,3 ** **** NM_ Gna14 guanine nucleotide binding protein, alpha **** 1.24 ** 1,6 *** 2.56 **** 1.38 ** 1.55 NM_ Grk5 G protein-coupled receptor kinase **** 1.45 *** 1,29 *** *** 1.66 **** 1,29 * NM_ Grtp1 GH regulated TBC protein **** **** -1,25 **** **** NM_ Mras muscle and microspikes RAS 1.25 * *** 1.4 *** NM_ Pde3b phosphodiesterase 3B, cgmp-inhibited **** ,46 *** *** 1.44 **** 1,21 **** NM_ Plcxd2 phosphatidylinositol-specific phospholipase C, X domain containing *** **** *** 1,21 *** NM_ Rasd2 RASD family, member *** **** 1.39 ** -1 NM_ Sdc1 syndecan **** 1.76 **** *** NM_ Sdc4 syndecan **** *** **** **** -1,18 *
SABiosciences PCR Array Catalog #: PAHS-021 SA+ SCF 4h stimulaton AVG(Ct) Position Unigene Refseq Symbol Description shcontr shcontr-4h shgskβ A01
 SABiosciences PCR Array Catalog #: PAHS-021 SA+ SCF 4h stimulaton AVG(Ct) Position Unigene Refseq Symbol Description shcontr shcontr-4h shgskβ A01 Hs.1274 NM_006129 BMP1 Bone morphogenetic protein 1 25.25
SABiosciences PCR Array Catalog #: PAHS-021 SA+ SCF 4h stimulaton AVG(Ct) Position Unigene Refseq Symbol Description shcontr shcontr-4h shgskβ A01 Hs.1274 NM_006129 BMP1 Bone morphogenetic protein 1 25.25
Supplementary Table 1. Primer sequences used for RT-qPCR validation of the microarray data. Primer. direction. Forward
 Supplementary Table 1. Primer sequences used for RT-qPCR validation of the microarray data Gene Glyceraldehyde 3-phosphate dehydrogenase (Gapdh) Cell death-inducing DFFAlike effector a (Cidea) Peroxisome
Supplementary Table 1. Primer sequences used for RT-qPCR validation of the microarray data Gene Glyceraldehyde 3-phosphate dehydrogenase (Gapdh) Cell death-inducing DFFAlike effector a (Cidea) Peroxisome
Η ΦΛΕΓΜΟΝΩ ΗΣ ΑΝΤΙ ΡΑΣΗ ΤΟΥ ΓΑΣΤΡΙΚΟΥ ΒΛΕΝΝΟΓΟΝΟΥ ΣΤΗ ΛΟΙΜΩΞΗ ΜΕ ΕΛΙΚΟΒΑΚΤΗΡΙ ΙΟ ΤΟΥ ΠΥΛΩΡΟΥ ΠΡΙΝ ΚΑΙ ΜΕΤΑ ΤΗ ΘΕΡΑΠΕΙΑ
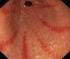 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ ΤΟΜΕΑΣ ΥΓΕΙΑΣ ΕΡΓΑΣΤΗΡΙΑΚΟΣ ΙΕΥΘΥΝΤΗΣ: Ο ΚΑΘΗΓΗΤΗΣ ΓΕΩΡΓΙΟΣ ΚΑΡΚΑΒΕΛΑΣ ΠΑΝΕΠ. ΕΤΟΣ 2008-2009 Αριθµ. 2084 Η ΦΛΕΓΜΟΝΩ ΗΣ ΑΝΤΙ
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ ΤΟΜΕΑΣ ΥΓΕΙΑΣ ΕΡΓΑΣΤΗΡΙΑΚΟΣ ΙΕΥΘΥΝΤΗΣ: Ο ΚΑΘΗΓΗΤΗΣ ΓΕΩΡΓΙΟΣ ΚΑΡΚΑΒΕΛΑΣ ΠΑΝΕΠ. ΕΤΟΣ 2008-2009 Αριθµ. 2084 Η ΦΛΕΓΜΟΝΩ ΗΣ ΑΝΤΙ
Supplemental Table 1. Oligonucleotides used to identify H-RAS, K-RAS and N-RAS mutations and PTEN gene expression.
 Supplemental Table 1. Oligonucleotides used to identify H-RAS, K-RAS and N-RAS mutations and PTEN gene expression. Gene Exon Forward primer Reverse primer Temp ( C) HRAS 2-3 ATGACGGAATATAAGCTGGT ATGGCAAACACACACAGGAA
Supplemental Table 1. Oligonucleotides used to identify H-RAS, K-RAS and N-RAS mutations and PTEN gene expression. Gene Exon Forward primer Reverse primer Temp ( C) HRAS 2-3 ATGACGGAATATAAGCTGGT ATGGCAAACACACACAGGAA
ΥΠΕΡΙΝΣΟΥΛΙΝΑΙΜΙΑ ΚΑΙ ΑΝΤΙΣΤΑΣΗ ΣΤΗΝ ΙΝΣΟΥΛΙΝΗ ΣΕ ΠΑΧΥΣΑΡΚΑ ΠΑΙΔΙΑ ΚΑΙ ΕΦΗΒΟΥΣ ΜΕ ΠΡΩΙΜΗ ΑΔΡΕΝΑΡΧΗ
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΚΑΙ ΠΡΟΛΗΠΤΙΚΗΣ ΙΑΤΡΙΚΗΣ ΕΡΓΑΣΤΗΡΙΟ ΒΙΟΧΗΜΕΙΑΣ ΔΙΕΥΘΥΝΤΡΙΑ: Η ΚΑΘΗΓΗΤΡΙΑ Ν. ΒΑΒΑΤΣΗ-ΧΡΙΣΤΑΚΗ ΠΑΝΕΠ. ΕΤΟΣ 2009-2010 Αριθμ.2456
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΒΙΟΛΟΓΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΚΑΙ ΠΡΟΛΗΠΤΙΚΗΣ ΙΑΤΡΙΚΗΣ ΕΡΓΑΣΤΗΡΙΟ ΒΙΟΧΗΜΕΙΑΣ ΔΙΕΥΘΥΝΤΡΙΑ: Η ΚΑΘΗΓΗΤΡΙΑ Ν. ΒΑΒΑΤΣΗ-ΧΡΙΣΤΑΚΗ ΠΑΝΕΠ. ΕΤΟΣ 2009-2010 Αριθμ.2456
Ο ΡΟΛΟΣ ΤΩΝ ΚΑΝΝΑΒΙΝΟΕΙ ΩΝ ΚΑΤΑ ΤΗΝ ΕΜΒΡΥΪΚΗ ΑΝΑΠΤΥΞΗ
 ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ Πρόγραµµα Μεταπτυχιακών Σπουδών Εφαρµογές στις Βασικές Ιατρικές Επιστήµες Εργαστήριο Ανατοµικής-Ιστολογίας-Εµβρυολογίας ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Ο ΡΟΛΟΣ ΤΩΝ ΚΑΝΝΑΒΙΝΟΕΙ
ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ Πρόγραµµα Μεταπτυχιακών Σπουδών Εφαρµογές στις Βασικές Ιατρικές Επιστήµες Εργαστήριο Ανατοµικής-Ιστολογίας-Εµβρυολογίας ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Ο ΡΟΛΟΣ ΤΩΝ ΚΑΝΝΑΒΙΝΟΕΙ
Supplementary figures for MED13 dependent signaling from the heart confers leanness by enhancing metabolism in adipose tissue and liver.
 Supplementary figures for MED13 dependent signaling from the heart confers leanness by enhancing metabolism in adipose tissue and liver. Content: Figure S1. Cardiac overexpression of MED13 increases metabolic
Supplementary figures for MED13 dependent signaling from the heart confers leanness by enhancing metabolism in adipose tissue and liver. Content: Figure S1. Cardiac overexpression of MED13 increases metabolic
Μελέτη της αντιμεταλλαξιγόνου δράσης φλαβονοειδών του φυτού Lotus Edulis με τη μέθοδο Ames test
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΙΑΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΒΙΟΧΗΜΕΙΑΣ & ΒΙΟΤΕΧΝΟΛΟΓΙΑΣ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Μελέτη της αντιμεταλλαξιγόνου δράσης φλαβονοειδών του φυτού Lotus Edulis με τη μέθοδο Ames test Επιμέλεια
ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΙΑΣ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΒΙΟΧΗΜΕΙΑΣ & ΒΙΟΤΕΧΝΟΛΟΓΙΑΣ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Μελέτη της αντιμεταλλαξιγόνου δράσης φλαβονοειδών του φυτού Lotus Edulis με τη μέθοδο Ames test Επιμέλεια
Debashish Sahay. To cite this version: HAL Id: tel
 Identification of genes activated and biological markers involved in lysophosphatidic acid (LPA)-induced breast cancer metastasis through its receptor LPA1 Debashish Sahay To cite this version: Debashish
Identification of genes activated and biological markers involved in lysophosphatidic acid (LPA)-induced breast cancer metastasis through its receptor LPA1 Debashish Sahay To cite this version: Debashish
Κυτταρική Επικοινωνία Cell Communication
 Κυτταρική Επικοινωνία Cell Communication Η μοίρα του κυττάρου ρυθμίζεται από εξωτερικά σήματα (από το περιβάλλον και άλλα κύτταρα) survival division differentiation apoptosis Είδη κυτταρικής επικοινωνίας
Κυτταρική Επικοινωνία Cell Communication Η μοίρα του κυττάρου ρυθμίζεται από εξωτερικά σήματα (από το περιβάλλον και άλλα κύτταρα) survival division differentiation apoptosis Είδη κυτταρικής επικοινωνίας
ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΣΤΙΣ «ΚΛΙΝΙΚΕΣ ΚΑΙ ΚΛΙΝΙΚΟΕΡΓΑΣΤΗΡΙΑΚΕΣ ΙΑΤΡΙΚΕΣ ΕΙΔΙΚΟΤΗΤΕΣ»
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΣΤΙΣ «ΚΛΙΝΙΚΕΣ ΚΑΙ ΚΛΙΝΙΚΟΕΡΓΑΣΤΗΡΙΑΚΕΣ ΙΑΤΡΙΚΕΣ ΕΙΔΙΚΟΤΗΤΕΣ» ΑΡΝΗΤΙΚΗ ΡΥΘΜΙΣΗ ΤΗΣ ΜΕΤΑΒΙΒΑΣΗΣ ΤΟΥ ΣΗΜΑΤΟΣ ΤΗΣ
ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΣΧΟΛΗ ΕΠΙΣΤΗΜΩΝ ΥΓΕΙΑΣ ΤΜΗΜΑ ΙΑΤΡΙΚΗΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΣΤΙΣ «ΚΛΙΝΙΚΕΣ ΚΑΙ ΚΛΙΝΙΚΟΕΡΓΑΣΤΗΡΙΑΚΕΣ ΙΑΤΡΙΚΕΣ ΕΙΔΙΚΟΤΗΤΕΣ» ΑΡΝΗΤΙΚΗ ΡΥΘΜΙΣΗ ΤΗΣ ΜΕΤΑΒΙΒΑΣΗΣ ΤΟΥ ΣΗΜΑΤΟΣ ΤΗΣ
BD Cytometric Bead Array Flex Set System
 BD Cytometric Bead Array Flex Set System BD Cytometric Bead Array System BD Biosciences Pharmingen ELISA Cytometric Bead Array CBA System Capture Caputure Serum Supernatant Lysate PE -Detection 1 2 PE
BD Cytometric Bead Array Flex Set System BD Cytometric Bead Array System BD Biosciences Pharmingen ELISA Cytometric Bead Array CBA System Capture Caputure Serum Supernatant Lysate PE -Detection 1 2 PE
Μελέτη της έκφρασης του ογκοκατασταλτικού γονιδίου Cyld στον καρκίνο του μαστού
 Σχολή Θετικών Επιστημών Τμήμα Βιολογίας Πρόγραμμα Μεταπτυχιακών Σπουδών Κατεύθυνση: Εφαρμοσμένη γενετική και βιοτεχνολογία ΜΕΤΑΠΤΥΧΙΑΚΗ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Μελέτη της έκφρασης του ογκοκατασταλτικού γονιδίου
Σχολή Θετικών Επιστημών Τμήμα Βιολογίας Πρόγραμμα Μεταπτυχιακών Σπουδών Κατεύθυνση: Εφαρμοσμένη γενετική και βιοτεχνολογία ΜΕΤΑΠΤΥΧΙΑΚΗ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Μελέτη της έκφρασης του ογκοκατασταλτικού γονιδίου
< (0.999) Graft (0.698) (0.483) <0.001 (0.698) (<0.001) (<0.001) 3 months (0.999) (0.483) (<0.001) 6 months (<0.
 Supplementary table 1. Correlation of endothelial cell density among graft, 3, 6, and 12 months after Descemet s automated stripping endothelial keratoplasty. Graft 3 months 6 months 12 months Graft
Supplementary table 1. Correlation of endothelial cell density among graft, 3, 6, and 12 months after Descemet s automated stripping endothelial keratoplasty. Graft 3 months 6 months 12 months Graft
Supplemental Figures and Tables
 KLF2 and KLF4 control endothelial identity and vascular integrity Panjamaporn Sangwung 1, 2,#, Guangjin Zhou 1,#, Lalitha Nayak 1,3, E. Ricky Chan 4, Sandeep Kumar 5, Dong-Won Kang 5, Rongli Zhang 1, Xudong
KLF2 and KLF4 control endothelial identity and vascular integrity Panjamaporn Sangwung 1, 2,#, Guangjin Zhou 1,#, Lalitha Nayak 1,3, E. Ricky Chan 4, Sandeep Kumar 5, Dong-Won Kang 5, Rongli Zhang 1, Xudong
Overlapping sets of transcripts from host and non-host interactions of tomato are expressed early during non-host resistance
 POJ 7(1):19-27 (2014) ISSN:1836-3644 Overlapping sets of transcripts from host and non-host interactions of tomato are expressed early during non-host resistance Battepati Uma and Appa Rao Podile* Supplementary
POJ 7(1):19-27 (2014) ISSN:1836-3644 Overlapping sets of transcripts from host and non-host interactions of tomato are expressed early during non-host resistance Battepati Uma and Appa Rao Podile* Supplementary
ΙΩΑΝΝΗΣ ΚΛΑΓΚΑΣ. Χημικός, MSc ΔΙΔΑΚΤΟΡΙΚΗ ΔΙΑΤΡΙΒΗ ΥΠΟΒΛΗΘΗΚΕ ΣΤΗΝ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΥ ΑΡΙΣΤΟΤΕΛΕΙΟΥ ΠΑΝΕΠΙΣΤΗΜΙΟΥ ΘΕΣΣΑΛΟΝΙΚΗΣ
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΦΥΣΙΟΛΟΓΙΑΣ ΦΑΡΜΑΚΟΛΟΓΙΑΣ B ΕΡΓΑΣΤΗΡΙΟ ΦΑΡΜΑΚΟΛΟΓΙΑΣ ΔΙΕΥΘΥΝΤΗΣ: ΚΑΘΗΓΗΤΗΣ ΓΙΩΡΓΟΣ ΚΑΡΑΚΙΟΥΛΑΚΗΣ ΠΑΝΕΠ. ΕΤΟΣ 2008-2009 ΑΡΙΘΜ. 2376 Η ΕΠΙΔΡΑΣΗ
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΦΥΣΙΟΛΟΓΙΑΣ ΦΑΡΜΑΚΟΛΟΓΙΑΣ B ΕΡΓΑΣΤΗΡΙΟ ΦΑΡΜΑΚΟΛΟΓΙΑΣ ΔΙΕΥΘΥΝΤΗΣ: ΚΑΘΗΓΗΤΗΣ ΓΙΩΡΓΟΣ ΚΑΡΑΚΙΟΥΛΑΚΗΣ ΠΑΝΕΠ. ΕΤΟΣ 2008-2009 ΑΡΙΘΜ. 2376 Η ΕΠΙΔΡΑΣΗ
ΜΟΡΙΑΚΕΣ ΜΕΘΟΔΟΙ ΚΡΙΤΗΡΙΑ ΕΠΙΛΟΓΗΣ ΑΞΙΟΛΟΓΗΣΗ
 - - ό ό : Ω ί ό ί ώ.. ά ή ί ός ή ς ύ ί ί ή έ ώ ό. ή ς ά ς ής ό ός ά ς ή έ ός ή ς ί ής ί ς. ό έ ώ - ώ ή ής ή ς- ί ά ά ς ί ς. ά ί ί έ έ ά ύ ή, ό ί ά, ό ό ά έ ά ής ί ύ George Wald ή ί έ ς ί ύ ό ς ί ς ά έ
- - ό ό : Ω ί ό ί ώ.. ά ή ί ός ή ς ύ ί ί ή έ ώ ό. ή ς ά ς ής ό ός ά ς ή έ ός ή ς ί ής ί ς. ό έ ώ - ώ ή ής ή ς- ί ά ά ς ί ς. ά ί ί έ έ ά ύ ή, ό ί ά, ό ό ά έ ά ής ί ύ George Wald ή ί έ ς ί ύ ό ς ί ς ά έ
Cellular Physiology and Biochemistry
 Original Paper 2016 The Author(s). 2016 Published The Author(s) by S. Karger AG, Basel Published online: November 25, 2016 www.karger.com/cpb Published by S. Karger AG, Basel 486 www.karger.com/cpb Accepted:
Original Paper 2016 The Author(s). 2016 Published The Author(s) by S. Karger AG, Basel Published online: November 25, 2016 www.karger.com/cpb Published by S. Karger AG, Basel 486 www.karger.com/cpb Accepted:
Κυτταρική Επικοινωνία Cell Communication
 Κυτταρική Επικοινωνία Cell Communication Κυτταρική Επικοινωνία Είναι η ικανότητα του κυττάρου να αποκρίνεται σε εξωτερικά σήματα (σήματα από το περιβάλλον του ή άλλα κύτταρα) Τα σήματα αυτά μπορεί να είναι
Κυτταρική Επικοινωνία Cell Communication Κυτταρική Επικοινωνία Είναι η ικανότητα του κυττάρου να αποκρίνεται σε εξωτερικά σήματα (σήματα από το περιβάλλον του ή άλλα κύτταρα) Τα σήματα αυτά μπορεί να είναι
Christopher Stephen Inchley, 2015
 Christopher Stephen Inchley, 2015 Series of dissertations submitted to the Faculty of Medicine, University of Oslo No. 2009 ISBN 978-82-8264-990-2 All rights reserved. No part of this publication may be
Christopher Stephen Inchley, 2015 Series of dissertations submitted to the Faculty of Medicine, University of Oslo No. 2009 ISBN 978-82-8264-990-2 All rights reserved. No part of this publication may be
Human angiogenin is a potent cytotoxin in the absence of ribonuclease inhibitor
 Human angiogenin is a potent cytotoxin in the absence of ribonuclease inhibitor SYDNEY P. THOMAS, 1 TRISH T. HOANG, 2 VALERIE T. RESSLER, 4 and RONALD T. RAINES 2,3,4 1 Graduate Program in Cell & Molecular
Human angiogenin is a potent cytotoxin in the absence of ribonuclease inhibitor SYDNEY P. THOMAS, 1 TRISH T. HOANG, 2 VALERIE T. RESSLER, 4 and RONALD T. RAINES 2,3,4 1 Graduate Program in Cell & Molecular
Chapter 1. Fingolimod attenuates ceramide induced blood-brain barrier dysfunction in multiple sclerosis by targeting reactive astrocytes
 Chapter 1 Fingolimod attenuates ceramide induced blood-brain barrier dysfunction in multiple sclerosis by targeting reactive astrocytes 1 1 1 1 CHAPTER SUMMARY FINGOLIMOD ATTENUATES CERAMIDE PRODUCTION
Chapter 1 Fingolimod attenuates ceramide induced blood-brain barrier dysfunction in multiple sclerosis by targeting reactive astrocytes 1 1 1 1 CHAPTER SUMMARY FINGOLIMOD ATTENUATES CERAMIDE PRODUCTION
Sar1B-mediated Regulation of Triglyceride and Cholesterol
 FIGURE S1. Comparing Sar1A- and Sar1B-flag Over-expression on RNA Profiles of McArdle RH7777 cells. RNA was prepared from two Sar1B:H79G-flag cell lines and two control cell lines plus two Sar1B-flag and
FIGURE S1. Comparing Sar1A- and Sar1B-flag Over-expression on RNA Profiles of McArdle RH7777 cells. RNA was prepared from two Sar1B:H79G-flag cell lines and two control cell lines plus two Sar1B-flag and
A rearrangement is established as a barrier in parapatry. The accumulation of incompatibilites starts. Chrom 1. Rearranged
 Figure S1. An ancestral species splits in two according to the model discussed in Navarro and Barton (Evolution. In press. 2003). A rearrangement in chromosome 1 was involved in the process. From the moment
Figure S1. An ancestral species splits in two according to the model discussed in Navarro and Barton (Evolution. In press. 2003). A rearrangement in chromosome 1 was involved in the process. From the moment
High mobility group 1 HMG1
 Vol. 29, pp.705 ~ 711, 2001 High mobility group 1 HMG1 13 12 20 anti-neutrophil cytoplasmic antibodies, ANCA ANCA 1982 Davies 1980 1 high mobility group HMG1 HMG2 30 kd high mobility group HMGHMG HMG1
Vol. 29, pp.705 ~ 711, 2001 High mobility group 1 HMG1 13 12 20 anti-neutrophil cytoplasmic antibodies, ANCA ANCA 1982 Davies 1980 1 high mobility group HMG1 HMG2 30 kd high mobility group HMGHMG HMG1
CPT. Tsuchiya. beta. quantitative RT PCR QIAGEN IGFBP. Fect Transfection Reagent sirna. RT PCR RNA Affymetrix GeneChip Expression Array
 7 PTEN p Tsuchiya DNA Hepatocyte nuclear factor beta HNF beta HNFbeta IGFBP GLUT G Pase MODY maturity onset diabetes of the young Tsuchiya HNFbeta HNFbeta HNFbeta sirna CPT HNFbeta HNFbeta TOVG KOC ESMCAS
7 PTEN p Tsuchiya DNA Hepatocyte nuclear factor beta HNF beta HNFbeta IGFBP GLUT G Pase MODY maturity onset diabetes of the young Tsuchiya HNFbeta HNFbeta HNFbeta sirna CPT HNFbeta HNFbeta TOVG KOC ESMCAS
Table S2. Sequences immunoprecipitating with anti-hif-1α
 Genome-wide analysis of HIF DNA-bdg-supplemental material Table S2. Sequences immunoprecipitatg with anti-α The table gives the chromosomal co-ordates of each anti-α immunoprecipitated locus meetg criteria
Genome-wide analysis of HIF DNA-bdg-supplemental material Table S2. Sequences immunoprecipitatg with anti-α The table gives the chromosomal co-ordates of each anti-α immunoprecipitated locus meetg criteria
Supplementary Figure 1. Therapeutic efficacy of combined NDV and anti-ctla-4 therapy is attenuated with a larger tumor challenge.
 Supplementary Figure 1. Therapeutic efficacy of combined NDV and anti-ctla-4 therapy is attenuated with a larger tumor challenge. Bilateral flank B16-F10 tumors were established as described and the animals
Supplementary Figure 1. Therapeutic efficacy of combined NDV and anti-ctla-4 therapy is attenuated with a larger tumor challenge. Bilateral flank B16-F10 tumors were established as described and the animals
Contents Part I Psychoneuroimmunology and Systems Biology Mechanisms 1 From Psychoneuroimmunology to Personalized, Systems, and Dynamical Medicine
 Contents Part I Psychoneuroimmunology and Systems Biology Mechanisms 1 From Psychoneuroimmunology to Personalized, Systems, and Dynamical Medicine... 3 1.1 Psychoneuroimmunology (PNI) and Systems Biology...
Contents Part I Psychoneuroimmunology and Systems Biology Mechanisms 1 From Psychoneuroimmunology to Personalized, Systems, and Dynamical Medicine... 3 1.1 Psychoneuroimmunology (PNI) and Systems Biology...
The NF-κB pathways in connective tissue diseases
 κ The NF-κB pathways in connective tissue diseases TETSUO KUBOTA (Associate Professor) Tokyo Medical and Dental University Graduate School of Health Care Sciences NF-κB nuclear factor-kappa B NF-κB in
κ The NF-κB pathways in connective tissue diseases TETSUO KUBOTA (Associate Professor) Tokyo Medical and Dental University Graduate School of Health Care Sciences NF-κB nuclear factor-kappa B NF-κB in
nasopharyngeal carcinoma NPC 30% 20% CNE-2R CNE-2 Human-6v3. 0 CNE-2R 2 CNE-2R CNE-2 2 Toll
 2011 4 31 2 Chin J Radiol Med Prot April 2011 Vol. 31 No. 2 167 CNE-2R CNE-2 CNE-2R Human-6v3. 0 2 CNE-2R CNE-2 2 374 Kegg Biocarta Toll IL-1R 13 JUN MYD88 CCL5 CXCL10 STAT1 LY96 FOS CCL3 IL-6 IL-8 IL-1α
2011 4 31 2 Chin J Radiol Med Prot April 2011 Vol. 31 No. 2 167 CNE-2R CNE-2 CNE-2R Human-6v3. 0 2 CNE-2R CNE-2 2 374 Kegg Biocarta Toll IL-1R 13 JUN MYD88 CCL5 CXCL10 STAT1 LY96 FOS CCL3 IL-6 IL-8 IL-1α
Hvordan påvirker CO 2 post-smolt i brakkvann RAS?
 Hvordan påvirker CO 2 post-smolt i brakkvann RAS? Tom O. Nilsen,. Mota, V.C., Kolarevic, J., Ytterborg, E., Ebbesson, Lars O.E., Handeland, S.O., Stiller, K., Terjesen, B.F SUNNDALSØRA, 12. NOVEMBER 2018
Hvordan påvirker CO 2 post-smolt i brakkvann RAS? Tom O. Nilsen,. Mota, V.C., Kolarevic, J., Ytterborg, E., Ebbesson, Lars O.E., Handeland, S.O., Stiller, K., Terjesen, B.F SUNNDALSØRA, 12. NOVEMBER 2018
Supplemental Figure 1. Pathways associated with the most significantly altered FGF-8b-regulated
 Supplemental Figure legends Supplemental Figure 1. Pathways associated with the most significantly altered FGF-8b-regulated genes. a) Mitotic roles of Polo-like kinase. b) Cell cycle regulation by BTG
Supplemental Figure legends Supplemental Figure 1. Pathways associated with the most significantly altered FGF-8b-regulated genes. a) Mitotic roles of Polo-like kinase. b) Cell cycle regulation by BTG
ΚΥΤΤΑΡΙΚΟΣ ΘΑΝΑΤΟΣ ΚΑΙ ΑΝΑΓΕΝΝΗΣΗ Β-ΚΥΤΤΑΡΟΥ ΝΕΟΤΕΡΑ ΔΕΔΟΜΕΝΑ ΓΙΑ ΤΗ ΘΕΡΑΠΕΙΑ ΤΟΥ ΣΑΚΧΑΡΩΔΗ ΔΙΑΒΗΤΗ
 ΚΥΤΤΑΡΙΚΟΣ ΘΑΝΑΤΟΣ ΚΑΙ ΑΝΑΓΕΝΝΗΣΗ Β-ΚΥΤΤΑΡΟΥ ΝΕΟΤΕΡΑ ΔΕΔΟΜΕΝΑ ΓΙΑ ΤΗ ΘΕΡΑΠΕΙΑ ΤΟΥ ΣΑΚΧΑΡΩΔΗ ΔΙΑΒΗΤΗ ΚΑΛΛΙΟΠΗ ΚΩΤΣΑ ΕΠΙΚ. ΚΑΘΗΓΗΤΡΙΑ ΕΝΔΟΚΡΙΝΟΛΟΓΙΑΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ Α.Π.Θ ΤΜΗΜΑ ΕΝΔΟΚΡΙΝΟΛΟΓΙΑΣ-ΔΙΑΒΗΤΟΛΟΓΙΚΟ
ΚΥΤΤΑΡΙΚΟΣ ΘΑΝΑΤΟΣ ΚΑΙ ΑΝΑΓΕΝΝΗΣΗ Β-ΚΥΤΤΑΡΟΥ ΝΕΟΤΕΡΑ ΔΕΔΟΜΕΝΑ ΓΙΑ ΤΗ ΘΕΡΑΠΕΙΑ ΤΟΥ ΣΑΚΧΑΡΩΔΗ ΔΙΑΒΗΤΗ ΚΑΛΛΙΟΠΗ ΚΩΤΣΑ ΕΠΙΚ. ΚΑΘΗΓΗΤΡΙΑ ΕΝΔΟΚΡΙΝΟΛΟΓΙΑΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ Α.Π.Θ ΤΜΗΜΑ ΕΝΔΟΚΡΙΝΟΛΟΓΙΑΣ-ΔΙΑΒΗΤΟΛΟΓΙΚΟ
α α Pneumocystis jirovecii
 α α Key words α α Pneumocystis jirovecii I α Table 1. Biologics currently approved in Japan for autoimmune inflammatory diseases (as of Dec, 2016) Classification Preparations that target cytokines or cytokine
α α Key words α α Pneumocystis jirovecii I α Table 1. Biologics currently approved in Japan for autoimmune inflammatory diseases (as of Dec, 2016) Classification Preparations that target cytokines or cytokine
Τα ηπατικά επίπεδα του FOXP3 mrna στη χρόνια ηπατίτιδα Β εξαρτώνται από την έκφραση των οδών Fas/FasL και PD-1/PD-L1
 Τα ηπατικά επίπεδα του FOXP3 mrna στη χρόνια ηπατίτιδα Β εξαρτώνται από την έκφραση των οδών Fas/FasL και PD-1/PD-L1 Γ. Γερμανίδης, Ν. Αργέντου, Θ. Βασιλειάδης, Κ. Πατσιαούρα, Κ. Μαντζούκης, A. Θεοχαρίδου,
Τα ηπατικά επίπεδα του FOXP3 mrna στη χρόνια ηπατίτιδα Β εξαρτώνται από την έκφραση των οδών Fas/FasL και PD-1/PD-L1 Γ. Γερμανίδης, Ν. Αργέντου, Θ. Βασιλειάδης, Κ. Πατσιαούρα, Κ. Μαντζούκης, A. Θεοχαρίδου,
SUPPLEMENTAL MATERIAL
 SUPPLEMENTAL MATERIAL 1. Methods MicroRNA TaqMan Low Density Array MiRNAs were reverse transcribed and amplified using the multiplex RT TaqMan MicroRNA Low Density Array (TLDA) (Life Technologies). Global
SUPPLEMENTAL MATERIAL 1. Methods MicroRNA TaqMan Low Density Array MiRNAs were reverse transcribed and amplified using the multiplex RT TaqMan MicroRNA Low Density Array (TLDA) (Life Technologies). Global
Long intergenic non-coding RNA expression signature in human breast cancer
 Long intergenic non-coding RNA expression signature in human breast cancer Yanfeng Zhang 1,5,#, Erin K. Wagner 2,#, Xingyi Guo 1, Isaac May 3, Qiuyin Cai 1, Wei Zheng 1, Chunyan He 2,4,*, Jirong Long 1,*
Long intergenic non-coding RNA expression signature in human breast cancer Yanfeng Zhang 1,5,#, Erin K. Wagner 2,#, Xingyi Guo 1, Isaac May 3, Qiuyin Cai 1, Wei Zheng 1, Chunyan He 2,4,*, Jirong Long 1,*
Growth Factors/Wachstumsfaktoren. Human (Hu) Cytokines and Growth Factors/Humane (Hu) Cytokine und Wachstumsfaktoren
 Growth Factors/Wachstumsfaktoren Human (Hu) Cytokines and Growth Factors/Humane (Hu) Cytokine und Wachstumsfaktoren 4-1BBr Hu 4-1BB Receptor rec. CB-1010100 5 μg CB-1010101 20 μg CB-1010102 1 mg Acrp30
Growth Factors/Wachstumsfaktoren Human (Hu) Cytokines and Growth Factors/Humane (Hu) Cytokine und Wachstumsfaktoren 4-1BBr Hu 4-1BB Receptor rec. CB-1010100 5 μg CB-1010101 20 μg CB-1010102 1 mg Acrp30
Μαρία Κατσιφοδήμου. Ο ρόλος της έκκρισης HLA-G από τα ανθρώπινα έμβρυα στην επιτυχία της εξωσωματικής γονιμοποίησης. Μεταπτυχιακή Διπλωματική Εργασία
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΘΕΤΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΤΜΗΜΑ ΒΙΟΛΟΓΙΑΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΚΑΤΕΥΘΥΝΣΗ: ΕΦΑΡΜΟΣΜΕΝΗ ΓΕΝΕΤΙΚΗ ΚΑΙ ΒΙΟΤΕΧΝΟΛΟΓΙΑ Ο ρόλος της έκκρισης HLA-G από τα ανθρώπινα
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΣΧΟΛΗ ΘΕΤΙΚΩΝ ΕΠΙΣΤΗΜΩΝ ΤΜΗΜΑ ΒΙΟΛΟΓΙΑΣ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥΔΩΝ ΚΑΤΕΥΘΥΝΣΗ: ΕΦΑΡΜΟΣΜΕΝΗ ΓΕΝΕΤΙΚΗ ΚΑΙ ΒΙΟΤΕΧΝΟΛΟΓΙΑ Ο ρόλος της έκκρισης HLA-G από τα ανθρώπινα
TFDP2 TFDP1 SUMO1 SERTAD1 RPA3 RBBP8 RAD9A RAD17 RAD1 NBN MRE11A MNAT1 KPNA2 KNTC1 HUS1 HERC5 GTSE1 GTF2H1 GADD45A E2F4 DNM2 DDX11 CUL3 SKP2 RBL1 RB1
 -8-6 -4-2 CHEK2 MKI67 RB1 RBL1 SKP2 Fold-Change Fold-Change -3-2 -1 CCND1 CCNE1 CDK4 CDK5R1 5 1 15 ATM ATR BAX BCCIP BRCA1 BRCA2 BL1AT ABL1A ABL1 CCNG1 CCNG2 CCNH CCNT1 CCNT2 CDC2 CDC34 CDKN1A CDKN1B CDKN2A
-8-6 -4-2 CHEK2 MKI67 RB1 RBL1 SKP2 Fold-Change Fold-Change -3-2 -1 CCND1 CCNE1 CDK4 CDK5R1 5 1 15 ATM ATR BAX BCCIP BRCA1 BRCA2 BL1AT ABL1A ABL1 CCNG1 CCNG2 CCNH CCNT1 CCNT2 CDC2 CDC34 CDKN1A CDKN1B CDKN2A
Supporting Information. Multigenerational Disruption of Thyroid Endocrine System in Marine Medaka
 Supporting Information Multigenerational Disruption of Thyroid Endocrine System in Marine Medaka after A Life-Cycle Exposure to Perfluorobutane Sulfonate (PFBS) Lianguo Chen,, Chenyan Hu #, Mirabelle M.
Supporting Information Multigenerational Disruption of Thyroid Endocrine System in Marine Medaka after A Life-Cycle Exposure to Perfluorobutane Sulfonate (PFBS) Lianguo Chen,, Chenyan Hu #, Mirabelle M.
Data-Driven Discovery of Extravasation Pathway in Circulating Tumor Cells
 Data-Driven Discovery of Extravasation Pathway in Circulating Tumor Cells Yadavalli S. 1, Jayaram S. 1,2, Manda SS. 1,3, Madugundu AK. 1,3, Nayakanti DS. 1, Tuan TZ. 6, Bhat R. 4, Rangarajan A. 4, Chatterjee
Data-Driven Discovery of Extravasation Pathway in Circulating Tumor Cells Yadavalli S. 1, Jayaram S. 1,2, Manda SS. 1,3, Madugundu AK. 1,3, Nayakanti DS. 1, Tuan TZ. 6, Bhat R. 4, Rangarajan A. 4, Chatterjee
Guo_Fig. S1. Atg7 +/+ Atg7 -/- FBP_M+6 DHAP_M+3. Glucose Uptake Rate G6P_M+6. nmol/ul cell/hr
 Guo_Fig. S1 A C D nmol/ul cell/hr nmol/ul cell/hr Glucose Uptake Rate.3.2.1 *. Lactate Secretion Rate.4.3.2.1. Atg7-/- B Glucose G3P Lac Glucose G6P FBP 3-PG Pyr DHAP cycle Carbon from labeled glucose
Guo_Fig. S1 A C D nmol/ul cell/hr nmol/ul cell/hr Glucose Uptake Rate.3.2.1 *. Lactate Secretion Rate.4.3.2.1. Atg7-/- B Glucose G3P Lac Glucose G6P FBP 3-PG Pyr DHAP cycle Carbon from labeled glucose
Βιταμίνη D και καρκίνος
 Βιταμίνη D και καρκίνος Αντώνης Πολυμέρης Ενδοκρινολόγος Επιμελητής Α Β Ενδοκρινολογικό Τμήμα ΓΝΑ «Αλεξάνδρα» Ιστορικό Αυξημένη επίπτωση καρκίνου παχέος εντέρου στον Βορρά σε σχέση με τον Νότο στις ΗΠΑ
Βιταμίνη D και καρκίνος Αντώνης Πολυμέρης Ενδοκρινολόγος Επιμελητής Α Β Ενδοκρινολογικό Τμήμα ΓΝΑ «Αλεξάνδρα» Ιστορικό Αυξημένη επίπτωση καρκίνου παχέος εντέρου στον Βορρά σε σχέση με τον Νότο στις ΗΠΑ
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ. ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥ ΩΝ «Ιατρική Ερευνητική Μεθοδολογία» ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ
 ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥ ΩΝ «Ιατρική Ερευνητική Μεθοδολογία» ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Νόσος Pompe: Νεότερα κλινικά, γενετικά, διαγνωστικά και θεραπευτικά
ΑΡΙΣΤΟΤΕΛΕΙΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΘΕΣΣΑΛΟΝΙΚΗΣ ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΠΡΟΓΡΑΜΜΑ ΜΕΤΑΠΤΥΧΙΑΚΩΝ ΣΠΟΥ ΩΝ «Ιατρική Ερευνητική Μεθοδολογία» ΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ Νόσος Pompe: Νεότερα κλινικά, γενετικά, διαγνωστικά και θεραπευτικά
BD OptEIA ELISA Sets ELISA
 BD OptEIA ELISA Sets ELISA BD OptEIA ELISA Sets ELISA CaptureDetection Detection 9620 170,000 ELISAEnzymed-Linked ImmunoSorbent Assay CaptureDetection BD OptEIA ELISA Set 2 BD OptEIA ELISA Set BD OptEIA
BD OptEIA ELISA Sets ELISA BD OptEIA ELISA Sets ELISA CaptureDetection Detection 9620 170,000 ELISAEnzymed-Linked ImmunoSorbent Assay CaptureDetection BD OptEIA ELISA Set 2 BD OptEIA ELISA Set BD OptEIA
abs acces acces Sample test results. Actual results may vary. Antioxidants B-Vitamins Minerals Order Today At Results Overview
 Order Today At www.accesalabs. Antioxidants B-Vitamins Minerals Vitamin C Pyridoxine - Biotin Vitamin A / Carotenoids Vitamin E / Tocopher α-lipoic A Co T ami B1 cer R bo a n - B2 N acin - B3 Results Overview
Order Today At www.accesalabs. Antioxidants B-Vitamins Minerals Vitamin C Pyridoxine - Biotin Vitamin A / Carotenoids Vitamin E / Tocopher α-lipoic A Co T ami B1 cer R bo a n - B2 N acin - B3 Results Overview
Supplemental Table S1. Tumor specific networks are enriched with somatically mutated genes (taken from the database COSMIC)
 Additional file 1 Supplemental Table S1. Tumor specific networks are enriched with somatically mutated genes (taken from the database COSMIC) COSMIC genes in tumor network COSMIC genes not in tumor network
Additional file 1 Supplemental Table S1. Tumor specific networks are enriched with somatically mutated genes (taken from the database COSMIC) COSMIC genes in tumor network COSMIC genes not in tumor network
Table S1A. Generic EMT signature for tumour
 Table S1A. Generic EMT signature for tumour Index Gene Symbol Gene Title GO Molecular Function Weight Weight.Zt ransform ed Category In Generic Cell Line EMT Signature = 1 1 KRT19 Keratin, type I cytoskeletal
Table S1A. Generic EMT signature for tumour Index Gene Symbol Gene Title GO Molecular Function Weight Weight.Zt ransform ed Category In Generic Cell Line EMT Signature = 1 1 KRT19 Keratin, type I cytoskeletal
Hepatic Stellate Cells: Multifunctional mesenchymal cells of the Liver
 Nagoya Med. J., 137 Hepatic Stellate Cells: Multifunctional mesenchymal cells of the Liver KAZUO IKEDA Department of Anatomy and Cell Biology, Graduate School of Medical Sciences, Nagoya City University
Nagoya Med. J., 137 Hepatic Stellate Cells: Multifunctional mesenchymal cells of the Liver KAZUO IKEDA Department of Anatomy and Cell Biology, Graduate School of Medical Sciences, Nagoya City University
5- CACGAAACTACCTTCAACTCC-3 beta actin-r 5- CATACTCCTGCTTGCTGATC-3 GAPDH-F GAPDH-R
 Table S1 Primers used in this work LncPPARδ-F 5- AGGATCCCAAGGCAGAAGTT -3 LncPPARδ-R 5- GAATAGAAAGGCGGGAAAGG -3 PPARδ-F 5- CACCCAGTGTCCTTCCATCT -3 PPARδ-R 5- CAGAATTGGGTGTGCTGCTT -3 ADRP-F 5- ACAACCGAGTGTGGTGACTC
Table S1 Primers used in this work LncPPARδ-F 5- AGGATCCCAAGGCAGAAGTT -3 LncPPARδ-R 5- GAATAGAAAGGCGGGAAAGG -3 PPARδ-F 5- CACCCAGTGTCCTTCCATCT -3 PPARδ-R 5- CAGAATTGGGTGTGCTGCTT -3 ADRP-F 5- ACAACCGAGTGTGGTGACTC
Supplementary Table 1. Primers used for RT-qPCR analysis of striatal and nigral tissue.
 Supplementary Table 1. Primers used for RT-qPCR analysis of striatal and nigral tissue. Gene Forward Primer (5-3 ) Reverse Primer (5-3 ) Dopaminergic Markers TH CTG GCC ATT GAT GTA CTG GA ACA CAC ATG GGA
Supplementary Table 1. Primers used for RT-qPCR analysis of striatal and nigral tissue. Gene Forward Primer (5-3 ) Reverse Primer (5-3 ) Dopaminergic Markers TH CTG GCC ATT GAT GTA CTG GA ACA CAC ATG GGA
BioVision Catalog Description Pack Size Supplier Price (RS)
 Catalog Description Pack Size Supplier Price (RS) K101-100 Annexin V-FITC Apoptosis Kit 100 assays BioVision 60991 K101-25 Annexin V-FITC Apoptosis Kit 25 assays BioVision 28373 K101-400 Annexin V-FITC
Catalog Description Pack Size Supplier Price (RS) K101-100 Annexin V-FITC Apoptosis Kit 100 assays BioVision 60991 K101-25 Annexin V-FITC Apoptosis Kit 25 assays BioVision 28373 K101-400 Annexin V-FITC
Growth Factors/Wachstumsfaktoren. Human (Hu) Cytokines and Growth Factors/Humane (Hu) Cytokine und Wachstumsfaktoren
 4-1BBr Hu 4-1BB Receptor rec. CB-1010100 5 μg CB-1010101 20 μg CB-1010102 1 mg Acrp30 Glob Hu Adiponectin Globular rec. CB-2800004 2 μg CB-2800005 10 μg CB-280000 1 mg Acrp30 HEK Hu Adiponectin glycosilated,
4-1BBr Hu 4-1BB Receptor rec. CB-1010100 5 μg CB-1010101 20 μg CB-1010102 1 mg Acrp30 Glob Hu Adiponectin Globular rec. CB-2800004 2 μg CB-2800005 10 μg CB-280000 1 mg Acrp30 HEK Hu Adiponectin glycosilated,
ΠΡΟΣΚΛΗΣΗ ΕΚΔΗΛΩΣΗΣ ΕΝΔΙΑΦΕΡΟΝΤΟΣ
 1 ΕΛΛΗΝΙΚΗ ΔΗΜΟΚΡΑΤΙΑ ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΡΗΤΗΣ ΕΙΔΙΚΟΣ ΛΟΓΑΡΙΑΣΜΟΣ ΑΝΑΡΤΗΤΕΑ ΣΤΟ ΔΙΑΔΙΚΤΥΟ ΠΡΟΣΚΛΗΣΗ ΕΚΔΗΛΩΣΗΣ ΕΝΔΙΑΦΕΡΟΝΤΟΣ Ηράκλειο, 04.02.2014 Αρ. πρωτ. 1054 Ο Ειδικός Λογαριασμός του Πανεπιστημίου Κρήτης
1 ΕΛΛΗΝΙΚΗ ΔΗΜΟΚΡΑΤΙΑ ΠΑΝΕΠΙΣΤΗΜΙΟ ΚΡΗΤΗΣ ΕΙΔΙΚΟΣ ΛΟΓΑΡΙΑΣΜΟΣ ΑΝΑΡΤΗΤΕΑ ΣΤΟ ΔΙΑΔΙΚΤΥΟ ΠΡΟΣΚΛΗΣΗ ΕΚΔΗΛΩΣΗΣ ΕΝΔΙΑΦΕΡΟΝΤΟΣ Ηράκλειο, 04.02.2014 Αρ. πρωτ. 1054 Ο Ειδικός Λογαριασμός του Πανεπιστημίου Κρήτης
Gene Primer sequence (5 to 3 ) Size (bp) E (%) GenBank
 Table S1 Primers used for quantitative real time PCR Gene Primer sequence (5 to 3 ) Size (bp) E (%) GenBank accession no: scd a got2a a ube2h a gpx4b a mknk2b a hsd11b2 b eef1a c b2m d F: ACCCGGAAGTCATCGAGAGA
Table S1 Primers used for quantitative real time PCR Gene Primer sequence (5 to 3 ) Size (bp) E (%) GenBank accession no: scd a got2a a ube2h a gpx4b a mknk2b a hsd11b2 b eef1a c b2m d F: ACCCGGAAGTCATCGAGAGA
Table S1: Inpatient Diet Composition
 Table S1: Inpatient Diet Composition Diet Components (% of Energy) Inpatient Period Baseline Intervention Protein 15.1 ± 0.0 15.0 ± 0.1 Total fat 30.0 ± 0.0 30.0 ± 0.1 Saturated fat 8.6 ± 0.8 8.4 ± 0.9
Table S1: Inpatient Diet Composition Diet Components (% of Energy) Inpatient Period Baseline Intervention Protein 15.1 ± 0.0 15.0 ± 0.1 Total fat 30.0 ± 0.0 30.0 ± 0.1 Saturated fat 8.6 ± 0.8 8.4 ± 0.9
Differentially expressed genes of HEK293/HeLa Myst4 sirna vs. controls.
 Supplemental data - Content Supplemental figures Figure 1 Figure 2 Figure 3 Figure 4 Western blot analysis of MYST4. Expression pattern of MYST4 isoforms. Differentially expressed genes of HEK293/HeLa
Supplemental data - Content Supplemental figures Figure 1 Figure 2 Figure 3 Figure 4 Western blot analysis of MYST4. Expression pattern of MYST4 isoforms. Differentially expressed genes of HEK293/HeLa
JCVI Locus Protein Name. JCVI Role Category JCVI Locus (LT2) Annotation name
 Additional File 4. Detailed information on all 41 genes that showed evidence for positive selection from positive selection analysis, in which gene Alignment Primary Gene Genbank Locus JCVI Locus Protein
Additional File 4. Detailed information on all 41 genes that showed evidence for positive selection from positive selection analysis, in which gene Alignment Primary Gene Genbank Locus JCVI Locus Protein
 ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΦΥΣΙΟΛΟΓΙΑΣ ΦΑΡΜΑΚΟΛΟΓΙΑΣ ΕΡΓΑΣΤΗΡΙΟ ΠΕΙΡΑΜΑΤΙΚΗΣ ΦΑΡΜΑΚΟΛΟΓΙΑΣ ΙΕΥΘΥΝΤΡΙΑ: ΚΑΘΗΓΗΤΡΙΑ ΒΑΣΙΛΙΚΗ ΜΗΡΤΣΟΥ-ΦΙ ΑΝΗ ΠΑΝΕΠ. ΕΤΟΣ 2005-2006 ΑΡΙΘΜ. 1844 ΙΕΡΕΥΝΗΣΗ ΤΟΥ ΡΟΛΟΥ ΑΜΙΝΟΞΕΩΝ, ΠΕΠΤΙ
ΙΑΤΡΙΚΗ ΣΧΟΛΗ ΤΟΜΕΑΣ ΦΥΣΙΟΛΟΓΙΑΣ ΦΑΡΜΑΚΟΛΟΓΙΑΣ ΕΡΓΑΣΤΗΡΙΟ ΠΕΙΡΑΜΑΤΙΚΗΣ ΦΑΡΜΑΚΟΛΟΓΙΑΣ ΙΕΥΘΥΝΤΡΙΑ: ΚΑΘΗΓΗΤΡΙΑ ΒΑΣΙΛΙΚΗ ΜΗΡΤΣΟΥ-ΦΙ ΑΝΗ ΠΑΝΕΠ. ΕΤΟΣ 2005-2006 ΑΡΙΘΜ. 1844 ΙΕΡΕΥΝΗΣΗ ΤΟΥ ΡΟΛΟΥ ΑΜΙΝΟΞΕΩΝ, ΠΕΠΤΙ
Structural and Biochemical Studies of Cotranslational Protein Transport across the Endoplasmic Reticulum
 Structural and Biochemical Studies of Cotranslational Protein Transport across the Endoplasmic Reticulum Inaugural-Dissertation zur Erlangung des akademischen Grades Doktor der Naturwissenschaften - Dr.
Structural and Biochemical Studies of Cotranslational Protein Transport across the Endoplasmic Reticulum Inaugural-Dissertation zur Erlangung des akademischen Grades Doktor der Naturwissenschaften - Dr.
A strategy for the identification of combinatorial bioactive compounds. contributing to the holistic effect of herbal medicines
 1 2 Supplementary information 3 4 A strategy for the identification of combinatorial bioactive compounds contributing to the holistic effect of herbal medicines 5 6 Fang Long 1, Hua Yang 1, Yanmin Xu,
1 2 Supplementary information 3 4 A strategy for the identification of combinatorial bioactive compounds contributing to the holistic effect of herbal medicines 5 6 Fang Long 1, Hua Yang 1, Yanmin Xu,
Supplementary Table 1: Primer sequence used in real-time PCR for binding site validation
 Supplementary Table 1: Primer sequence used in real-time PCR for binding site validation Gene Region (hg19) Primer TCF7L2 chr10:114709983-114710213 TCGCCCTGTCAATAATCTCC AGCCGAAGATACAGGAGGTG TCF7L2 chr10:114767138-114767346
Supplementary Table 1: Primer sequence used in real-time PCR for binding site validation Gene Region (hg19) Primer TCF7L2 chr10:114709983-114710213 TCGCCCTGTCAATAATCTCC AGCCGAAGATACAGGAGGTG TCF7L2 chr10:114767138-114767346
Πειραματικός Διαβήτης
 Πειραματικός Διαβήτης Δέσποινα Ν. Περρέα Αναπληρώτρια Καθηγήτρια Ιατρικής Σχολής Πανεπιστημίου Αθηνών Διευθύντρια Εργαστηρίου Πειραματικής Χειρουργικής και Χειρουργικής Έρευνας «Ν.Σ. Χρηστέας» ΤΡΩΚΤΙΚΑ
Πειραματικός Διαβήτης Δέσποινα Ν. Περρέα Αναπληρώτρια Καθηγήτρια Ιατρικής Σχολής Πανεπιστημίου Αθηνών Διευθύντρια Εργαστηρίου Πειραματικής Χειρουργικής και Χειρουργικής Έρευνας «Ν.Σ. Χρηστέας» ΤΡΩΚΤΙΚΑ
Ενδοκυττάρια ιαµερίσµατα, ιαλογή και µεταφορά πρωτεινών
 Ενδοκυττάρια ιαµερίσµατα, ιαλογή και µεταφορά πρωτεινών Ένα ηπατικό κύτταρο σε διατοµή Η.Μ Compartment Main Function Cytosol contains many metabolic pathways (Chapters 3 and 4); protein synthesis (Chapter
Ενδοκυττάρια ιαµερίσµατα, ιαλογή και µεταφορά πρωτεινών Ένα ηπατικό κύτταρο σε διατοµή Η.Μ Compartment Main Function Cytosol contains many metabolic pathways (Chapters 3 and 4); protein synthesis (Chapter
Biological marks of early-life socioeconomic experience is detected in the adult inflammatory transcriptome
 Biological marks of early-life socioeconomic experience is detected in the adult inflammatory transcriptome Raphaële Castagné 1,2,3, Michelle Kelly-Irving 2,3, Gianluca Campanella 1, Florence Guida 1,
Biological marks of early-life socioeconomic experience is detected in the adult inflammatory transcriptome Raphaële Castagné 1,2,3, Michelle Kelly-Irving 2,3, Gianluca Campanella 1, Florence Guida 1,
NES: normalized enrichment score (analyzed using KEGG pathway gene sets in the GSEA software); FDR:
 Supplementary table1. List of gene sets with simultaneously altered the enrichment upon SAP18 overexpression and knockdown. Gene sets enriched by SAP18 and reduced by shsap18 SAP18 against LacZ shsap18
Supplementary table1. List of gene sets with simultaneously altered the enrichment upon SAP18 overexpression and knockdown. Gene sets enriched by SAP18 and reduced by shsap18 SAP18 against LacZ shsap18
ΙΑΤΑΡΑΧΕΣ ΜΕΤΑΒΟΛΙΣΜΟΥ ΛΙΠΑΡΩΝ ΟΞΕΩΝ
 Ηµ/νία Εξέτασης : ΙΑΤΑΡΑΧΕΣ ΜΕΤΑΒΟΛΙΣΜΟΥ ΛΙΠΑΡΩΝ ΟΞΕΩΝ Medium Chain Acyl CoA Dehydrogenase C6< 0.24 C8< 0.35 C10< 0.3 C10:1 < 0.22 C8/C2< 0.02 C8/C10 < 3 Long Chain 3 Hydroxyacyl Coa Dehydrogenase C16OH
Ηµ/νία Εξέτασης : ΙΑΤΑΡΑΧΕΣ ΜΕΤΑΒΟΛΙΣΜΟΥ ΛΙΠΑΡΩΝ ΟΞΕΩΝ Medium Chain Acyl CoA Dehydrogenase C6< 0.24 C8< 0.35 C10< 0.3 C10:1 < 0.22 C8/C2< 0.02 C8/C10 < 3 Long Chain 3 Hydroxyacyl Coa Dehydrogenase C16OH
3 Το επίθημα -άσ(η) στη βιο-χημεία. Suffix -άσ(η) in Biochemistry. Άννα Αναστασιάδη-Συμεωνίδη, Ασημάκης Φλιάτουρας
 3 Το επίθημα -άσ(η) στη βιο-χημεία Άννα Αναστασιάδη-Συμεωνίδη, Ασημάκης Φλιάτουρας ΠΕΡΙΛΗΨΗ 1 Περιγράφεται το επιστημονικό επίθημα -άση που χρησιμεύει στην ορολογία της βιοχημείας στο να κατασκευάζει ουσιαστικά
3 Το επίθημα -άσ(η) στη βιο-χημεία Άννα Αναστασιάδη-Συμεωνίδη, Ασημάκης Φλιάτουρας ΠΕΡΙΛΗΨΗ 1 Περιγράφεται το επιστημονικό επίθημα -άση που χρησιμεύει στην ορολογία της βιοχημείας στο να κατασκευάζει ουσιαστικά
Supplementary Table 1. Demographic characteristics of the included ADNI subjects. rfmri. MMSE tics (n=907; (n=1092; n long =107.
 Supplementary Fig. 1. Assumed disease state (DS) vs aging relationships. In a) the DS increases linearly with age, while in b) it follows a sigmoid relationship with age (as suggested by 1 ). In both cases,
Supplementary Fig. 1. Assumed disease state (DS) vs aging relationships. In a) the DS increases linearly with age, while in b) it follows a sigmoid relationship with age (as suggested by 1 ). In both cases,
JCVI Role Category_1 JCVI Role Category_2. histidinol phosphatase-related protein Unknown function NT01ST0303 AAL19211 STM0248
 Additional Table 3. Detailed information on all 81 genes that showed evidence for positive selection from initial positive selection analysis, in Protein Name Gene name JCVI Role Category_1 JCVI Role Category_2
Additional Table 3. Detailed information on all 81 genes that showed evidence for positive selection from initial positive selection analysis, in Protein Name Gene name JCVI Role Category_1 JCVI Role Category_2
Study of the function of PGC-1β in white adipose tissue and its contribution to the development of insulin resistance
 Study of the function of PGC-1β in white adipose tissue and its contribution to the development of insulin resistance Natalia Enguix Elena Phd Thesis Barcelona, 2014 Laboratory of Metabolism and Obesity
Study of the function of PGC-1β in white adipose tissue and its contribution to the development of insulin resistance Natalia Enguix Elena Phd Thesis Barcelona, 2014 Laboratory of Metabolism and Obesity
DrugBank. Vol. 39 Issue 13 July NECT BAI51B CXTD JYBZZ-XS-110. Tel
 * * 100102 - DrugBank - - - - 1 - - 5 2 3-4 - 2013-11-27 81373985 81173568 NECT-11-0605 2008BAI51B01 2011-6 CXTD-11 2013-JYBZZ-XS-110 * Tel 010 84738620 E-mail wangyun @ bucm. edu. cn * 010 84738620 E-mail
* * 100102 - DrugBank - - - - 1 - - 5 2 3-4 - 2013-11-27 81373985 81173568 NECT-11-0605 2008BAI51B01 2011-6 CXTD-11 2013-JYBZZ-XS-110 * Tel 010 84738620 E-mail wangyun @ bucm. edu. cn * 010 84738620 E-mail
rp, ribosomal protein 60S rp mrna rpl30 rps14 RT-PCR mrna nonsense-mediated mrna decay smg-2 smg-2 mrna rp rpl-1 rpl-1
 rp, ribosomal protein 60S 47 rpl 40S 32 rps rp mrna rpl30 rps14 rpl12 rpl3 rps13 rp mrna nonsense-mediated mrna decay (NMD) C. elegans smg-2 smg-2 mrna 50 rp 7 rp 4 rpl 4 rp NMD mrna 6 rp mrna rp mrna
rp, ribosomal protein 60S 47 rpl 40S 32 rps rp mrna rpl30 rps14 rpl12 rpl3 rps13 rp mrna nonsense-mediated mrna decay (NMD) C. elegans smg-2 smg-2 mrna 50 rp 7 rp 4 rpl 4 rp NMD mrna 6 rp mrna rp mrna
ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΤΜΗΜΑ ΒΙΟΛΟΓΙΑΣ ΤΟΜΕΑΣ ΓΕΝΕΤΙΚΗΣ, ΒΙΟΛΟΓΙΑΣ ΚΥΤΤΑΡΟΥ & ΑΝΑΠΤΥΞΗΣ
 ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΤΜΗΜΑ ΒΙΟΛΟΓΙΑΣ ΤΟΜΕΑΣ ΓΕΝΕΤΙΚΗΣ, ΒΙΟΛΟΓΙΑΣ ΚΥΤΤΑΡΟΥ & ΑΝΑΠΤΥΞΗΣ «Επίδραση Gram+ και Gram- βακτηρίων στην παραγωγή των κυτταροκινών INF-γ και TNF-α, από ανθρώπινα NK κύτταρα» ΠΡΟΓΡΑΜΜΑ
ΠΑΝΕΠΙΣΤΗΜΙΟ ΠΑΤΡΩΝ ΤΜΗΜΑ ΒΙΟΛΟΓΙΑΣ ΤΟΜΕΑΣ ΓΕΝΕΤΙΚΗΣ, ΒΙΟΛΟΓΙΑΣ ΚΥΤΤΑΡΟΥ & ΑΝΑΠΤΥΞΗΣ «Επίδραση Gram+ και Gram- βακτηρίων στην παραγωγή των κυτταροκινών INF-γ και TNF-α, από ανθρώπινα NK κύτταρα» ΠΡΟΓΡΑΜΜΑ
Η παθογένεια της απώλειας οστικής μάζας στις φλεγμονώδεις αρθροπάθειες
 Ανασκόπηση EΛΛΗΝΙΚΗ ΡΕΥΜΑΤΟΛΟΓΙΑ 2007,18(1):60-75 Η παθογένεια της απώλειας οστικής μάζας στις φλεγμονώδεις αρθροπάθειες Ν.Γ. ΓΑΛΑΝΟΠΟΥΛΟΣ Ι.Α. ΠΑΠΑΔΟΠΟΥΛΟΣ Γ.Π. ΚΑΜΠΑΚΗΣ K. ΜΠΛΑΜΗΣ ΠΕΡΙΛΗΨΗ Οι φλεγμονώδεις
Ανασκόπηση EΛΛΗΝΙΚΗ ΡΕΥΜΑΤΟΛΟΓΙΑ 2007,18(1):60-75 Η παθογένεια της απώλειας οστικής μάζας στις φλεγμονώδεις αρθροπάθειες Ν.Γ. ΓΑΛΑΝΟΠΟΥΛΟΣ Ι.Α. ΠΑΠΑΔΟΠΟΥΛΟΣ Γ.Π. ΚΑΜΠΑΚΗΣ K. ΜΠΛΑΜΗΣ ΠΕΡΙΛΗΨΗ Οι φλεγμονώδεις
Research progress in inducing stem cells to differentiate toward the β-like cells of pancreatic islet
 19 5 2007 10 Chinese Bulletin of Life Sciences Vol. 19, No. 5 Oct., 2007 1004-0374(2007)05-0526-05 β ( 510632) β 1 β Q813 R587.1 Research progress in inducing stem cells to differentiate toward the β-like
19 5 2007 10 Chinese Bulletin of Life Sciences Vol. 19, No. 5 Oct., 2007 1004-0374(2007)05-0526-05 β ( 510632) β 1 β Q813 R587.1 Research progress in inducing stem cells to differentiate toward the β-like
Σύμφωνα με την Αμερικάνικη Εθνική Επιτροπή πρόληψης, διάγνωσης, εκτίμησης
 34 Σακχαρώδης διαβήτης - αρτηριακή υπέρταση Γ. ΠΙΑΔΙΤΗΣ Δ/ντής Ενδοκρινολογικού Τμήματος - Κέντρου Διαβήτη Γ.Κ.Ν.Α. «Γ. Γεννηματάς» Σύμφωνα με την Αμερικάνικη Εθνική Επιτροπή πρόληψης, διάγνωσης, εκτίμησης
34 Σακχαρώδης διαβήτης - αρτηριακή υπέρταση Γ. ΠΙΑΔΙΤΗΣ Δ/ντής Ενδοκρινολογικού Τμήματος - Κέντρου Διαβήτη Γ.Κ.Ν.Α. «Γ. Γεννηματάς» Σύμφωνα με την Αμερικάνικη Εθνική Επιτροπή πρόληψης, διάγνωσης, εκτίμησης
Reviews and Monographs
 Reviews and Monographs Progress in Biochemistry and Biophysics 2009, 36(12): 1516~1522 wwwpibbaccn * Toll ** ( 100005) Toll (Toll-like receptors TLRs) TLRs TLRs TLRs TLRs Toll (TLRs) Q93 DOI: 103724/SPJ1206200900213
Reviews and Monographs Progress in Biochemistry and Biophysics 2009, 36(12): 1516~1522 wwwpibbaccn * Toll ** ( 100005) Toll (Toll-like receptors TLRs) TLRs TLRs TLRs TLRs Toll (TLRs) Q93 DOI: 103724/SPJ1206200900213
Supplementary Table 1. Composition of the diets
 Supplementary Table 1. Composition of the diets Standard diet High Fat diet Energy content (kcal/kg) 2508 3701 Protein (g/kg) 200 170 Carbohydrate (g/kg) 720 670 Total fat (g/kg) 80 160 Saturated fatty
Supplementary Table 1. Composition of the diets Standard diet High Fat diet Energy content (kcal/kg) 2508 3701 Protein (g/kg) 200 170 Carbohydrate (g/kg) 720 670 Total fat (g/kg) 80 160 Saturated fatty
Τι το νεότερο στη φαρμακευτική θεραπεία; Παραμένουν οι στατίνες ως φάρμακα επιλογής;
 Τι το νεότερο στη φαρμακευτική θεραπεία; Παραμένουν οι στατίνες ως φάρμακα επιλογής; Άννα Ταυρίδου,, PhD Εργαστήριο Φαρμακολογίας, Τμήμα Ιατρικής, ημοκρίτειο Πανεπιστήμιο Θράκης Υπάρχει ανάγκη για καινούργια
Τι το νεότερο στη φαρμακευτική θεραπεία; Παραμένουν οι στατίνες ως φάρμακα επιλογής; Άννα Ταυρίδου,, PhD Εργαστήριο Φαρμακολογίας, Τμήμα Ιατρικής, ημοκρίτειο Πανεπιστήμιο Θράκης Υπάρχει ανάγκη για καινούργια
ΓΕΩΠΟΝΙΚΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΑΘΗΝΩΝ ΤΜΗΜΑ ΕΠΙΣΤΗΜΗΣ ΤΡΟΦΙΜΩΝ ΚΑΙ ΔΙΑΤΡΟΦΗΣ ΤΟΥ ΑΝΘΡΩΠΟΥ
 ΓΕΩΠΟΝΙΚΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΑΘΗΝΩΝ ΤΜΗΜΑ ΕΠΙΣΤΗΜΗΣ ΤΡΟΦΙΜΩΝ ΚΑΙ ΔΙΑΤΡΟΦΗΣ ΤΟΥ ΑΝΘΡΩΠΟΥ ΜΕΤΑΠΤΥΧΙΑΚΟ ΠΡΟΓΡΑΜΜΑ ΣΠΟΥΔΩΝ «ΕΠΙΣΤΗΜΗΣ ΚΑΙ ΤΕΧΝΟΛΟΓΙΑΣ ΤΡΟΦΙΜΩΝ ΚΑΙ ΔΙΑΤΡΟΦΗΣ ΤΟΥ ΑΝΘΡΩΠΟΥ» ΕΡΓΑΣΤΗΡΙΟ ΓΑΛΑΚΤΟΚΟΜΙΑΣ
ΓΕΩΠΟΝΙΚΟ ΠΑΝΕΠΙΣΤΗΜΙΟ ΑΘΗΝΩΝ ΤΜΗΜΑ ΕΠΙΣΤΗΜΗΣ ΤΡΟΦΙΜΩΝ ΚΑΙ ΔΙΑΤΡΟΦΗΣ ΤΟΥ ΑΝΘΡΩΠΟΥ ΜΕΤΑΠΤΥΧΙΑΚΟ ΠΡΟΓΡΑΜΜΑ ΣΠΟΥΔΩΝ «ΕΠΙΣΤΗΜΗΣ ΚΑΙ ΤΕΧΝΟΛΟΓΙΑΣ ΤΡΟΦΙΜΩΝ ΚΑΙ ΔΙΑΤΡΟΦΗΣ ΤΟΥ ΑΝΘΡΩΠΟΥ» ΕΡΓΑΣΤΗΡΙΟ ΓΑΛΑΚΤΟΚΟΜΙΑΣ
Science of Sericulture
 Science of Sericulture 2012 38 1 0065-0069 ISSN 0257-4799 CN 32-1115 /S E-mail CYKE@ chinajournal. net. cn 215123 25 0 ~ 24 h GSH GSSG GSH + 2GSSG GSH /GSSG S- GST TPX 23% GR 57% GSH TPX GST GSSG 61% GSH
Science of Sericulture 2012 38 1 0065-0069 ISSN 0257-4799 CN 32-1115 /S E-mail CYKE@ chinajournal. net. cn 215123 25 0 ~ 24 h GSH GSSG GSH + 2GSSG GSH /GSSG S- GST TPX 23% GR 57% GSH TPX GST GSSG 61% GSH
Το Βιολογικό Ρολόι: Θεµελιώδης Ρυθµιστής της Φυσιολογίας των Οργανισµών
 Ιούλιος 2005 Το Βιολογικό Ρολόι: Θεµελιώδης Ρυθµιστής της Φυσιολογίας των Οργανισµών ρ. Α. Προµπονά Εργαστήριο Ρύθµισης της Μεταγραφής από το Βιολογικό Ρολόι Κιρκαδικό Ρολόι Ρυθµοί µε περίοδο ~ 24 ωρών
Ιούλιος 2005 Το Βιολογικό Ρολόι: Θεµελιώδης Ρυθµιστής της Φυσιολογίας των Οργανισµών ρ. Α. Προµπονά Εργαστήριο Ρύθµισης της Μεταγραφής από το Βιολογικό Ρολόι Κιρκαδικό Ρολόι Ρυθµοί µε περίοδο ~ 24 ωρών
Αξιοποίηση Φυσικών Αντιοξειδωτικών στην Εκτροφή των Αγροτικών
 Ζώων για Παραγωγή Προϊόντων Ποιότητας» 1 Αξιοποίηση Φυσικών Αντιοξειδωτικών στην Εκτροφή των Αγροτικών Ζώων για Παραγωγή Προϊόντων Ποιότητας Γεωπονικό Πανεπιστήμιο Αθηνών Εργαστήριο Ζωοτεχνίας MIS 380231
Ζώων για Παραγωγή Προϊόντων Ποιότητας» 1 Αξιοποίηση Φυσικών Αντιοξειδωτικών στην Εκτροφή των Αγροτικών Ζώων για Παραγωγή Προϊόντων Ποιότητας Γεωπονικό Πανεπιστήμιο Αθηνών Εργαστήριο Ζωοτεχνίας MIS 380231
CRC Croce Tili List of patients for mir125b Metabolomic study Clinical data update 13/01/12
 AGGRESSIVE CRC Croce Tili List of patients for mir125b Metabolomic study Clinical data update 13/01/12 INDOLENT Table S1. Clinical data of the patients used for the study ID ZAP %VH WBC Dx date First TX
AGGRESSIVE CRC Croce Tili List of patients for mir125b Metabolomic study Clinical data update 13/01/12 INDOLENT Table S1. Clinical data of the patients used for the study ID ZAP %VH WBC Dx date First TX
Αντιδράσεις οξειδωτικού μέρους. Μη οξειδωτικές αντιδράσεις ΔΡΟΜΟΣ ΤΩΝ ΦΩΣΦΟΤΙΚΩΝ ΠΕΝΤΟΖΩΝ. 6-Ρ γλυκόζη. 5-Ρ ριβουλόζη
 Αντιδράσεις οξειδωτικού μέρους 6-Ρ γλυκόζη 5-Ρ ριβουλόζη ΔΡΟΜΟΣ ΤΩΝ ΦΩΣΦΟΤΙΚΩΝ ΠΕΝΤΟΖΩΝ 5-Ρ ριβόζη (C 5 ) 5-Ρ ξυλουλόζη (C 5 ) GAP (C 3 ) 7-Ρ σεδοεπτουλόζη (C 7 ) 6-Ρ φρουκτόζη(c 6 ) 4-Ρ ερυθρόζη (C 5
Αντιδράσεις οξειδωτικού μέρους 6-Ρ γλυκόζη 5-Ρ ριβουλόζη ΔΡΟΜΟΣ ΤΩΝ ΦΩΣΦΟΤΙΚΩΝ ΠΕΝΤΟΖΩΝ 5-Ρ ριβόζη (C 5 ) 5-Ρ ξυλουλόζη (C 5 ) GAP (C 3 ) 7-Ρ σεδοεπτουλόζη (C 7 ) 6-Ρ φρουκτόζη(c 6 ) 4-Ρ ερυθρόζη (C 5
RHOA inactivation enhances Wnt signaling and promotes colorectal cancer
 RHOA and colorectal cancer Paulo Rodrigues et al 2014 SUPPLEMENTARY INFORMATION RHOA inactivation enhances Wnt signaling and promotes colorectal cancer Paulo Rodrigues, Irati Macaya, Sarah Bazzocco, Rocco
RHOA and colorectal cancer Paulo Rodrigues et al 2014 SUPPLEMENTARY INFORMATION RHOA inactivation enhances Wnt signaling and promotes colorectal cancer Paulo Rodrigues, Irati Macaya, Sarah Bazzocco, Rocco
I ΙΑΙΤΕΡΟΤΗΤΕΣ ΤΗΣ ΙΑΠΙΣΤΕΥΣΗΣ ΤΩΝ ΜΙΚΡΟΒΙΟΛΟΓΙΚΩΝ ΕΡΓΑΣΤΗΡΙΩΝ ΝΕΡΟΥ ΚΑΤΑ ISO 17025. ΠΗΓΕΣ ΑΝΟΜΟΙΟΓΕΝΕΙΑΣ ΚΑΙ ΛΑΘΩΝ
 I ΙΑΙΤΕΡΟΤΗΤΕΣ ΤΗΣ ΙΑΠΙΣΤΕΥΣΗΣ ΤΩΝ ΜΙΚΡΟΒΙΟΛΟΓΙΚΩΝ ΕΡΓΑΣΤΗΡΙΩΝ ΝΕΡΟΥ ΚΑΤΑ ISO 17025. Ρ. Αθηνά Μαυρίδου Καθ. Μικροβιολογίας ΤΕΙ Αθήνας ΠΗΓΕΣ ΑΝΟΜΟΙΟΓΕΝΕΙΑΣ ΚΑΙ ΛΑΘΩΝ Φυσική ανοµοιογένεια νερού στην προέλευση
I ΙΑΙΤΕΡΟΤΗΤΕΣ ΤΗΣ ΙΑΠΙΣΤΕΥΣΗΣ ΤΩΝ ΜΙΚΡΟΒΙΟΛΟΓΙΚΩΝ ΕΡΓΑΣΤΗΡΙΩΝ ΝΕΡΟΥ ΚΑΤΑ ISO 17025. Ρ. Αθηνά Μαυρίδου Καθ. Μικροβιολογίας ΤΕΙ Αθήνας ΠΗΓΕΣ ΑΝΟΜΟΙΟΓΕΝΕΙΑΣ ΚΑΙ ΛΑΘΩΝ Φυσική ανοµοιογένεια νερού στην προέλευση
The effect of curcumin on the stability of Aβ. dimers
 The effect of curcumin on the stability of Aβ dimers Li Na Zhao, See-Wing Chiu, Jérôme Benoit, Lock Yue Chew,, and Yuguang Mu, School of Physical and Mathematical Sciences, Nanyang Technological University,
The effect of curcumin on the stability of Aβ dimers Li Na Zhao, See-Wing Chiu, Jérôme Benoit, Lock Yue Chew,, and Yuguang Mu, School of Physical and Mathematical Sciences, Nanyang Technological University,
Επιγενετικοί παράγοντες και αύξηση σωματικού βάρους
 Επιγενετικοί παράγοντες και αύξηση σωματικού βάρους Αναστάσιος Μόρτογλου, Ενδοκρινολόγος Δντης τομέα Ενδοκρινολογίας, Διαβήτου και Μεταβολισμού Ιατρικού Κέντρου Αθηνών 9 ο Πανελλήνιο Συνέδριο Παχυσαρκίας
Επιγενετικοί παράγοντες και αύξηση σωματικού βάρους Αναστάσιος Μόρτογλου, Ενδοκρινολόγος Δντης τομέα Ενδοκρινολογίας, Διαβήτου και Μεταβολισμού Ιατρικού Κέντρου Αθηνών 9 ο Πανελλήνιο Συνέδριο Παχυσαρκίας
Ινσουλίνη και γήρανση
 Ινσουλίνη και γήρανση Γεώργιος Δ. Δημητριάδης Καθηγητής Παθολογίας Διευθυντής Β Προπαιδευτικής Παθολογικής Κλινικής, Μονάδας Έρευνας και Διαβητολογικού Κέντρου Πανεπιστημίου Αθηνών, ΠΓΝΑ Αττικόν Πρόεδρος
Ινσουλίνη και γήρανση Γεώργιος Δ. Δημητριάδης Καθηγητής Παθολογίας Διευθυντής Β Προπαιδευτικής Παθολογικής Κλινικής, Μονάδας Έρευνας και Διαβητολογικού Κέντρου Πανεπιστημίου Αθηνών, ΠΓΝΑ Αττικόν Πρόεδρος
Εθνικό Μετσόβιο Πολυτεχνείο ΑΝΑΠΤΥΞΗ ΤΕΧΝΙΚΗΣ ΜΕΤΡΗΣΗΣ ΕΚΚΡΙΣΗΣ ΠΡΩΤΕΪΝΩΝ ΚΟΝΤΑ ΣΤΗ ΚΥΤΤΑΡΙΚΗ ΕΠΙΦΑΝΕΙΑ
 Εθνικό Μετσόβιο Πολυτεχνείο Σχολή Μηχανολόγων Μηχανικών ΕΡΓΑΣΤΗΡΙΟ ΕΜΒΙΟΜΗΧΑΝΙΚΗΣ ΚΑΙ ΣΥΣΤΗΜΙΚΗΣ ΒΙΟΛΟΓΙΑΣ ΤΟΜΕΑΣ ΜΗΧΑΝΟΛΟΓΙΚΩΝ ΚΑΤΑΣΚΕΥΩΝ ΚΑΙ ΑΥΤΟΜΑΤΟΥ ΕΛΕΓΧΟΥ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ ΑΝΑΠΤΥΞΗ ΤΕΧΝΙΚΗΣ ΜΕΤΡΗΣΗΣ
Εθνικό Μετσόβιο Πολυτεχνείο Σχολή Μηχανολόγων Μηχανικών ΕΡΓΑΣΤΗΡΙΟ ΕΜΒΙΟΜΗΧΑΝΙΚΗΣ ΚΑΙ ΣΥΣΤΗΜΙΚΗΣ ΒΙΟΛΟΓΙΑΣ ΤΟΜΕΑΣ ΜΗΧΑΝΟΛΟΓΙΚΩΝ ΚΑΤΑΣΚΕΥΩΝ ΚΑΙ ΑΥΤΟΜΑΤΟΥ ΕΛΕΓΧΟΥ ΔΙΠΛΩΜΑΤΙΚΗ ΕΡΓΑΣΙΑ ΑΝΑΠΤΥΞΗ ΤΕΧΝΙΚΗΣ ΜΕΤΡΗΣΗΣ
Αθήνα, 15 Οκτωβρίου 2014 Αρ. Πρωτ.: 2988
 ΕΛΛΗΝΙΚΗ ΔΗΜΟΚΡΑΤΙΑ ΥΠΟΥΡΓΕΙΟ ΠΑΙΔΕΙΑΣ ΚΑΙ ΘΡΗΣΚΕΥΜΑΤΩΝ ΓΕΝΙΚΗ ΓΡΑΜΜΑΤΕΙΑ ΕΡΕΥΝΑΣ & ΤΕΧΝΟΛΟΓΙΑΣ Αθήνα, 15 Οκτωβρίου 2014 Αρ. Πρωτ.: 2988 Θέμα: Έγκριση πρόσκλησης εκδήλωσης ενδιαφέροντος με απευθείας ανάθεση
ΕΛΛΗΝΙΚΗ ΔΗΜΟΚΡΑΤΙΑ ΥΠΟΥΡΓΕΙΟ ΠΑΙΔΕΙΑΣ ΚΑΙ ΘΡΗΣΚΕΥΜΑΤΩΝ ΓΕΝΙΚΗ ΓΡΑΜΜΑΤΕΙΑ ΕΡΕΥΝΑΣ & ΤΕΧΝΟΛΟΓΙΑΣ Αθήνα, 15 Οκτωβρίου 2014 Αρ. Πρωτ.: 2988 Θέμα: Έγκριση πρόσκλησης εκδήλωσης ενδιαφέροντος με απευθείας ανάθεση
19 Ενδοκρινολόγος. Φάρµακα που στοχεύουν το β- κύτταρο ΕΥΡΥΔΙΚΗ ΠΑΠΑΔΟΠΟΥΛΟΥ ΕΙΣΑΓΩΓΗ ΜΗΧΑΝΙΣΜΟΣ ΔΡΑΣΗΣ
 Φάρµακα που στοχεύουν το β- κύτταρο ΕΥΡΥΔΙΚΗ ΠΑΠΑΔΟΠΟΥΛΟΥ 19 Ενδοκρινολόγος ΕΙΣΑΓΩΓΗ Οι σουλφονυλουρίες είναι η πρώτη κατηγορία υπογλυκαιμικών φαρμάκων που χορηγήθηκαν για την αντιμετώπιση του διαβήτη
Φάρµακα που στοχεύουν το β- κύτταρο ΕΥΡΥΔΙΚΗ ΠΑΠΑΔΟΠΟΥΛΟΥ 19 Ενδοκρινολόγος ΕΙΣΑΓΩΓΗ Οι σουλφονυλουρίες είναι η πρώτη κατηγορία υπογλυκαιμικών φαρμάκων που χορηγήθηκαν για την αντιμετώπιση του διαβήτη
Διάλεξη 5. Μπράλιου Γεωργία, PhD, Τμήμα Πληροφορικής με Εφαρμογές στη Βιοϊατρική Πανεπιστήμιο Θεσσαλίας
 Διάλεξη 5 Δομή μεμβρανών (πάχος 5nm, 50 άτομα) Μεμβρανική μεταφορά Φραγμοί Μεμβρανικές πρωτεΐνες Λειτουργίες μεμβρανικών πρωτεϊνών Δίαυλοι-μεταφορείς-δυναμικό της μεμβράνης G protein coupled receptors
Διάλεξη 5 Δομή μεμβρανών (πάχος 5nm, 50 άτομα) Μεμβρανική μεταφορά Φραγμοί Μεμβρανικές πρωτεΐνες Λειτουργίες μεμβρανικών πρωτεϊνών Δίαυλοι-μεταφορείς-δυναμικό της μεμβράνης G protein coupled receptors
Αναπαραγωγική Γήρανση Replicative Senescence
 Αναπαραγωγική Γήρανση Replicative Senescence Αναπαραγωγική Γήρανση Ονομάζεται το σταμάτημα του κυτταρικού πολλαπλασιασμού μετά από περιορισμένο αριθμό κυτταρικών διαιρέσεων (π.χ. 25-50 διαιρέσεις για ινοβλάστες
Αναπαραγωγική Γήρανση Replicative Senescence Αναπαραγωγική Γήρανση Ονομάζεται το σταμάτημα του κυτταρικού πολλαπλασιασμού μετά από περιορισμένο αριθμό κυτταρικών διαιρέσεων (π.χ. 25-50 διαιρέσεις για ινοβλάστες
Γλυκόζη. Ανασκόπηση μεταβολισμού υδατανθρακών ΗΠΑΡ. ΤΡΟΦΗ Γλυκόζη. Κυκλοφορία. Κυτταρόπλασμα ΓΛΥΚΟΓΟΝΟ. Οδός Φωσφ. Πεντοζών.
 ΓΛΥΚΟΛΥΣΗ ΙΙ Ανασκόπηση μεταβολισμού υδατανθρακών ΗΠΑΡ ΤΡΟΦΗ Γλυκόζη Κυκλοφορία ΓΛΥΚΟΓΟΝΟ Γλυκόζη ΗΠΑΡ Κυτταρόπλασμα ΗΠΑΡ (Οξέωση) NADH ΟΞΕΙΔ. ΦΩΣΦ. ATP CO 2 Φρουκτόζη Γαλακτόζη 2ATP+2NADH Αναερόβια Γαλακτικό
ΓΛΥΚΟΛΥΣΗ ΙΙ Ανασκόπηση μεταβολισμού υδατανθρακών ΗΠΑΡ ΤΡΟΦΗ Γλυκόζη Κυκλοφορία ΓΛΥΚΟΓΟΝΟ Γλυκόζη ΗΠΑΡ Κυτταρόπλασμα ΗΠΑΡ (Οξέωση) NADH ΟΞΕΙΔ. ΦΩΣΦ. ATP CO 2 Φρουκτόζη Γαλακτόζη 2ATP+2NADH Αναερόβια Γαλακτικό
